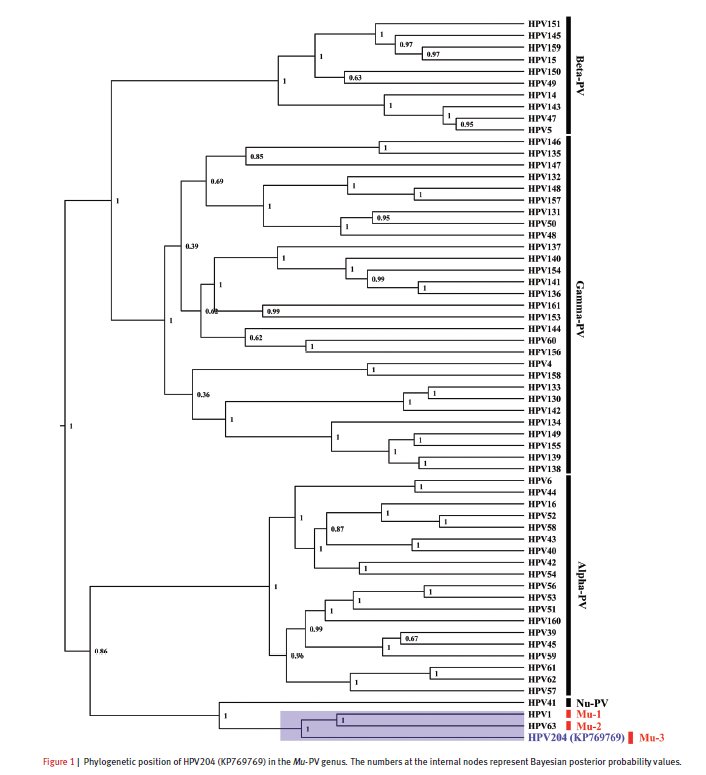
As of May 30, 2015, 201 different HPV types had been completely sequenced and officially recognized and divided into five PV-genera: Alpha-, Beta-, Gamma-, Mu-, and Nupapillomavirus.
根据文献,我找到了hpv所有已知测序种类的参考基因组网站:
http://www.hpvcenter.se/html/refclones.html
到目前(2015年7月31日15:17:59)已经有了205种,我爬取它们的genebank ID号,然后用python程序批量下载了它们的序列,能下载的序列共179条,都是8K左右的碱基序列。
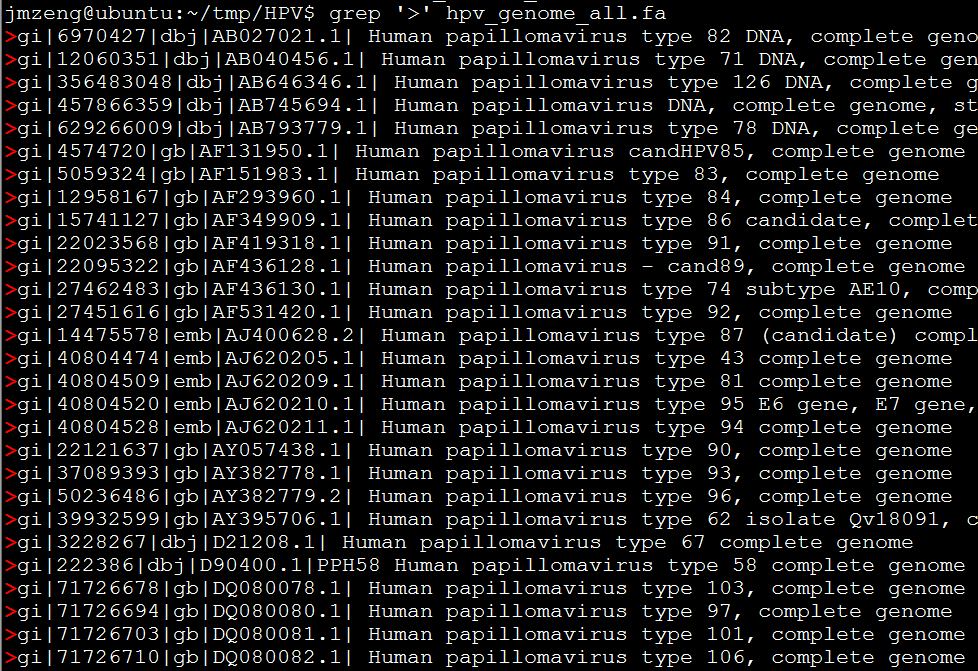
根据genebank ID或者其它ID号批量下载核酸序列的脚本如下:
[python]</pre>
import sys
import time
import random
from Bio import Entrez
ids=[]
infile=sys.argv[1]
for line in open(infile,'r'):
line=line.strip()
ids.append(line)
for i in range(1,len(ids)):
# t = random.randrange(0,5)
handle =
Entrez.efetch(db="nucleotide", id=ids[i],rettype="fasta",email="jmzeng1314@163.com")
# time.sleep(t)
print handle.read()
[/python]
脚本使用很简单,保持输入文件是一行一个ID号即可。
同时,根据文献我们也能得到hbv病毒提取方法
当然,我是看不懂的。

同样的拿到下载的178条序列我们可以做一个进化树,当然,这个文章已经做好了,我就不做了,进化树其实蛮简单的。
下载179条hpv序列,每条序列都是8KB左右
我还用了R脚本批量下载
library(ape)
a=read.table("hpv_all.ID") #输入文件是一行一个ID号即可
for (i in 1:nrow(a)){
tmp=read.GenBank(a[i,1],seq.names = a[1,1],as.character = T)
write.dna(tmp,"tmp.fa",format="fasta", append=T,colsep = "")
}
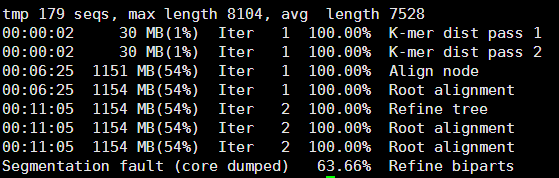
然后用muscle做比对,参照我之前的笔记
http://www.bio-info-trainee.com/?p=659
http://www.bio-info-trainee.com/?p=660
http://www.bio-info-trainee.com/?p=626
muscle -in mouse_J.pro -out mouse_J.pro.a
muscle -maketree -in mouse_J.pro.a -out mouse_J.phy
貌似时间有点长呀,最后还莫名其妙的挂掉了,可能是我的服务器配置有点低。
进化树如下所示: