懂点芯片数据分析的都应该知道,芯片设计的时候,对一个基因设计了多个探针,这样设计是为了更好的捕获某些难以发现的基因,或者重点研究某些基因。
但是对我们的差异分析不方便,所以我们只分析哪些有对应了entrez ID的探针,而且对每个entrez ID,我们只需要挑选它表达量最高的那个探针。
所以就演化为一个编程问题:分组求最大值,多公共列合并!
如果是在R语言里面,那么首先这个table的表示形式如下
> esetDataTable[1:10,c(7,8)]
EGID rowMeans
1000_at 5595 1840.04259751826
1001_at 7075 799.075414422572
1002_f_at 1557 50.4884096416177
1003_s_at 643 142.372008051308
1004_at 643 211.65300963049
1005_at 1843 4281.29318032004
1006_at 4319 38.5784289213085
1007_s_at NA 1489.98158531843
1008_f_at 5610 4013.576753977
1009_at 3094 3070.50648167305
我们首先看看一个R语言函数处理方式吧,这个是比较容易想到的算法,但是用到了循环,非常的不经济,计算量很大。
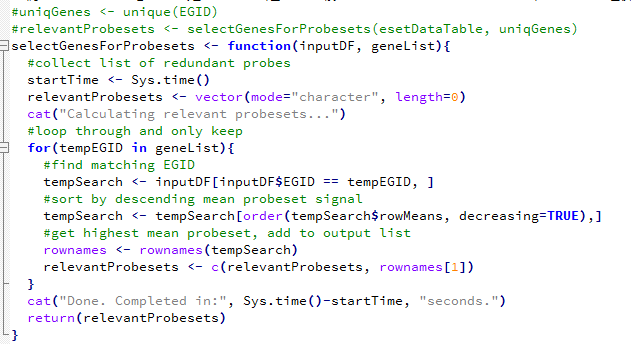
因为里面涉及到了双重循环。我进行了人工计时,这个程序耗费了一分钟十二秒,程序里面的计时器有点问题。
然后我再讲一个精简版的算法
dat=esetDataTable[,c(7,8)]
dat=as.data.frame(apply(dat,2,as.numeric))
dat$prob=rownames(esetDataTable)
首先可以得到需要提取的数据所在行的两个值
match_row=aggregate(dat,by=list(dat[,1]),max)
类似于这句SQL语句:SELECT max(a.rowMeans) as val, a.EGID FROM test.esetDataTable a group by a.EGID
现在要根据match_row表去原表esetDataTable里面提取我们的探针ID数据。

这样就完美解决了问题,我们的合并后的数据的prob列,就是前面那个函数计算了一分多钟的返回的非冗余探针ID向量,relevantProbesets,但是这次只花了不到一秒钟。
tmp_prob=merge(dat,match_row,by=c("EGID","rowMeans"))
setdiff(as.character(relevantProbesets),as.character(tmp_prob$prob))
length(union(as.character(relevantProbesets),as.character(tmp_prob$prob)))
setdiff(as.character(tmp_prob$prob),as.character(relevantProbesets))
我顺便检查了一下上面那个复杂的R函数跟我这次精简版的结果,发现这次才是对的,上面那个错了。
你们能发现上面那个为什么错了吗?
如果是在mysql里面它的表现形式如下;
mysql> select row_names,EGID,rowMeans from esetDataTable order by EGID limit 10;
+------------+-------+------------------+
| row_names | EGID | rowMeans |
+------------+-------+------------------+
| 38912_at | 10 | 85.8820246154773 |
| 41654_at | 100 | 301.720067595558 |
| 907_at | 100 | 273.100008206028 |
| 2053_at | 1000 | 354.207060199715 |
| 2054_g_at | 1000 | 33.8472900312781 |
| 40781_at | 10000 | 425.007640082848 |
| 35430_at | 10001 | 152.885791914329 |
| 35431_g_at | 10001 | 181.915087187117 |
| 35432_at | 10001 | 184.869017764782 |
| 31366_at | 10002 | 44.9716205901791 |
+------------+-------+------------------+
10 rows in set (0.05 sec)
如果要用SQL来做同样的事情需要下面这个语句,这个就非常简单啦!
select b.*
from test.esetDataTable b
inner join (
SELECT max(a.rowMeans) as val, a.EGID FROM test.esetDataTable a group by a.EGID
) as c on b.EGID=c.EGID and b.rowMeans=c.val
结果是:8640 rows in set (4.56 sec) 看起来mysql还没有R语言快速,但是这个mysql语句很明显也不是很高效,但是我的mysql水平有限。
稍微解释一下这个mysql语句,其中SELECT max(a.rowMeans) as val, a.EGID FROM test.esetDataTable a group by a.EGID类似于R里面的match_row=aggregate(dat,by=list(dat[,1]),max),就是把每个entrez ID对应着的最大值提取出来成一个新的表
而inner join on b.EGID=c.EGID and b.rowMeans=c.val 就是我们的tmp_prob=merge(dat,match_row,by=c("EGID","rowMeans")) 根据两列来合并两个数据框