最近学徒在交流群里面点名了一个新鲜出炉( 2023年12月5日)的cell期刊的文章,因为里面有常规转录组,单细胞转录组,以及空间单细胞转录组,主流生物信息学技术都被囊括了,非常适合初学者练手。通讯作者是中山大学孙逸仙纪念医院的苏士成,他最近非常出名,被选为2023年“新基石研究员”,获2500万元资助。
这个文章标题短小精悍:《Choroid plexus mast cells drive tumor-associated hydrocephalus》,相关数据在:https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE207546
可以看到是常规转录组,单细胞转录组,以及空间单细胞转录组,如下所示:
- sn-seq: two exprimental groups: normal and EO771-VM-bearing mice, 3 independent replicates with 5 mice per group in each replicate.
- ST, 3 brain sections from 3 EO771-VM mice.
- 3.RNA-seq: human choroid plexus epithelial cell line (HCPEpiC) treated with or without tryptase (3 independent replicates per group).
让我们来看看文章对这个数据做了什么样的分析吧!
首先是单细胞转录组(核)
单细胞核转录组跟普通的单细胞转录组在数据分析层面大差不差,都是降维聚类分群,但是它的质量控制标准里面需要注意线粒体和核糖体的阈值哦!文章做了第一层次降维聚类分群后,取里面的免疫细胞亚群进行细分,如下所示:
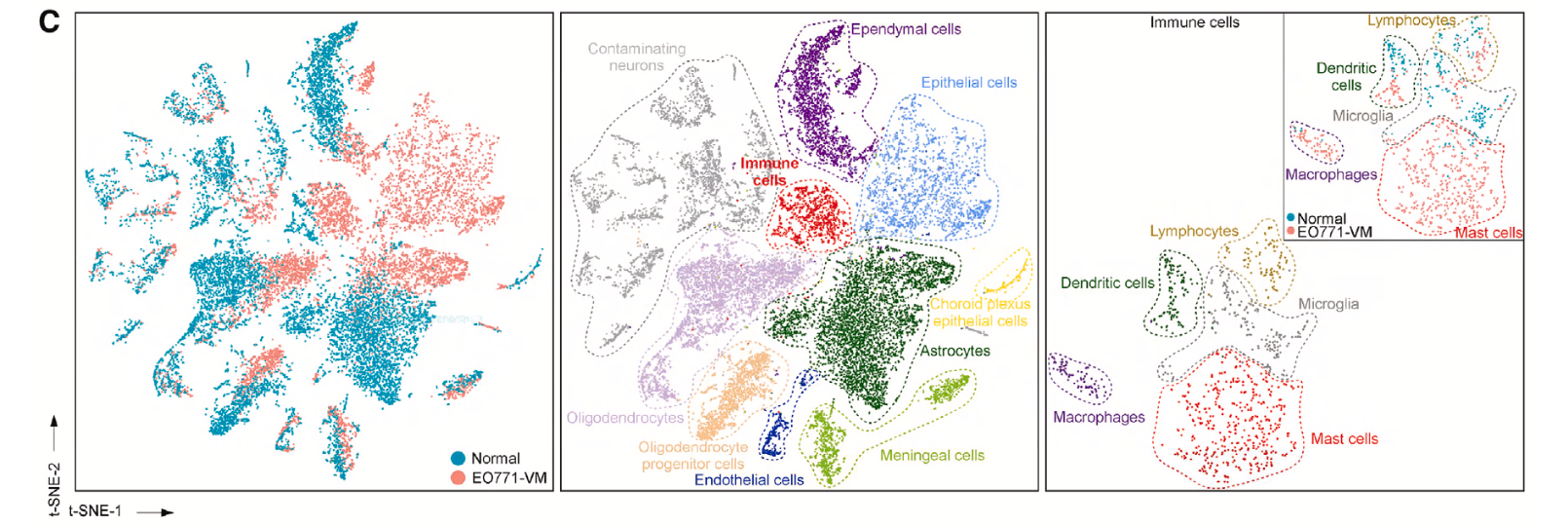
因为这里面的是小鼠大脑,几年前我在:两个神经退行性疾病的单细胞核转录组队列的细胞亚群及其标记基因的比较,列出来了一些大脑的单细胞亚群基因:
astrocytes = c("AQP4", "ADGRV1", "GPC5", "RYR3")
endothelial = c("CLDN5", "ABCB1", "EBF1")
excitatory = c("CAMK2A", "CBLN2", "LDB2")
inhibitory = c("GAD1", "LHFPL3", "PCDH15")
microglia = c("C3", "LRMDA", "DOCK8")
oligodendrocytes = c("MBP", "PLP1", "ST18")
OPC='Tnr,Igsf21,Neu4,Gpr17'
Ependymal='Cfap126,Fam183b,Tmem212,pifo,Tekt1,Dnah12'
pericyte=c( 'DCN', 'LUM', 'GSN' ,'FGF7','MME', 'ACTA2','RGS5')
# 下面是4种神经细胞
# excitatory (SLC17A6),
# inhibitory (GAD2),
# GABAergic (GAD2/GRIK1),
# dopaminergic neurons(TH)
第一个奇怪的地方是,文章拿到的他自己的第一层次降维聚类分群后的结果里面的特异性高表达量基因都是非常的冷门,如下所示:
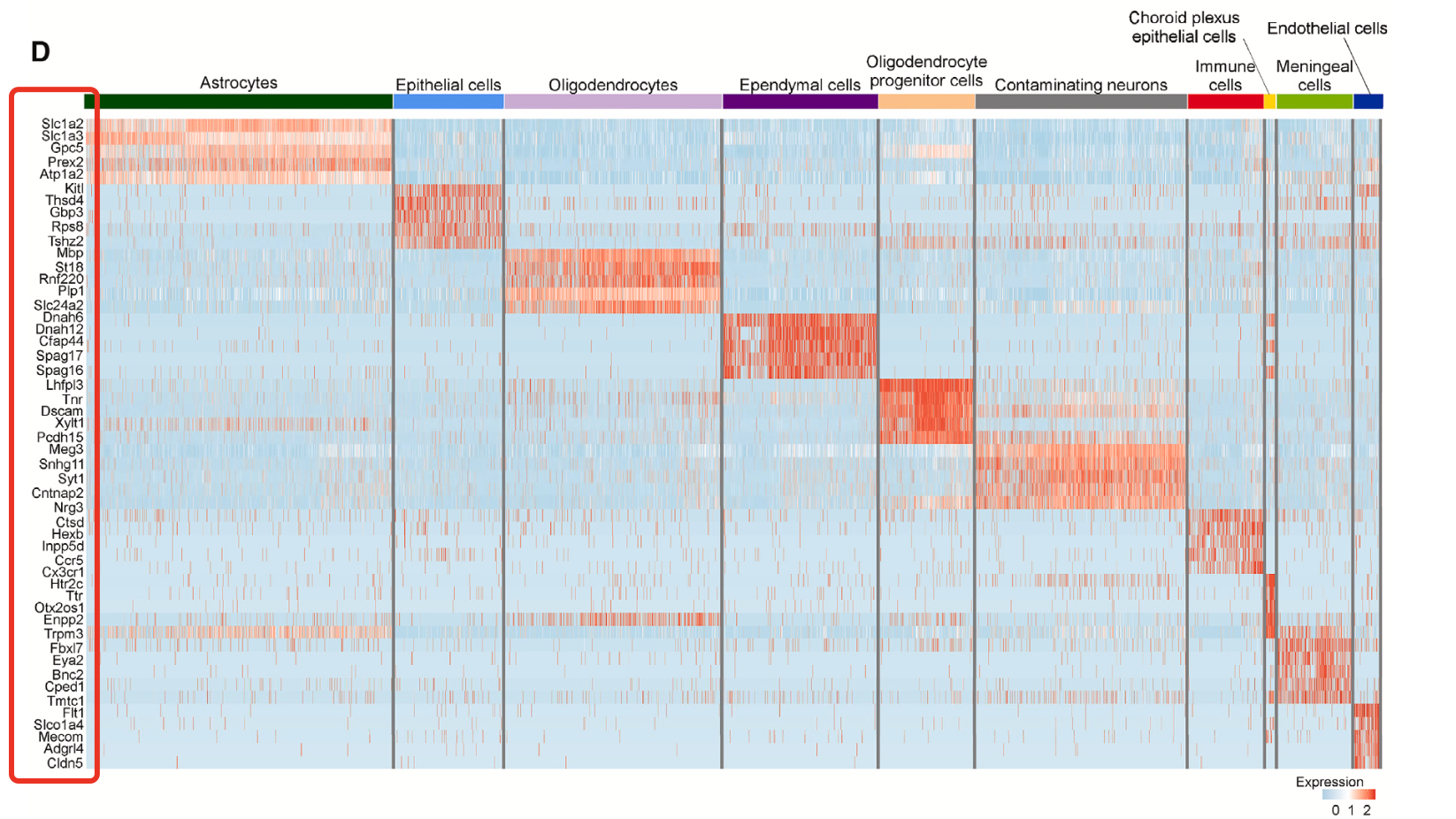
而且如果是使用我们的前面整理好的在:两个神经退行性疾病的单细胞核转录组队列的细胞亚群及其标记基因的比较,列出来了一些大脑的单细胞亚群基因,发现在这个文章的数据集里面略微不是那么的明显啦:
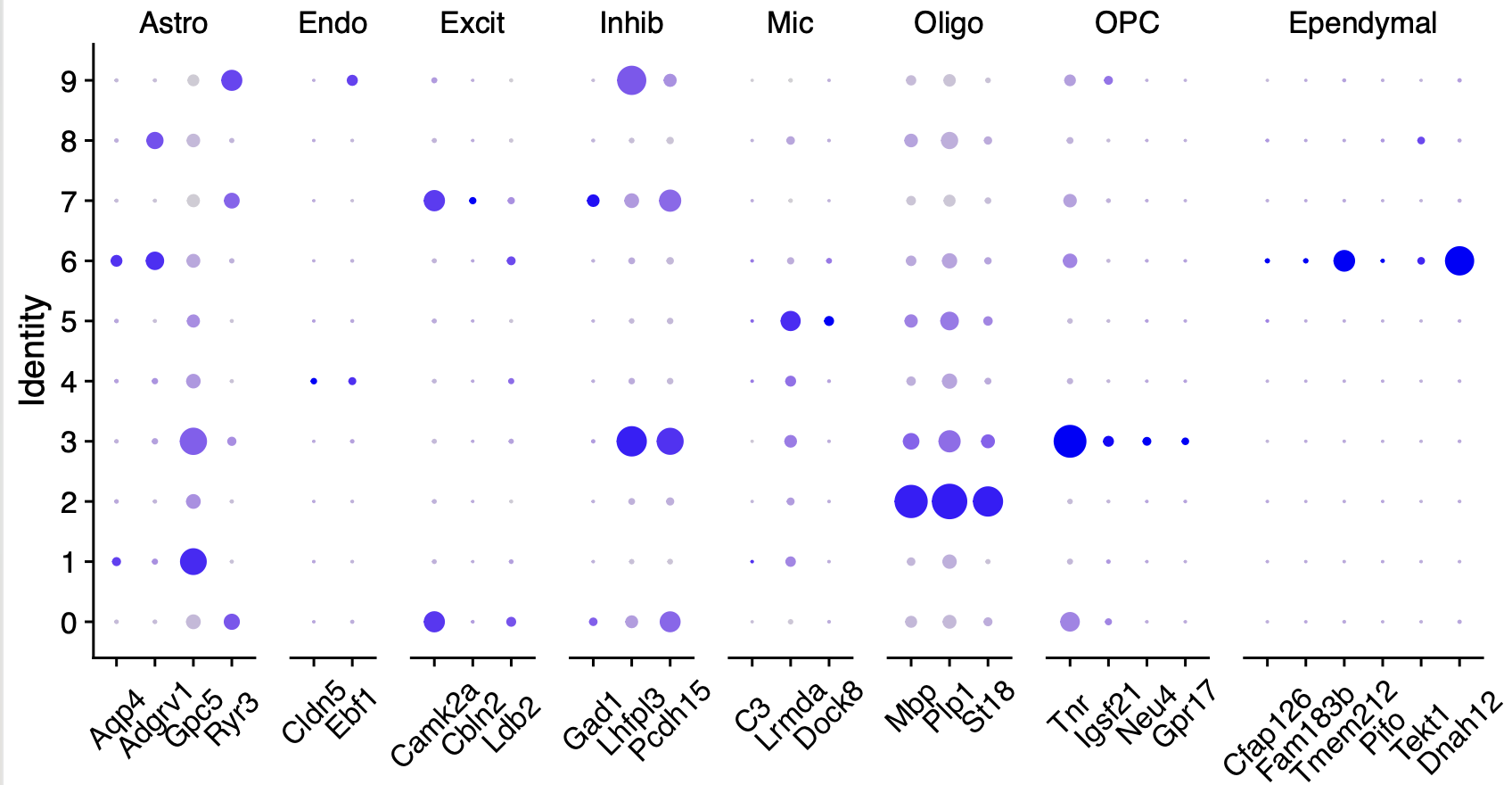
虽然说并不是完全不可以区分,起码没有出现违背常识的现象,虽然冷门但是可以接受。不过接下来的免疫细胞的细分亚群,就有点尴尬了,如下所示:
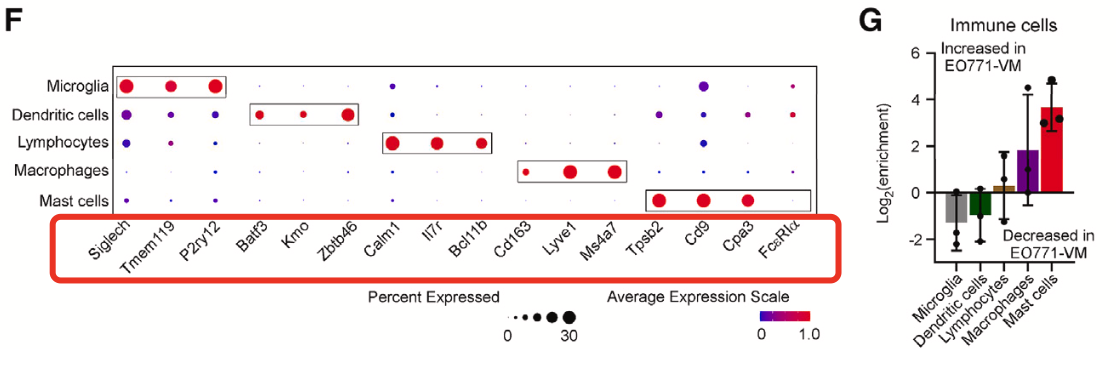
其实免疫细胞的细分亚群就无需理会它是来源于大脑了,通常我们拿到了肿瘤相关的单细胞转录组的表达量矩阵后的第一层次降维聚类分群通常是:
- immune (CD45+,PTPRC),
- epithelial/cancer (EpCAM+,EPCAM),
- stromal (CD10+,MME,fibro or CD31+,PECAM1,endo)
参考我前面介绍过 CNS图表复现08—肿瘤单细胞数据第一次分群通用规则,这3大单细胞亚群构成了肿瘤免疫微环境的复杂。绝大部分文章都是抓住免疫细胞亚群进行细分,包括淋巴系(T,B,NK细胞)和髓系(单核,树突,巨噬,粒细胞)的两大类作为第二次细分亚群。但是也有不少文章是抓住stromal 里面的 fibro 和endo进行细分,并且编造生物学故事的。前面我们已经介绍了心肝脾肺肾等多个器官的上皮细胞的细分亚群, 以及免疫细胞里面的髓系和B细胞细分亚群:
理论上这些背景知识足够支撑我们对这个大脑数据集里面的免疫细胞进行细分了,但是上面的图F里面的基因仍然是大冷门!如果是免疫细胞细分亚群就有问题, 那么文章最大的结论(肿瘤相关脑积水病发时,脉络丛肥大细胞增加。)就站不住脚了。
这就是让我很费劲,虽然说 Single-nucleus RNA sequencing (snRNA-seq) 很多时候可能是质量并不是普通的常规的单细胞转录组,但是也不知道差异如此大。因为绝大部分大脑相关研究仍然是首选的snRNA-seq,说明它也是经过了市场的检验!
单细胞核转录组表达量矩阵是公开的
可以看到https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE207546 数据集里面的单细胞核转录组是六个样品:
GSM6294356 normal1
GSM6294357 normal2
GSM6294358 normal3
GSM6294359 treatment1
GSM6294360 treatment2
GSM6294361 treatment3
每个样品都是有3个文件,如下所示:
GSM6294356_normal1_barcodes.tsv.gz 21.5 Kb
GSM6294356_normal1_genes.tsv.gz 231.4 Kb
GSM6294356_normal1_matrix.mtx.gz 10.1 Mb
全部下载后简单的一个批处理即可;
fs=list.files('./','genes.tsv.gz')
fs
samples1=gsub('_genes.tsv.gz','',fs)
samples1
library(stringr)
samples2=str_split(samples1,'_',simplify = T)[,1]
samples2 =samples1
length(unique(samples2))
samples2
#samples2 = samples1
lapply(1:length(samples2), function(i){
x=samples2[i]
y=samples1[i]
dir.create(x,recursive = T)
file.copy(from=paste0(y,'_genes.tsv.gz'),
to=file.path(x, 'features.tsv.gz' ))
file.copy(from=paste0(y,'_matrix.mtx.gz'),
to= file.path(x, 'matrix.mtx.gz' ) )
file.copy(from=paste0(y,'_barcodes.tsv.gz'),
to= file.path(x, 'barcodes.tsv.gz' ))
})
每个样品一个独立的文件夹,每个里面都是3个文件,名字必须是一模一样的哦,如下所示:
然后我们就可以读取这个文件夹走降维聚类分群啦, 参考前面的收录于《生信技能树》公众号的《单细胞2024》专辑,而且我们从2024开始的教程都是基于Seurat的V5版本啦,之前已经演示了如何读取不同格式的单细胞转录组数据文件,如下所示:
当然了,如果你顺利的感觉我们的教程完成了这个数据分析,就会发现一个很诡异的现象,这里面的免疫细胞相关的很多特异性(耳熟能详)基因是缺少的,尤其是t淋巴细胞相关的,如下所示:
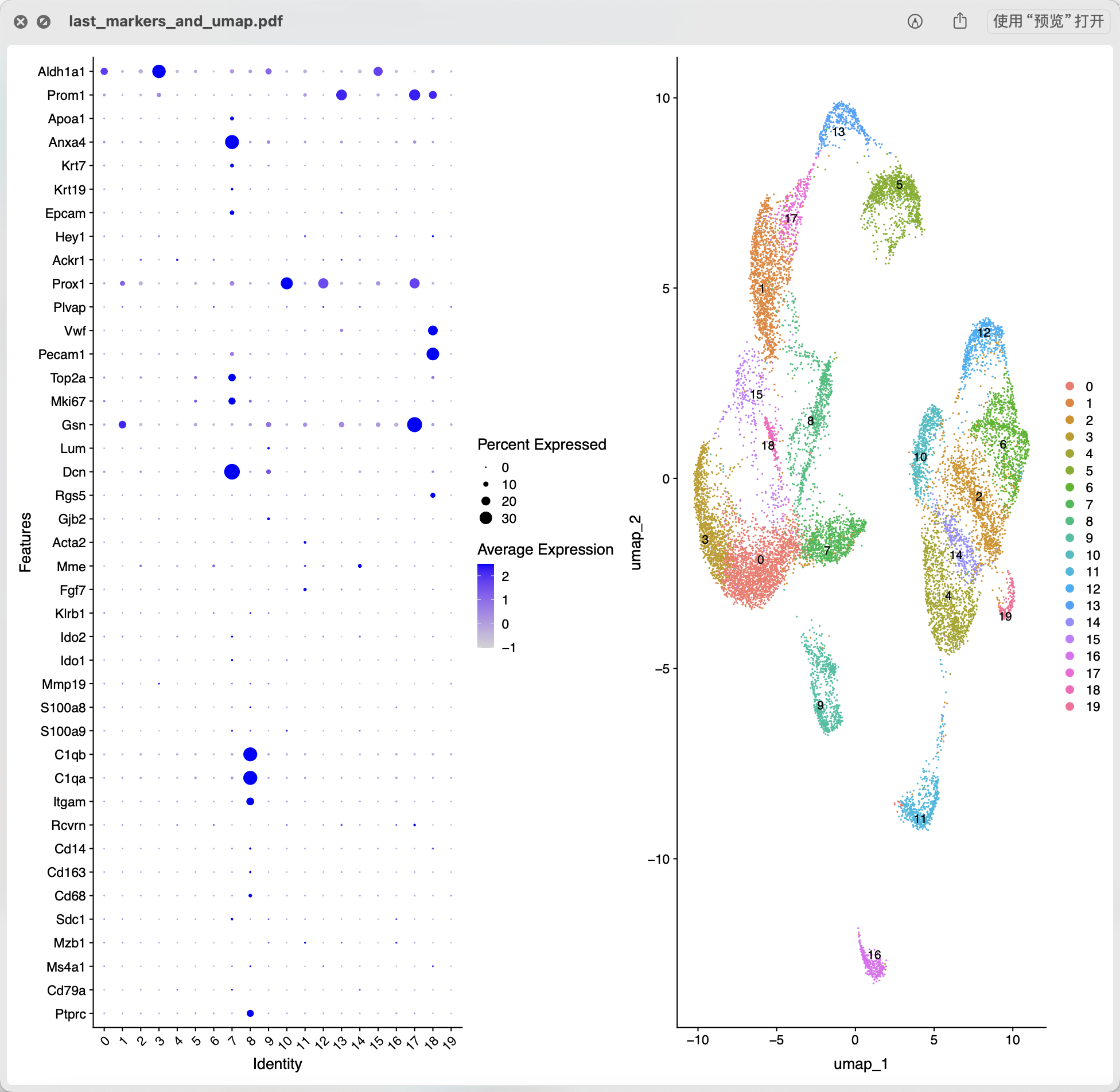
比如:’CD3D’, ‘CD3E’, ‘CD4’,’CD8A’ 等等。。。。
其它组学我们后续慢慢解读
总体上来说,这篇文章的数据分析环节还是有很多值得探讨的地方,当然了这个并不能主要是怪研究者,因为可能是数据产出的实验环节本身就有问题,辣鸡数据进来肯定是辣鸡结果出去,神仙来了也拯救不了。
另外,值得注意的是,我看到一些公众号讨论第一作者的年龄问题:
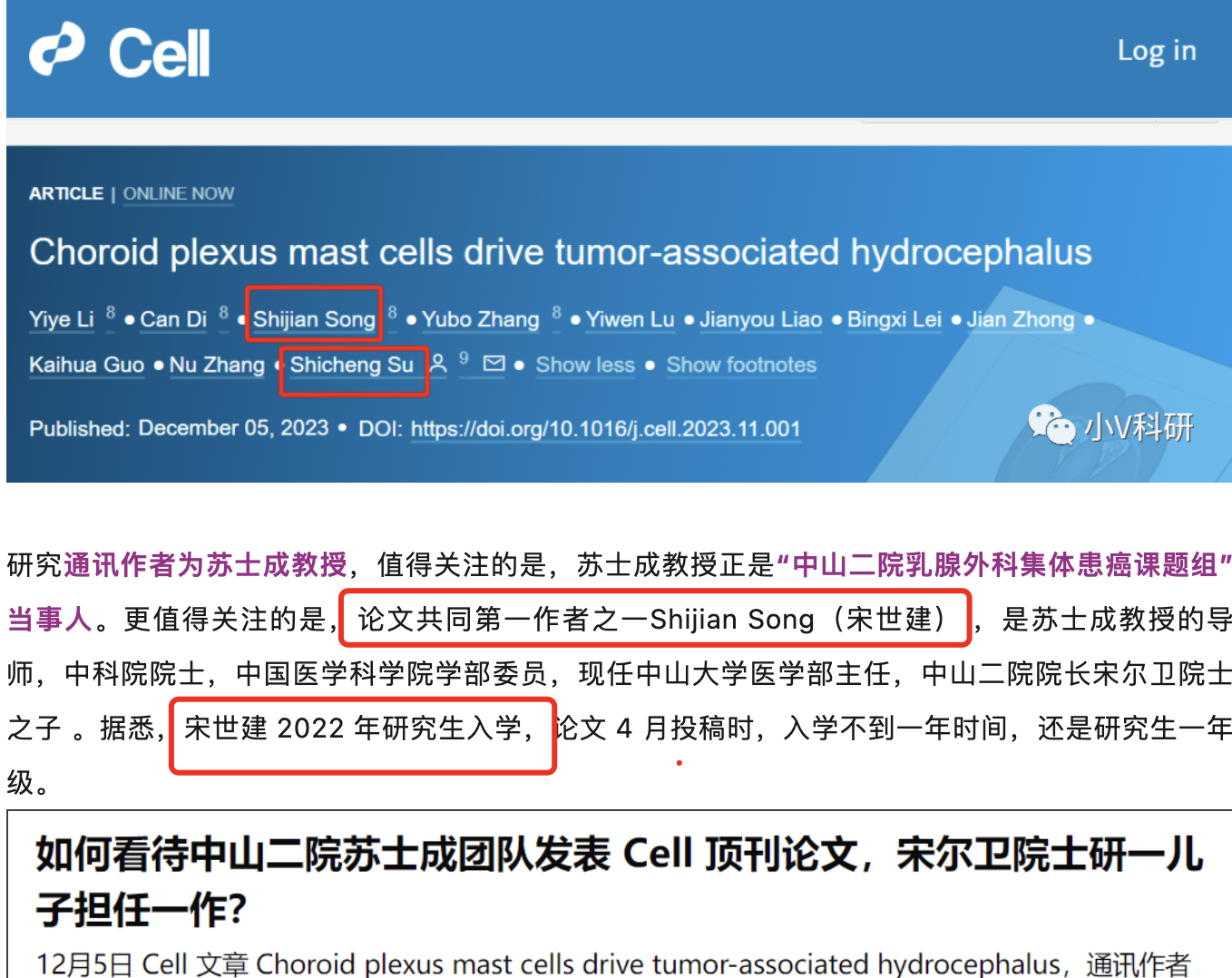
蛮有意思的,一般来说我们18岁读大学,22岁开始研究生,那就是这个人是00后咯。我现在特别的佩服00后,昨天还看到了211高校华中某业大学动物Y养系黄某若教授十几年如一日的学术造假行为被他们课题组的十一名成员联合,共同实名举报,详见:众叛亲离了
我们可以组建一个00后科研局,邀请大家一起来学习这个( 2023年12月5日)的cell期刊的文章的常规转录组,单细胞转录组,以及空间单细胞转录组。我们会提供全部的数据分析代码和前因后果,一起看看里面的数据分析细节是否有瑕疵哈。客观看待生物信息学技术,如果不是主观造假,我觉得数据分析环节本来就是螺旋上升的,早期(2018附近)的单细胞转录组数据分析文章现在看过来也是很多漏洞百出,但是他们因为是先烈所以也很难指责什么。不过这个( 2023年12月5日)的cell期刊的文章是新鲜出炉的,所以还是得讨论讨论哈!