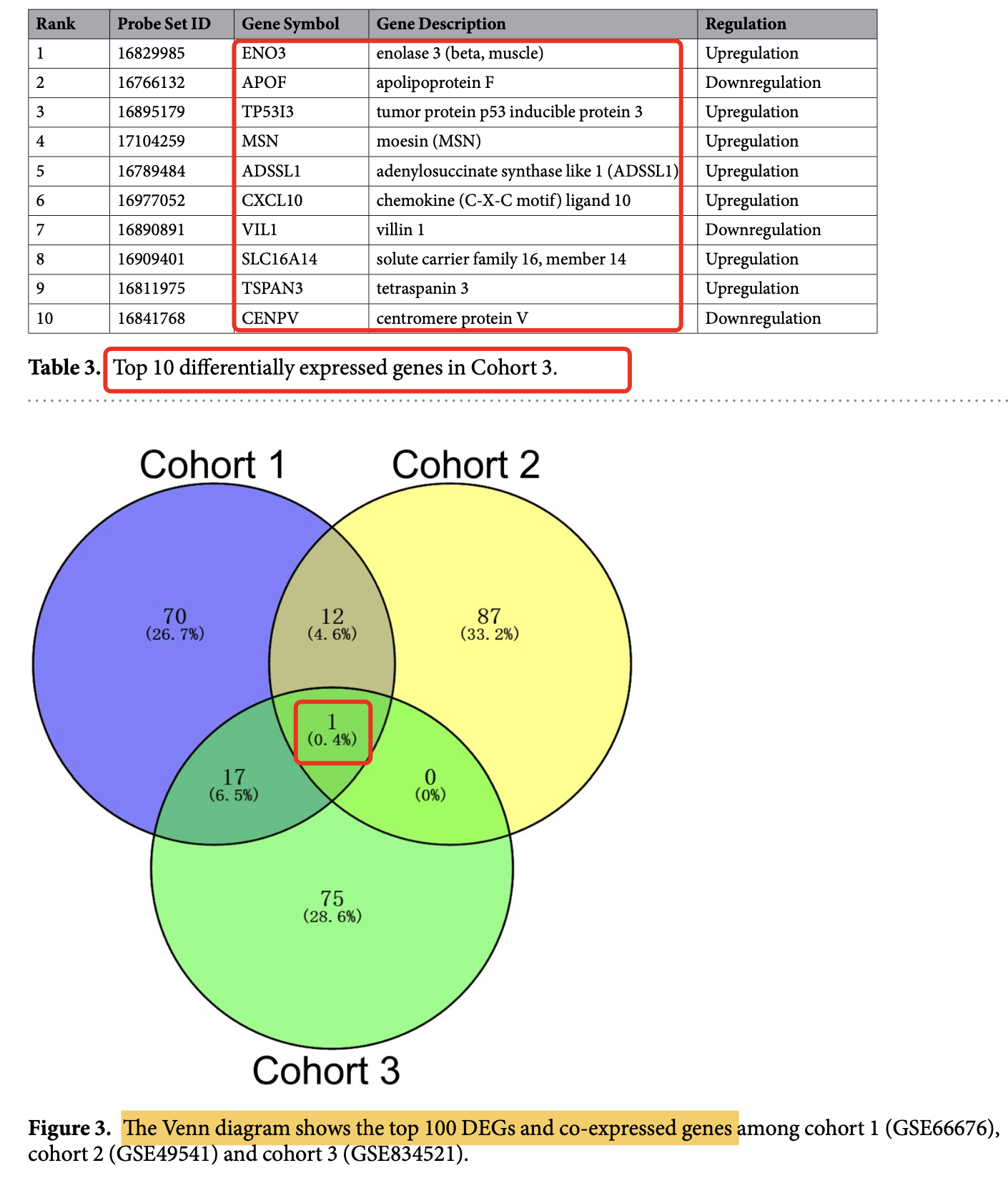
批改了一个学员的表达量芯片作业题,发现她复现的文章很有意思,里面是3个数据集的各自的差异分析但是他们的基因交集是少得可怜, 如下所示;
感兴趣的小伙伴可以看看这个文章哈, 标题是:《A comprehensive bioinformatics analysis on multiple Gene Expression Omnibus datasets of nonalcoholic fatty liver disease and nonalcoholic steatohepatitis》
是如下所示的3个数据集,各自的分组后差异分析:
- GSE66676 (containing 33 NAFLD/NASH tissues and 34 normal liver tissues) (a),
- GSE49541 (containing contained 32 advanced NAFLD tissues and 40 mild NAFLD tissues) (b),
- GSE834521 (126 NASH tissues and 98 normal liver tissues)
这3个数据集各自的差异分析后的基因数量分别是:We identified 8503, 1538, and 94 potential DEGs in GSE66676, GSE49541, and GSE834521, respectively
目前简单的差异分析流程,基本上转录组测序技术和芯片技术拿到的表达量矩阵后续分析大同小异,公众号推文在: - 解读GEO数据存放规律及下载,一文就够
- 解读SRA数据库规律一文就够
- 从GEO数据库下载得到表达矩阵 一文就够
- GSEA分析一文就够(单机版+R语言版)
- 根据分组信息做差异分析- 这个一文不够的
- 差异分析得到的结果注释一文就够
可以看到,不同的数据集的差异分析后的统计学显著的基因数量差异非常恐怖,所以文章上面的韦恩图是每个数据集的差异基因的top100后的交集。
值得注意的是,文章最后是有实验验证的哦。Tissue specimens, RNA extraction and qRT-PCR analysis. - 15 healthy liver tissues and 10 fatty liver tissues from liver donors were enrolled in our study to validate the expression levels of co-expressed DEGs.
- we used real-time qPCR to detect the expression of 8 DEGs using clinical samples, including CD24, PZP, COL1A1, COL1A2, LUM, VCAN, THBS2 and EPHA3
实验是在 First Affiliated Hospital, Sun Yat-sen University 做的。可能的原因
那么,这样的多个表达量芯片数据集,同样的实验设计,但是他们的差异分析结果里面的基因交集很少,说明了什么呢?chatGPT告诉了我们可能有几种解释:
- 技术差异: 不同的芯片平台可能有不同的检测灵敏度、动态范围和特异性。这些技术差异可能导致每个平台检测到的差异基因集合存在差异。
- 生物差异: 即使实验设计相同,不同的生物样本可能存在生物差异,导致每个数据集中的差异基因不同。这可能是由于实验中的一些变化,如批次效应、个体差异或其他未知因素。
- 分析差异: 不同的分析方法和参数设置可能导致不同的差异基因集合。这可能包括数据预处理、差异表达分析算法、多重检验校正等方面的差异。
- 样本大小: 样本大小可能会影响差异分析的结果。如果某个数据集的样本量较小,可能会导致检测到的差异基因较少。
在解释这种情况时,建议进行一些深入的分析,例如:
- 检查每个数据集中的样本是否有明显的差异,比如批次效应、实验日期等。
- 比较每个数据集的数据质量和分布。
- 考虑使用不同的差异分析工具和参数,看是否会对结果产生影响。
- 进行更详细的生物学分析,看看是否有某些生物过程或通路在一个数据集中更为显著。
更好的对比
其实如果看具体的基因那么好难保证不同数据集的统一性,交集很少完全不能说明任何问题,因为我们不看质量看数量,而且我在生信技能树的教程:《你确定你的差异基因找对了吗?》提到过,必须要对你的转录水平的全局表达矩阵做好质量控制,最好是看到标准3张图:
- 左边的热图,说明我们实验的两个分组,normal和npc的很多基因表达量是有明显差异的
- 中间的PCA图,说明我们的normal和npc两个分组非常明显的差异
- 右边的层次聚类也是如此,说明我们的normal和npc两个分组非常明显的差异
如果分组在3张图里面体现不出来,实际上后续差异分析是有风险的。这个时候需要根据你自己不合格的3张图,仔细探索哪些样本是离群点,自行查询中间过程可能的问题所在,或者检查是否有其它混杂因素,都是会影响我们的差异分析结果的生物学解释。学徒作业
- 首先是同样的3次差异分析,但是针对3次分析的logFC结果两两之间汇总相关性散点图
- 其次真的3次差异分析后的基因排序(按照logFC)进行gsea分析后看不同数据集的通路的打分的两两之间汇总相关性散点图