目前的生命科学领域研究仍然是“草莽”阶段,基本上落脚点都需要是一个基因。可以是该基因在不同分组有差异,或者说该基因的高低跟肿瘤病人的生存情况关联起来了。有了基因,就可以对它进行敲减过表达各种干扰,看它的下游影响。但是会陷入一个有意思的玄学:过表达一个基因和敲除它的作用一定是相反的吗
虽然落脚点是一个基因,但是对它的量化也多层次的,比如某基因的突变情况咋样,是SNV还是CNV,该基因的表达量如何量化,该基因是否被甲基化修饰,该基因是否的蛋白产物是否正常。其中对我们来说,最简单的就是表达量啦,而在各个数据挖掘文章里面看到的量化表达量的技术目前以免疫组化和RNA-seq为主。
我们以2019年发表在《Cell Research》文章为例:《Protein C receptor is a therapeutic stem cell target in a distinct group of breast cancers》,链接是:https://www.nature.com/articles/s41422-019-0225-9
比如下面的PROCR这个基因在乳腺癌的不同亚型表达量有差异,就是免疫组化技术的结果 :
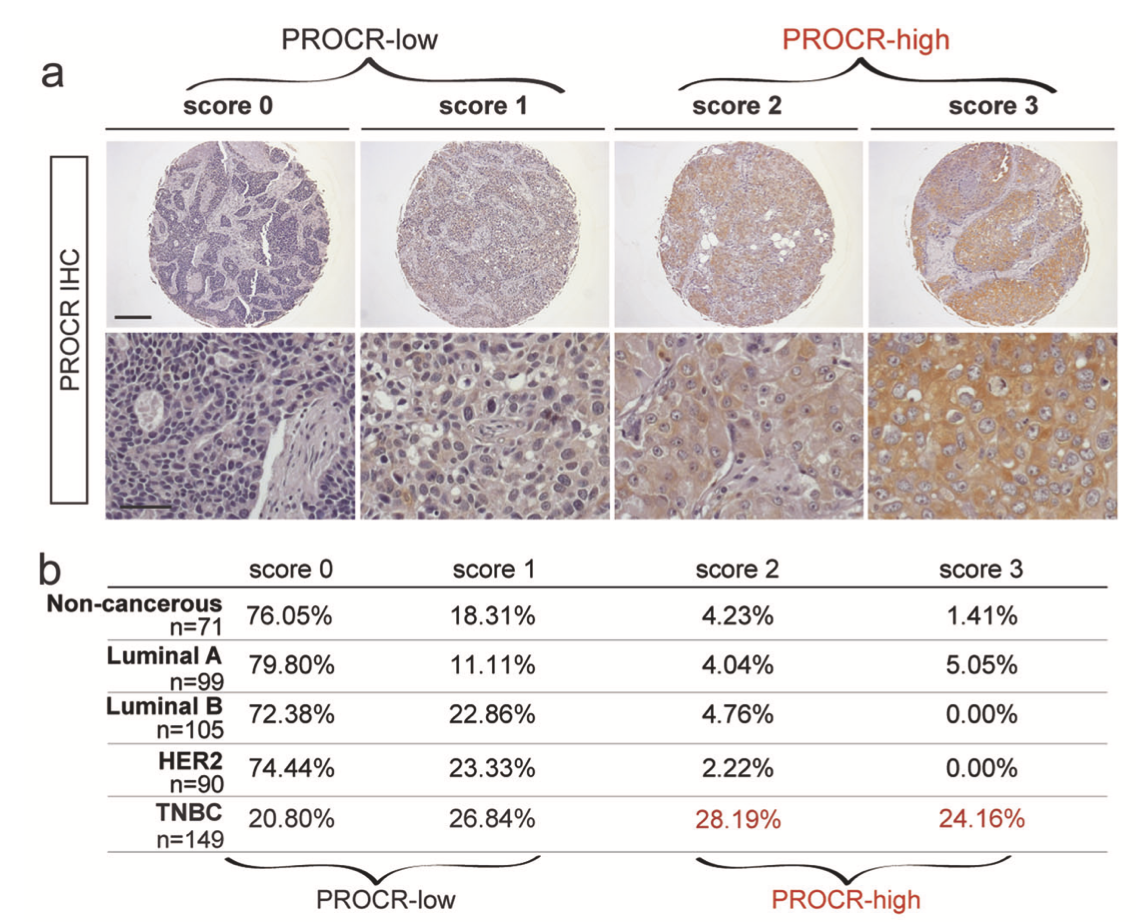
上面的 tissue microarrays (TMAs) 病人队列主要是根据 ER, PR, and HER2 的状态对乳腺癌患者进行区分:
- luminal A subtype (ER+ and/or PR+, low Ki67),
- luminal B subtype (ER+ and/or PR+, high Ki67 or HER2+),
- HER2+ subtype (HER2+, ER− and PR−),
- triple-negative subtype (ER−, PR−, and HER2−)
基本的结论就是PROCR这个基因在乳腺癌的癌症组织里面的表达量是高于正常对照无癌组织的,而且是在TNBC这个癌症里面特异性的高表达。
而且同样是这个 tissue microarrays (TMAs) 病人队列,他们临床预后信息收集整理的很好,所以可以做生存分析,发现这个PROCR基因在TNBC这个癌症里面特异性的高表达而且是风险因子,表达量越高的病人死亡的风险越大 :
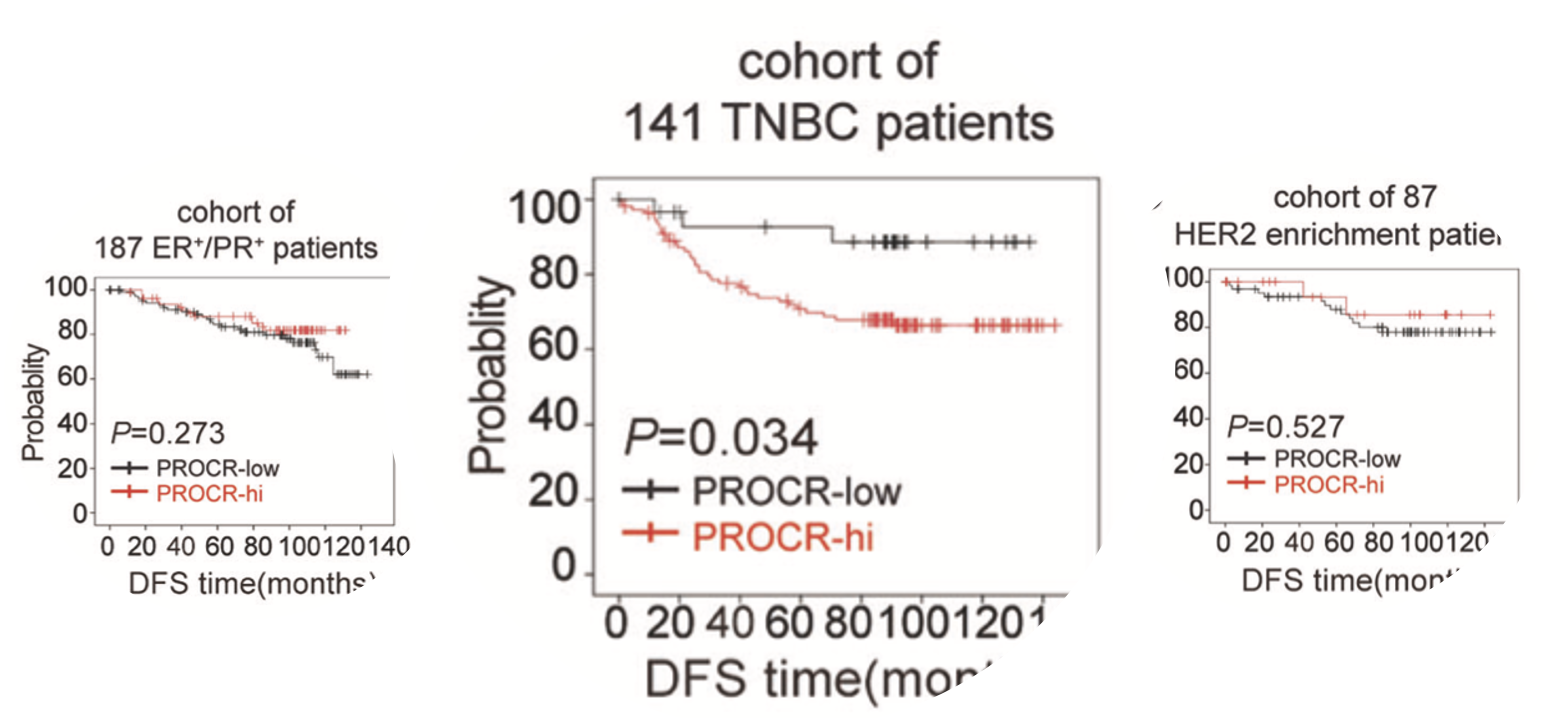
多么完美的结论啊,一个基因在三阴性乳腺癌里面高表达,而且呢,它表达量越高病人死亡风险越大。其实这样的基因超级多,只要你收集好一个病人队列,有他们全部的基因的表达量矩阵,以及全部病人的临床信息!以TCGA的RNA-seq数据为例子
我们可以去 https://xenabrowser.net/heatmap/ 简单的选取 TCGA Breast Cancer (BRCA) 队列,就可以看到,这个PROCR 首先并不是在肿瘤里面比正常对照高表达,其次也不是在TNBC亚型里面比其它乳腺癌表达量更高。最后生存分析也是一个玄学啊!
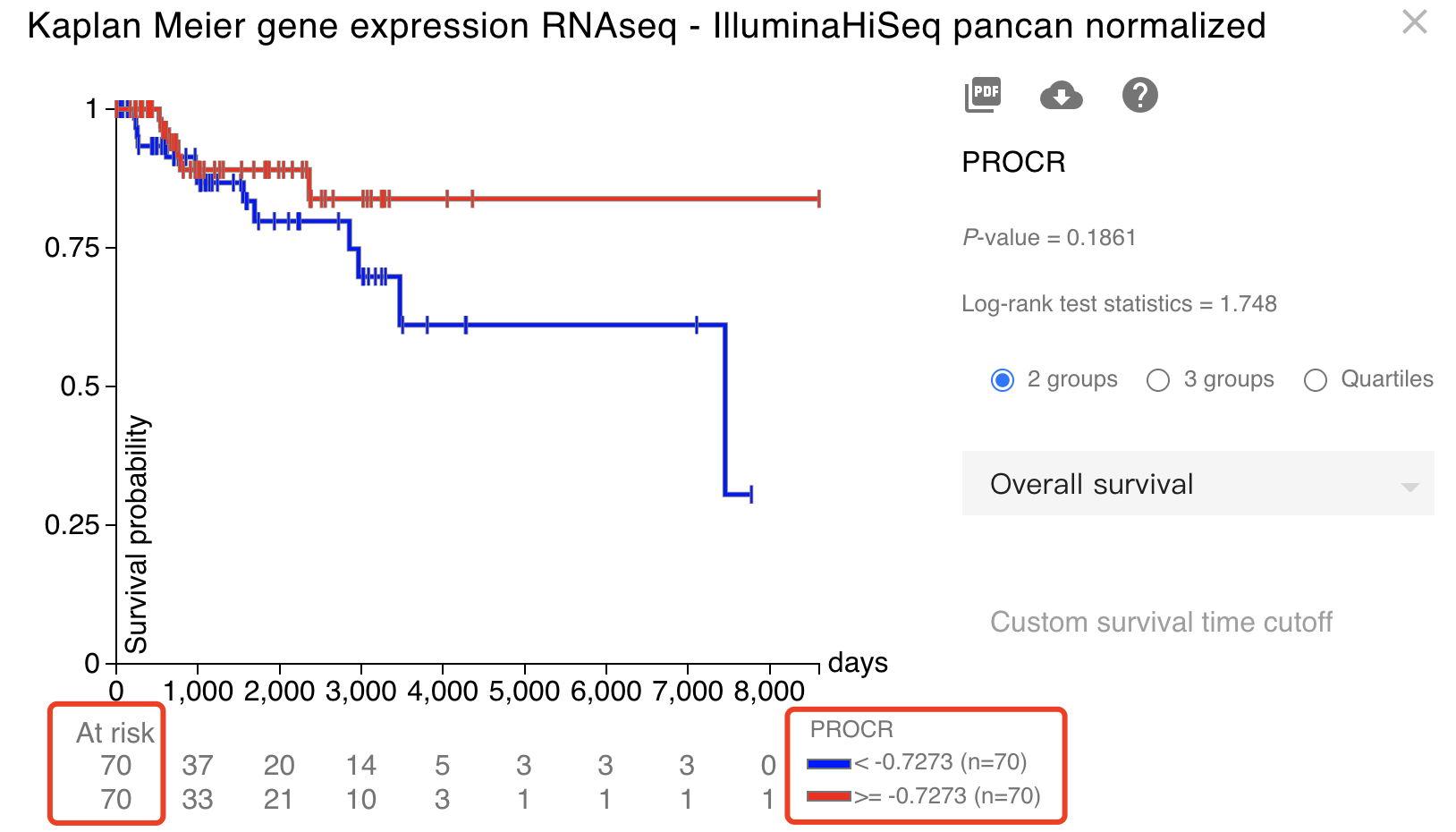
它在TNBC或者说basal队列里面,反而去表达量越低,死亡的风险越大!
是这个2019年发表在《Cell Research》文章的免疫组化的结论更可靠呢,还是说TCGA数据库里面的RNA-seq更可靠呢?
如果是TCGA数据库更好,以后大家的科研经费就可以大批量的省下来了,毕竟在医院收集好几百人的病人队列,取样做实验费用都很高啊,如果同样的结果可以是有数据库来说明,就可以跳过这个实验阶段了吧?学徒作业:
邵志敏科研团队发表了一个纯粹的TNBC队列, 在其文章:Multi-omics profiling reveals distinct microenvironment characterization and suggests immune escape mechanisms of triple-negative breast cancer 里面提到了数据:
The sequencing data is also available in GSE118527 (OncoScan), GSE76250 (HTA 2.0) and SRP157974 (WES and RNAseq)
有临床信息,也表达量矩阵,可以验证一下这个 个PROCR基因是不是在TNBC这个癌症里面特异性的高表达而且是风险因子,表达量越高的病人死亡的风险越大 。