看到很多小伙伴翻来覆去就是TCGA数据挖掘,而且绝大部分都仅限于转录组数据,虽然会根据基因性质拆分成为蛋白编码基因和非编码,但是数据多样性真的好有限。非常有必要系统性整理一些公共数据资源推广开。
比如在李程老师的交流群看到了Roy et al., 于2021年11月发表在, Immunity 杂志的文章:《DNA methylation signatures reveal that distinct combinations of transcription factors specify human immune cell epigenetic identity》,链接是:https://doi.org/10.1016/j.immuni.2021.10.001
就做了6种免疫细胞的850K甲基化芯片和转录组测序数据,数据都是公开是:
Methylation data GEO: GSE184269
RNASeq data This paper GEO: GSE184264
EBF1 Chip-Seq data GEO: GSE183537
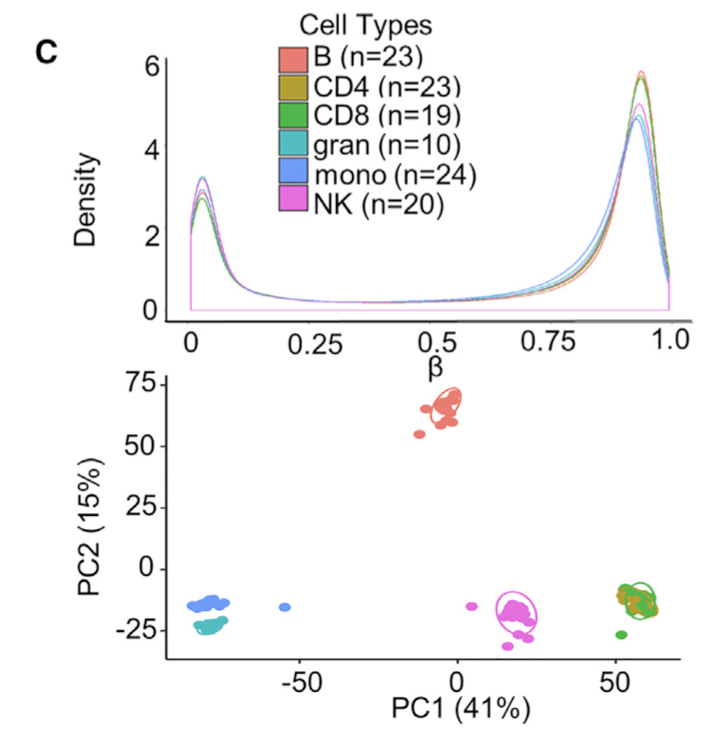
可以很清楚看到免疫细胞分类情况:
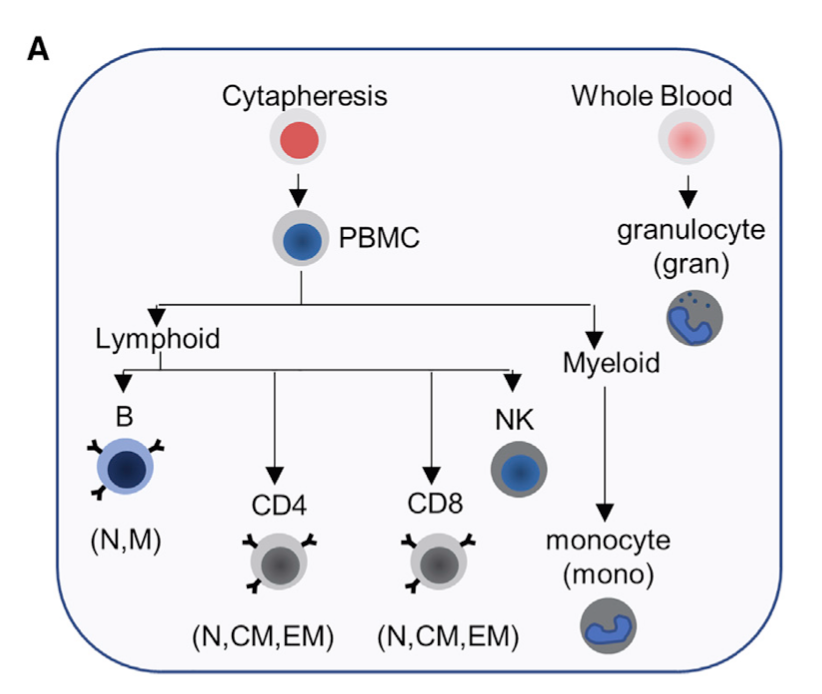
主要是淋巴系和髓系免疫细胞,做过单细胞转录组数据处理的小伙伴应该是很熟悉了,淋巴系(T,B,NK细胞)和髓系(单核,树突,巨噬,粒细胞)。
首先是6种免疫细胞的850K甲基化芯片数据
链接是;https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE184269 使用的是Infinium MethylationEPIC芯片,就是850K甲基化芯片,有167个样品:
差异分析策略如下所示:
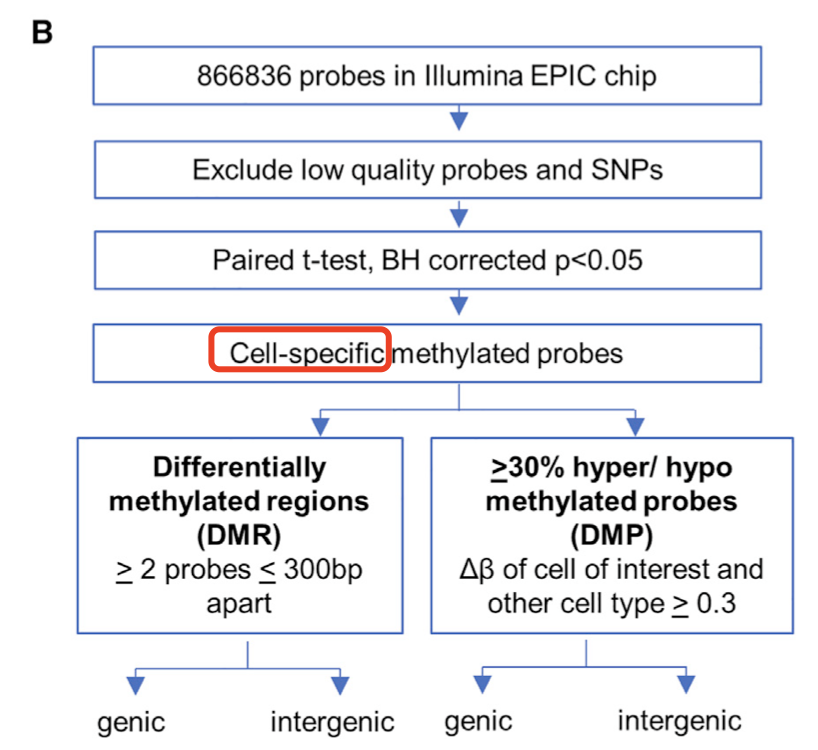
可以看到,每次差异分析都是取其中一个分组都是跟其它的全部样品进行差异分析,差异分析结果可视化:
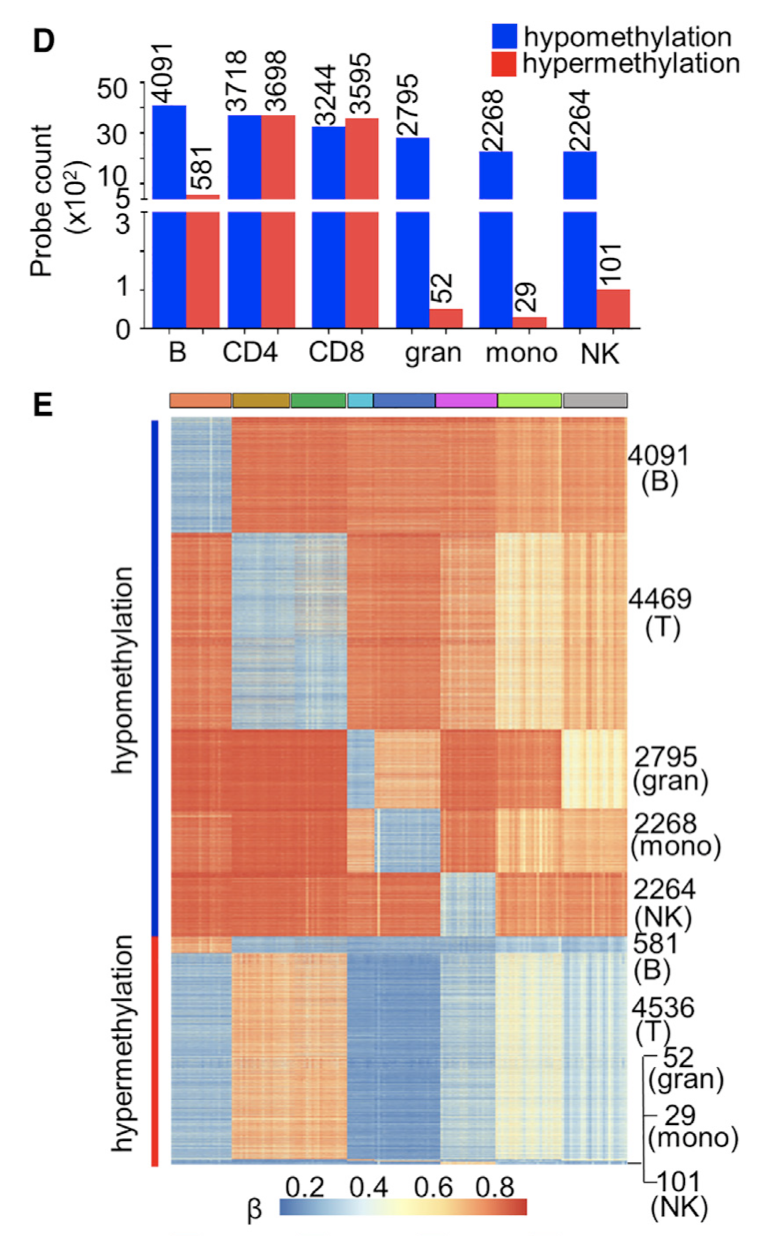
主要是统计了各个细胞亚群跟其它样品的甲基化差异数量,而且取这些位点进行热图可视化!
有差异的甲基化位点对应的基因的生物学功能数据库注释:
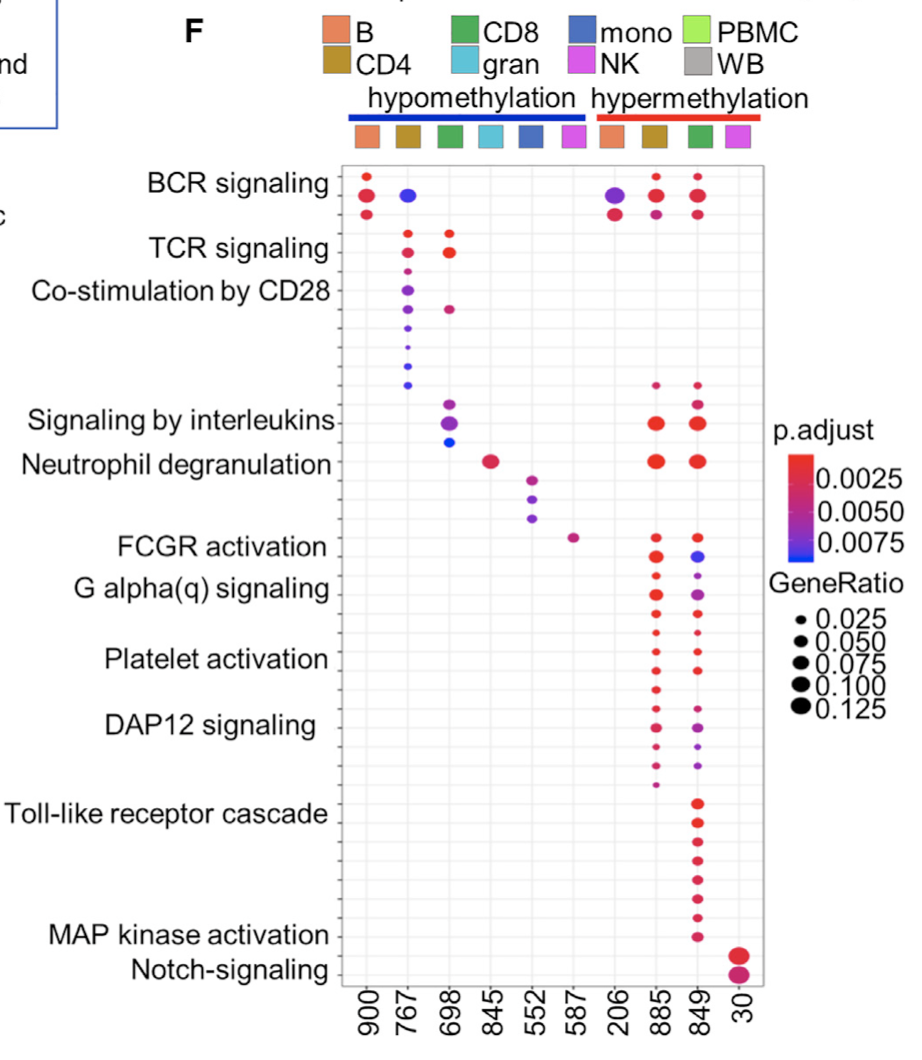
差异分析的前提是数据质量OK,哪怕是甲基化矩阵,也需要同样的质量控制 :
甲基化测序的 WGBS和RRBS,还有 芯片是最高频的甲基化技术,其中甲基化芯片数据处理我是有视频课程的,首先需要阅读我在生信技能树的甲基化系列教程,目录如下:
- 01-甲基化的一些基础知识.pdf
- 02-甲基化芯片的一般分析流程.pdf
- 03-甲基化芯片数据下载的多种技巧.pdf
- 04-甲基化芯片数据下载如何读入到R里面.pdf
- 05-甲基化芯片数据的一些质控指标.pdf
- 06-甲基化信号值矩阵差异分析哪家强.pdf
- 07-甲基化芯片信号值矩阵差异分析的标准代码.pdf
- 08-TCGA数据库的各个癌症甲基化芯片数据重新分析.pdf
- 09-TCGA数据库的癌症甲基化芯片数据重分析.pdf
- 10-TCGA数据辅助甲基化区域的功能研究.pdf
- 11-按基因在染色体上的顺序画差异甲基化热图.pdf
- 850K甲基化芯片数据的分析.pdf
- 使用DSS包多种方式检验差异甲基化信号区域.pdf
然后就可以看我在B站免费分享的视频课程《甲基化芯片(450K或者850K)数据处理 》 - 教学视频免费在:https://www.bilibili.com/video/BV177411U7oj
- 课程配套思维导图:https://mubu.com/doc/1cwlFgcXMg
然后是6大组蛋白的修饰的ChIP-seq数据
文章里面的ChIP-seq数据并不研究者自己产出的, 来自于 Roadmap 表观计划, 是:6 chromatin marks H3K4me3, H3K4me1, H3K36me3, H3K27me3, H3K9me3 and H3K27ac) ,
如果你对这些组蛋白修饰不理解,可以去参考2013年npg的一篇review:《Histone modifications for human epigenome analysis》,介绍了 不同组蛋白修饰的不同生物学功能哦。通常组蛋白修饰得到的是ChIP-seq数据,如果要分析: - 首先走上游ChIP-seq流程,即选取唯一比对的bam的序列拿去走MACS2找peaks
- 然后学会deeptools工具探索比对结果,及信号特征:包括信号热图,信号强度profile,样本相关性图
IGV可视化 - 学会 ChIPQC 和 DiffBind 两个R包
- bedtools对bam文件看 全基因组各个染色体的reads密度图
- peaks的数量,长度特特征,基因特征(外显子,内含子,UTR,启动子)注释及motif查找
如果想掌握上面的流程,也可以看我们《生信技能树》的B站免费NGS数据处理视频课程: - 免费视频课程《ChIP-seq数据分析》
- 免费视频课程《ATAC-seq数据分析》
最后是转录组测序数据
虽然中文仅仅是展现了差异基因的热图,但是背后提炼的信息也不少,而且跟差异甲基化位点进行了结合:
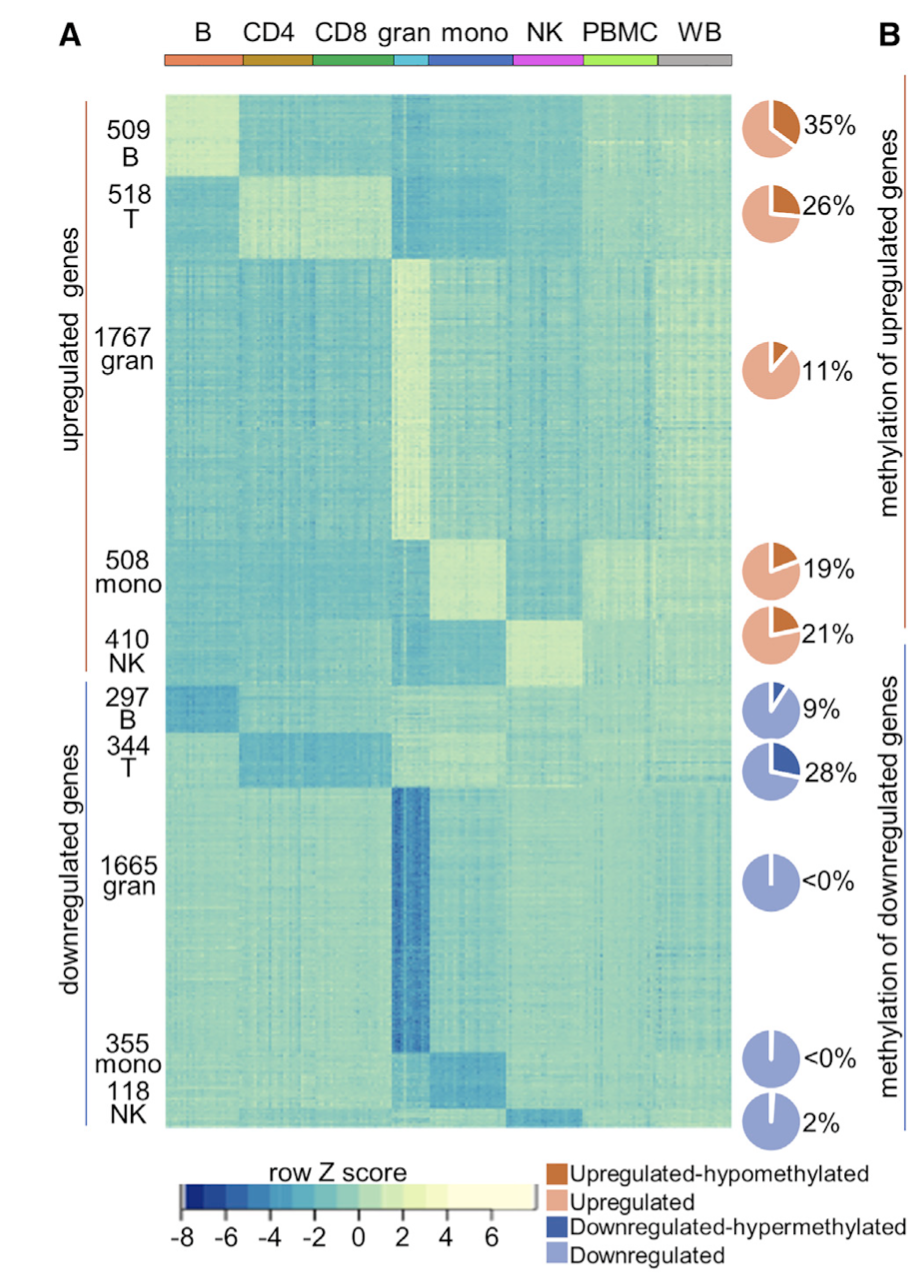
有意思的是这个热图的图例还蛮丰富的:
(A) RNA-seq was carried out with RNA from each cell type from 26 donors across the age range (Figures S6A and S6B). Normalized counts per million (CPM) values were compared pairwise within individuals using DESeq2 to identify differentially expressed genes. Genes with R4-fold change in a cell type compared to all other cell types within an individual and shared by all individuals irrespective of age are referred to as cell selective. Row Z score derived from normalized CPM for each cell type as well as PBMCs and whole blood are shown for each donor (columns). Darker shade in the pie charts show the proportion of cell-selective genes that are present in the MethylationEPIC array and contain cell-specific hypo- (orange) or hypermethylated (dark blue) sites.