看到一个很简单的临床工作文章,标题是:《Association of Body Mass Index With Somatic Mutations in Breast Cancer》,发表在 Front. Oncol., 01 April 2021 | https://doi.org/10.3389/fonc.2021.613933 ,纳入了421个乳腺癌患者,根据体重分成了3组:
- normal weight group (NW);
- overweight group (OW);
- underweight group (UW).
文章的测序数据上传到了咱们国产数据库,链接是:http://www.biosino.org/node/project/detail/OEP001295.
使用的肿瘤测序panel是:Supplementary Table S3. The genes list of 520 cancer-related genes (OncoScreen Plus) ,也是一个国产的panel。 有意思的是 - 纳入的病人队列:421 female breast cancer patients from the Guangdong Provincial People’s Hospital (GDPH), from June 1, 2017 to September 27, 2018.
- 但是A total of 3547 mutations were detected in 390 genes.
- 突变的分类:
- 1765 single nucleotide variants (SNVs),
- 1645 copy number (CN) amplifications,
- 54 insertions or deletions (Indels),
- 63 fusions, 15 deletions, and 5 large genomic rearrangements (LGR)
也就是说这个队列里面并不是所有的肿瘤测序panel里面的520个基因都有突变,病人汇总信息如下:
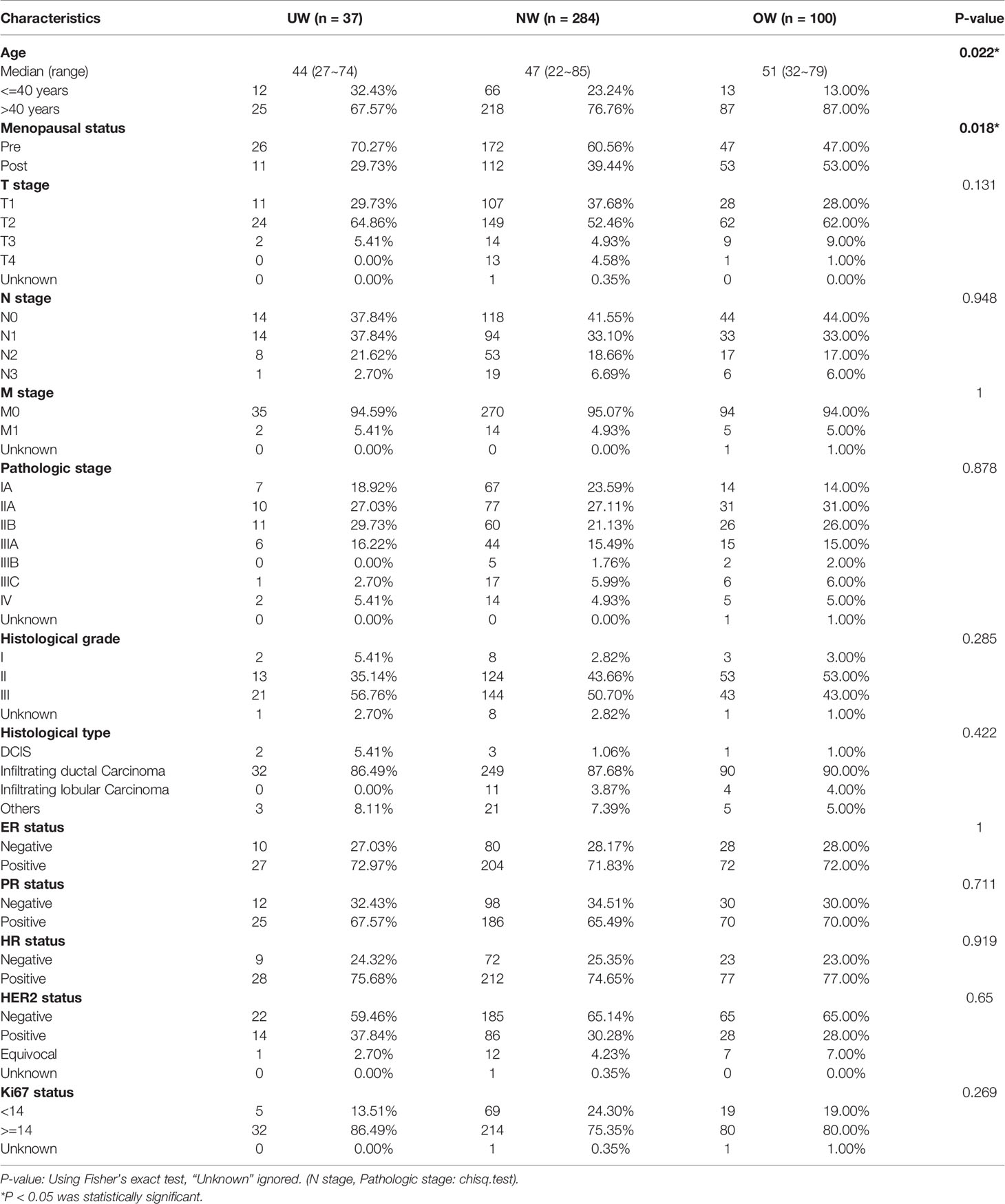
假如有这个文章的突变maf文件,倒是可以复现出文章的几个图表。学徒作业
自己去下载TCGA的somatic突变的maf文件,然后看看一千多个比如是否能找到体重信息进行分组,如果没有的话,就按照人种分组。作业就是尽可能的复现出文章图表,主要是统计可视化的理解。
另外,为了力求真实,需要大家首先过滤一下基因,因为文章使用的是肿瘤测序panel里面的520个基因,如下所示:
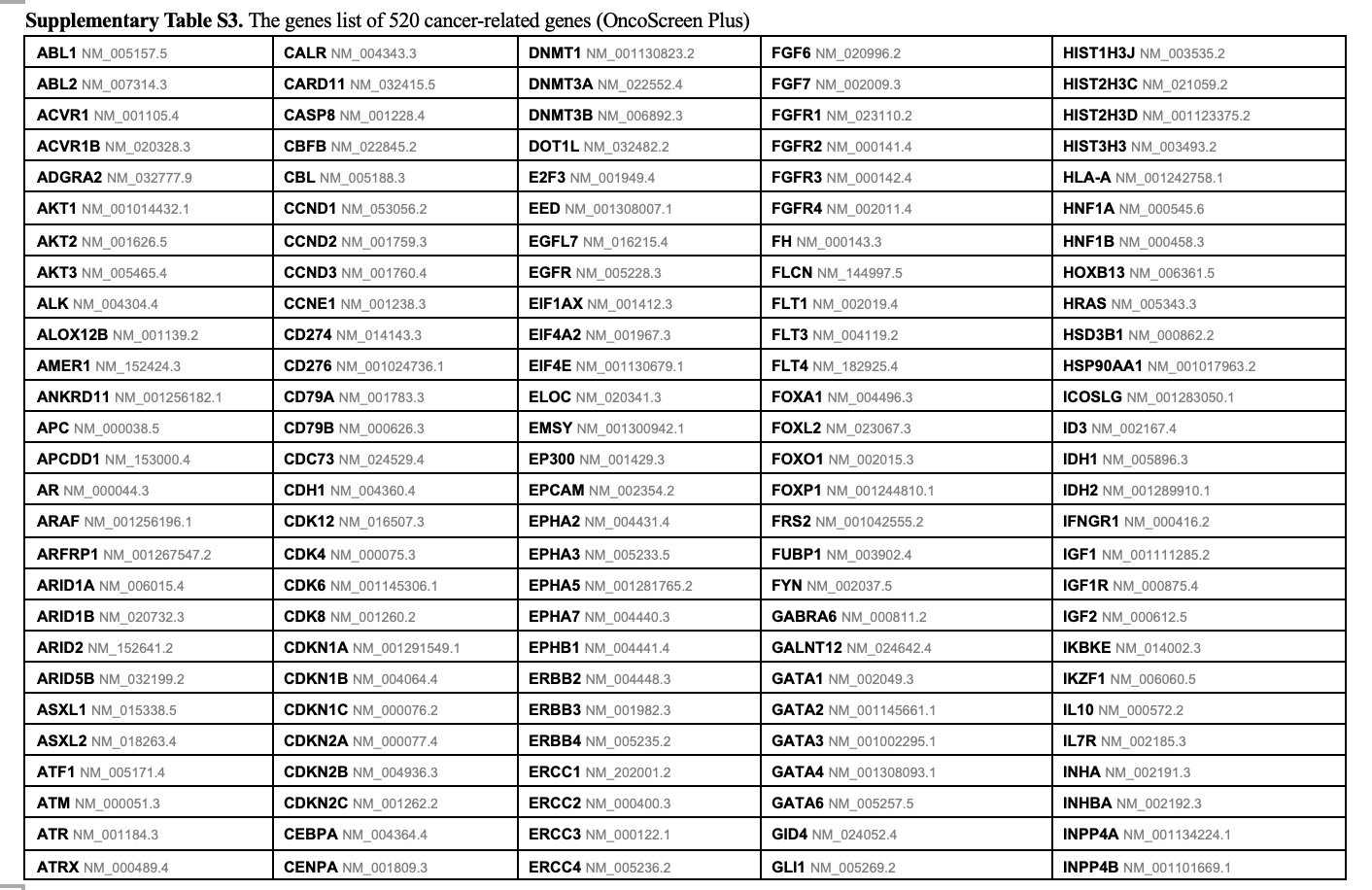
文章附件就有基因列表,很容易去过滤TCGA的somatic突变的maf文件。这个文章的缺陷很明显
其实是乳腺癌本身的异质性问题,不同亚型本来就是有突变层级的差异,体重的分组效应是小于乳腺癌亚型的。
但是作者在强调自己研究的几点限制性,提到了:
- First, all the enrolled patients were Chinese.
- The mutational landscape and genomic signatures differ across ethnicities
我觉得这个并不是什么很严重的限制,反而绝大部分中国人的科研成果都是把这一点强调为优点啊!因为弥补了国际上对于这个人群研究的不足!所以问题来了,这个到底是优点还是缺点呢?难道正说反说都有理?
但是作者在第三点确实是实话实说了: Third, we were unable to perform statistical analysis for investigating the survival and prognosis of these patients, because all the patients were followed-up for less than five years. 也就是说这个数据集不能做生存分析,那么这样的研究的临床意义就大打折扣,也很难进行二次挖掘了。
不过呢,咱们有公共数据库啊,TCGA的BRCA队列很难直接使用,因为人种不一样,说服力会很勉强。