我们在前面的 明码标价之公共数据库探索 提到了文章作者比较了TCGA数据库的ESCC的N0和N3时期的肿瘤样品表达量样品的差异,从而开启一个科研项目,让大家误会了,以为那个文章就这么简单。
其实这个数据挖掘步骤仅仅是该文章的一个引子,后续关于TGFβ2的探索也很精彩啊,该研究于2020年发表在《 Adv Sci 》杂志的文章《Direct Targeting of CREB1 with Imperatorin Inhibits TGFβ2‐ERK Signaling to Suppress Esophageal Cancer Metastasis》,链接是:https://onlinelibrary.wiley.com/doi/full/10.1002/advs.202000925 。
前面的公共数据库挖掘定位到了TGFβ2这个关键基因,然后各种实验验证了TGFβ2是如何 Promotes ESCC Invasion and Metastasis的,后面就可以顺理成章的过渡到针对TGFβ2的靶向药物筛选,并且可以探索药物靶向的机理。
TGFβ2的靶向药物筛选
流程图如下所示,涉及到了一个药物筛选库:
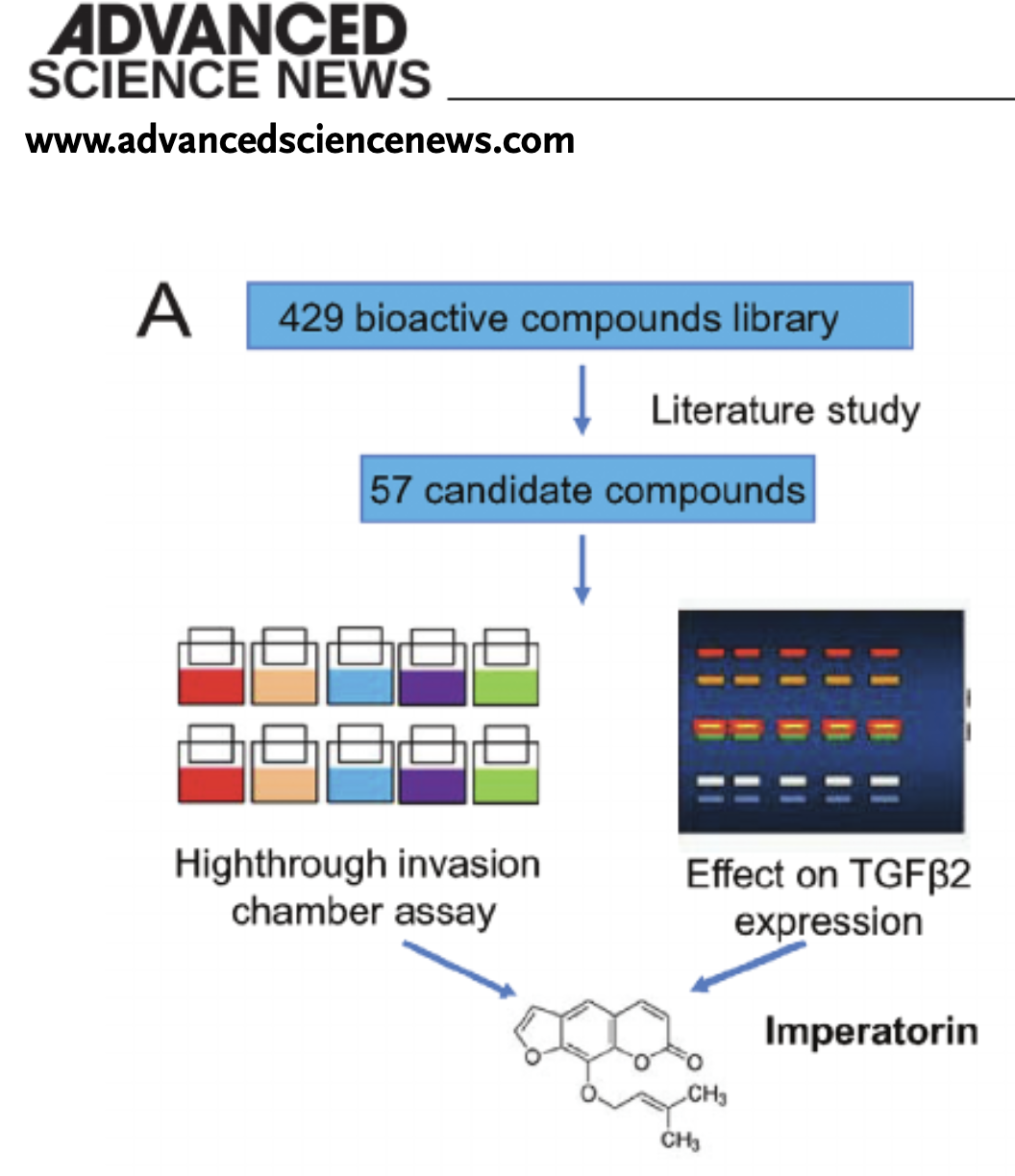
然后就定位到了 白茅苷 这个药物。
接下来就集中精力在白茅苷 这个药物,为了研究它抗肿瘤的潜在机制,对ESCC细胞系做该药物处理前后的蛋白质测序。
药物处理前后蛋白质组学
蛋白质测序技术是: Stable Isotope Labeling by Amino Acids in Cell Culture (SILAC)-based quantitative proteomics,筛选到2963个定量信息的蛋白,其中有127个差异表达蛋白。对127个差异蛋白进行网络分析,其中TGF2-ERK信号转导在白茅苷的作用机制中发挥枢纽作用,如下所示的网络图:
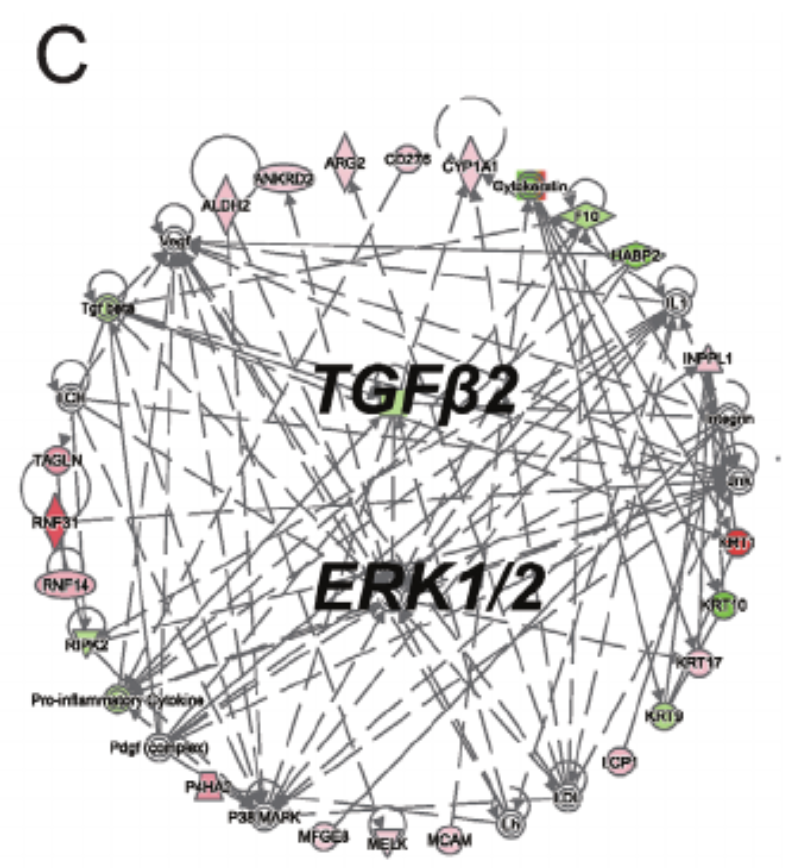
一般来说,从差异分析到功能富集并不会那么的顺利,这个时候仍然是无法排除研究者们先入为主的选择性探讨他们感兴趣的通路的可能性哦!
正常的差异分析到功能富集看我六年前的笔记即可:
- 解读GEO数据存放规律及下载,一文就够
- 解读SRA数据库规律一文就够
- 从GEO数据库下载得到表达矩阵 一文就够
- GSEA分析一文就够(单机版+R语言版)
- 根据分组信息做差异分析- 这个一文不够的
- 差异分析得到的结果注释一文就够
如果一定要验证这个通路非常重要,需要更多的实验,比如研究者们就是使用U0126阻断MEK信号显著废除了TGF2对ESCC细胞系侵袭的促进作用和EMT表型(Figure 3F,G)药物如何发挥作用呢
还需要进一步探究白茅苷调节TGF2的分子机制,结果显示白茅苷显著降低了TGF2的mRNA表达。构建TGF2启动子区域的克隆载体,发现白茅苷一剂量依赖的方式降低了TGF2启动子的转录活性(Figure 5A,B)。这些暗示着白茅苷可能通过与TGF2启动子相互作用,负调控TGF2的表达。此外,作者检测了白茅苷处理后的转录因子的表达,但是除了CREB1表达呈剂量依赖性下调外,其余的表达不受白茅苷处理的影响(Figure 3C)。重要的是,核转录因子CREB1的表达被白茅苷显著抑制,提示CREB1在白茅苷调控TGF2转录扮演着重要作用(Figure 5D)。
接下来,检测白茅苷是否直接靶向CREB1,鉴于其蛋白结构至今未有报道,所以使用SWISS预计CREB1的蛋白3D结构模型(Figure 5E)。分子对接与动力学模拟显示白茅苷可以与CREB1蛋白相互作用,且很大可能是通过疏水区的Lys-304和Lys-309相互作用或者Lys-305与形成氢键(Figure 5F)。为了验证这个猜测,作者纯化了CREB1蛋白,CREB1K304E和CREB1K305E蛋白,这两个蛋白的Lys-304和Lys-305已经突变了,而CREB1K309E蛋白的纯化失败了(Figure 5G)。表面等离子(SPR)实验显示白茅苷可以与CREB1结合,而当Lys-304和Lys-305区域突变后结合失败了(Figure 5H)。这些结果为白茅苷优先结合野生型CREB1蛋白提供了直接证据。
为了检测白茅苷与CREB1相互作用的生物学功能,进行了ChIP实验,结果显示CREB1和TGF2启动子的相互作用被白茅苷显著抑制(Figure 5I)。双荧光素酶实验显示白茅苷显著降低了TGF2启动子的活性,但当与CREB1结合时这种作用被废止了(Figure 5J)。这些结果表明白茅苷直接靶向抑制CREB1蛋白结合TGF2启动子随后抑制了TGF2的转录和表达。靶基因的重要性
接下来又得证明 CREB1 基因的重要性!但是我们前面提到过:如果依据ENCODE的ChIP-seq数据结果来查询对应的基因的靶基因,会发现有1.3万基因都是CREB1 的靶基因!见:什么?1.3万基因都是你的靶基因? 它的重要性其实不言而喻了。
首先, CREB1 基因在肿瘤里面具有较高的表达量,而且呢, CREB1 基因的高表达量对应的病人死得快,如下所示:
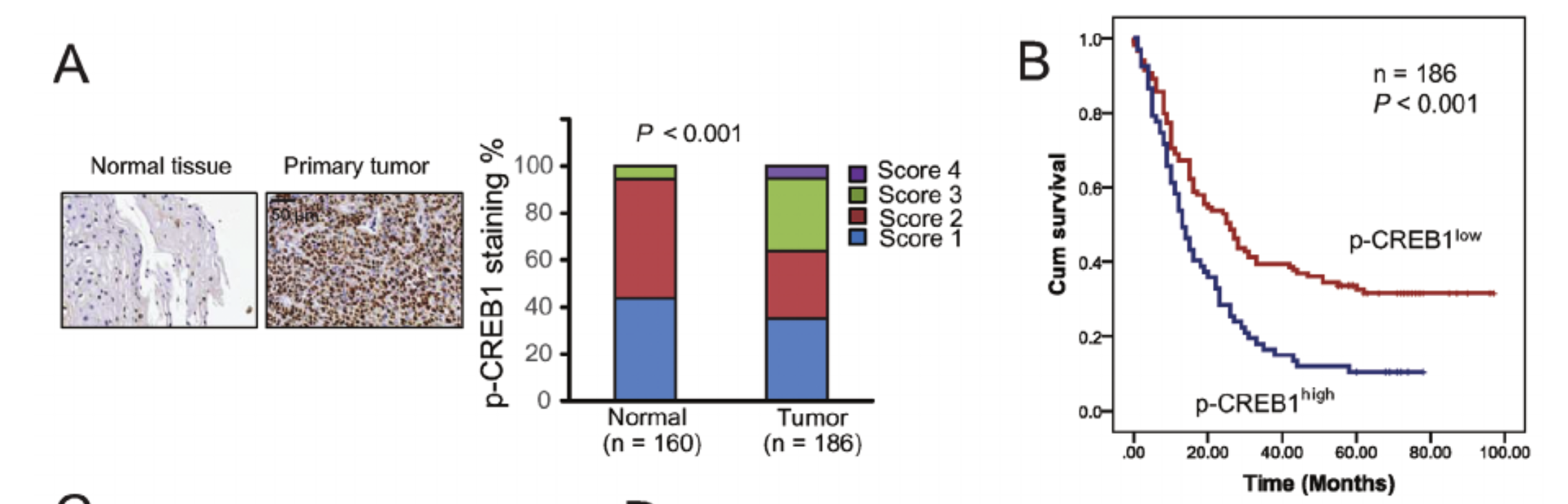
看完了肿瘤和正常组织的 CREB1 基因的差异,还可以看它跟肿瘤转移的差异,非常巧的是,它恰好是在转移瘤表达量又高于原位肿瘤,如下所示:
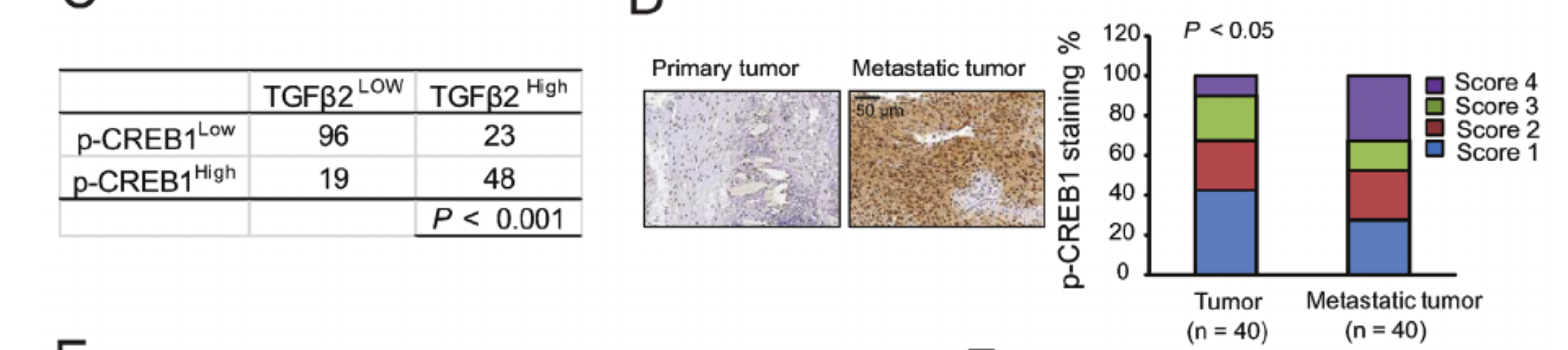
你说巧不巧呢?
但是,这个时候,如果为了节省经费,直接去使用TCGA数据库,可能就得不到这样的结论了,我看了看:https://xenabrowser.net/
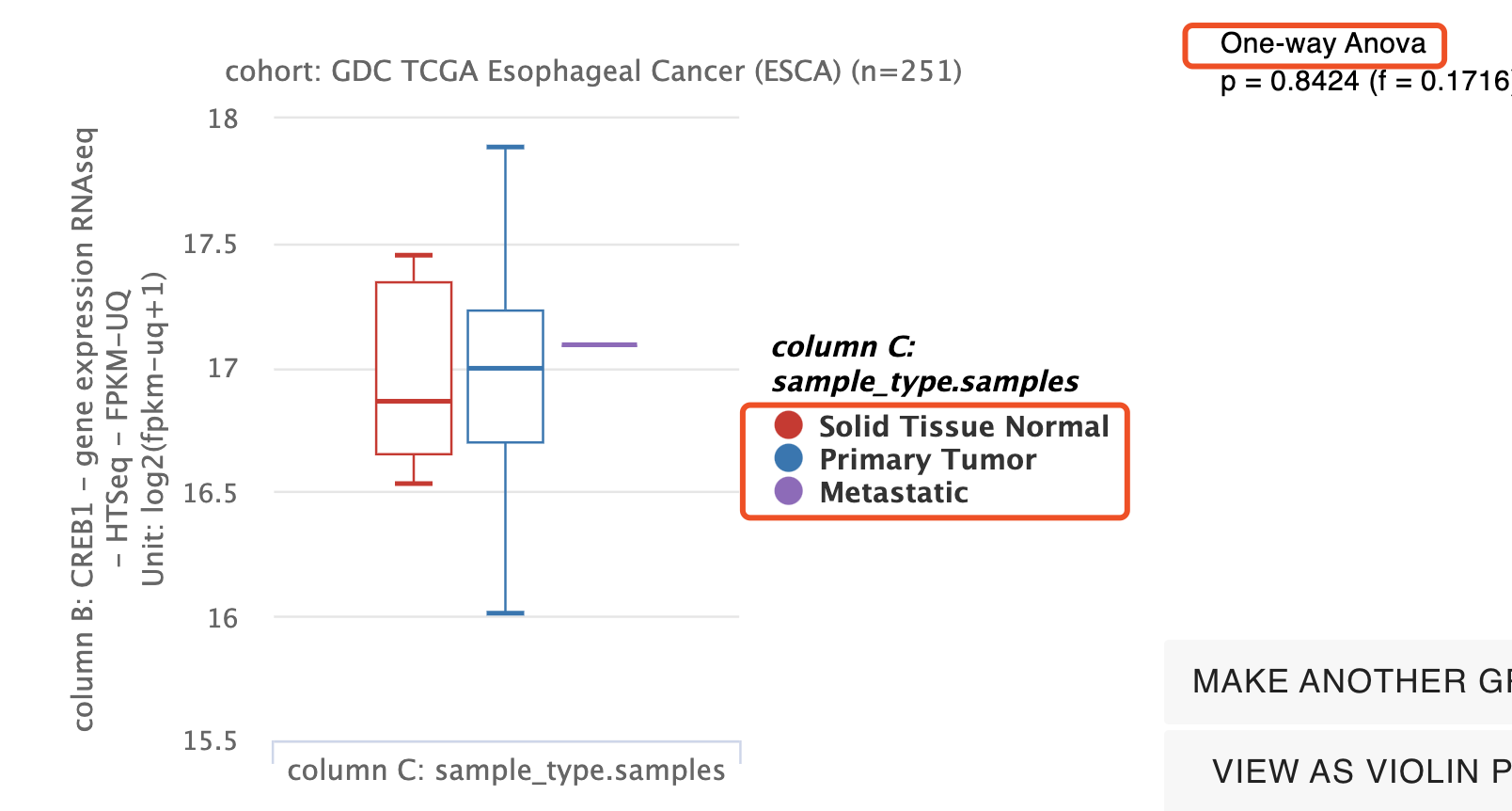
可以看到,在TCGA数据库的ESCA这个癌症里面,肿瘤里面的CREB1 基因的表达量确实是比正常组织高,但其实并没有达到统计学显著。
唉,感觉这样的科研跟套娃一样。