一般来说,我们进行数据库注释,基因数量在20到500之间,其实100个左右是比较理想的!比如使用Y叔的clusterProfiler进行gsea分析,就有 minGSSize = 10, 和 maxGSSize = 200, 的设置,全部代码如下所示:
rm(list = ls())
options(stringsAsFactors = F)
lapply(c('clusterProfiler','enrichplot','patchwork'),
function(x) {library(x, character.only = T)})
# Please go to https://yulab-smu.github.io/clusterProfiler-book/ for the full vignette.
data(geneList, package="DOSE")
#4312 8318 10874 55143 55388 991
#4.572613 4.514594 4.418218 4.144075 3.876258 3.677857
class(geneList)
#[1] "numeric"
kk2 <- gseKEGG(geneList = geneList,
organism = 'hsa',
nPerm = 10000,
minGSSize = 10,
maxGSSize = 200,
pvalueCutoff = 0.05,
pAdjustMethod = "none" )
- gseKEGG输入形式:将基因按照logFC进行从高到低排序,只需要基因列和logFC
- organism:物种,http://www.genome.jp/kegg/catalog/org_list.html
- nPerm:permutation numbers
- minGSSize:通路最小基因数
- maxGSSize:通路最大基因数
- pvalueCutoff:最小p值
- pAdjustMethod:p值校正方法,”BH”
一般可通过改变minGSSize,maxGSSize数目调整通路大小,但是默认设置肯定是有自己的道理。
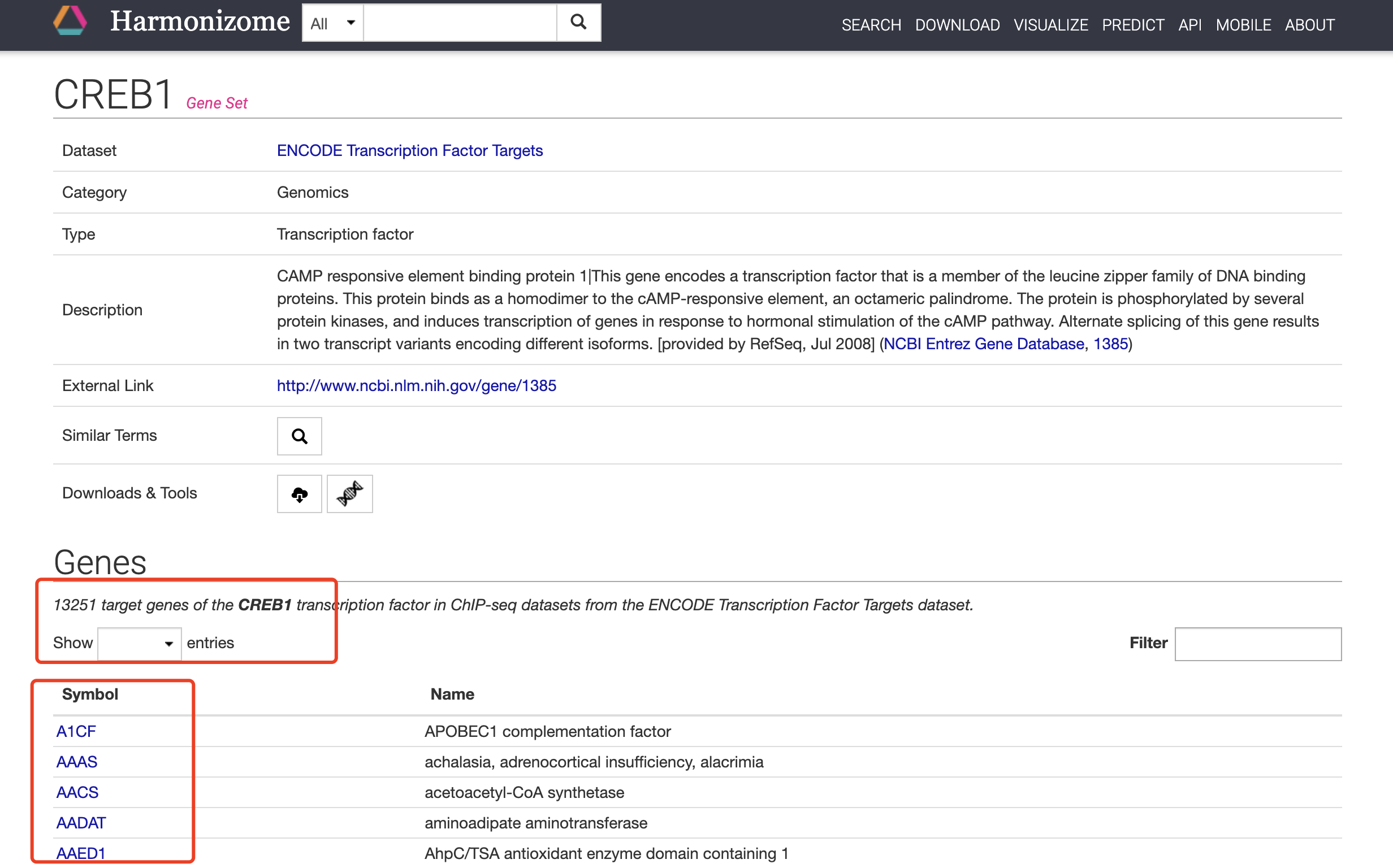
但是最近有粉丝在交流群里提问, 他做一个基因CREB1的靶基因调控网络,但是节点太多了,导入cytoscape就奔溃,希望有一个解决方案。我简单看了看,他使用的数据库:Harmonizome ,可以依据ENCODE的ChIP-seq数据结果来查询对应的基因的靶基因:
确实啊! 13251 target genes of the CREB1 transcription factor in ChIP-seq datasets from the ENCODE Transcription Factor Targets dataset.
也就是说,非常的震惊,1.3万基因都是CREB1 的靶基因!
另外,这个基因有一个网页工具数据库,早在2005就发表在了PNAS杂志:Genome-wide analysis of cAMP-response element binding protein occupancy, phosphorylation, and target gene activation in human tissues
数据库链接是: http://natural.salk.edu/CREB/ ,有意思的是,那个时间窗口(2005)是根本就没有ChIP-seq这样的技术来找其靶基因的,还是处于芯片早期发展阶段。
主要是一个ChIP-chip和一个表达量芯片的数据,这个工具就是整合两个数据结果,供读者查询罢了。
那么,问题来了,ChIP的技术看结合,与敲减基因来干扰基因表达获取靶基因哪个好?
欢迎畅所欲言!
如果是干扰基因表达获取靶基因通常是差异分析
简单的差异分析看我六年前的表达芯片的公共数据库挖掘系列推文即可哈 :
- 解读GEO数据存放规律及下载,一文就够
- 解读SRA数据库规律一文就够
- 从GEO数据库下载得到表达矩阵 一文就够
- GSEA分析一文就够(单机版+R语言版)
- 根据分组信息做差异分析- 这个一文不够的
- 差异分析得到的结果注释一文就够
如果是ChIP的技术看结合
我们也有免费视频课程《ChIP-seq数据分析》,视频观看方式
- 首先视频免费共享在B站:https://www.bilibili.com/video/BV16s411T7Fh
- ChIP-SEQ实战演练的素材:链接:https://share.weiyun.com/53CwQ8B 密码:ju3rrh, 包括一些公司PPT,综述以及文献
- ChIP-SEQ 实战演练的思维导图:文档链接:https://mubu.com/doc/11taEb9ZYg 密码:wk29
目前,上面的链接都是亲测有效的,如果你看完发现链接无法打开,说明已经里面被举报而封杀了,只能是去交流群拿到最新链接了。