肿瘤微环境这个热点应该不仅仅是集中在免疫细胞,其实还有基质细胞,其中热度最高的基质细胞应该是Cancer-associated fibroblasts (CAFs) 。
但是目前呢,学界对CAFs的来源本来就是并不那么清晰,理论上不可能存的单一的标记基因来区分出来CAFs。通常CAFs有4种来源:
- The primary source is normal local fibroblasts, which are activated by stimuli from the tumor microenvironment.
- Mesenchymal stem cells (MSCs) and other mesenchymal precursor cells are other sources.
- Endothelial cells and epithelial cells do not belong to the fibroblast lineage, but they could transdifferentiate into CAFs-state cells.
- Finally, a self-renewable CAFs-stem cell population might exist in the hierarchical organization, and these cells share similar characteristics as MSCs.
如果要筛选CAFs,首先要去除4个基因表达量为阳性的细胞亚群 : - CD31 (an endothelial marker)
- CD45 (a hematopoietic cell marker),
- desmin (a smooth muscle cell marker),
- EPCAM (epithelial cell adhesion molecule, an epithelial cell marker).
然后各种文献整理一下,发现它的阳性标记基因可能是有:α-SMA, Collegen1A1, FAP, FSP1, PDGFRα and PDGFRβ, Podoplanin, and vimentin, 这么多。
以上资料整理来源于新鲜出炉的综述:Front. Cell Dev. Biol., 04 February 2021 | https://doi.org/10.3389/fcell.2021.613534 标题是:《Cancer-Associated Fibroblasts Suppress Cancer Development: The Other Side of the Coin》,前面我们在推文 细胞亚群的特异性标记基因也许真的很难提到的Cancer-associated fibroblasts (CAFs)是比较难以精确的细分亚群。
有单细胞测序手段的时候,尚且如此难弄清楚Cancer-associated fibroblasts (CAFs),那么在没有单细胞的年代,到底该如何研究它呢?
我看到了一个2011的文章:《Prognostic gene-expression signature of carcinoma-associated fibroblasts in non-small cell lung cancer》,在 Proc Natl Acad Sci U S A 2011 Apr 26;108(17):7160-5. PMID: 21474781
We established primary cultures of CAFs and matched normal fibroblasts (NFs) from 15 resected NSCLC.
数据在:https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE22874
包括了两个表达量芯片数据集:GSE22862 [expression profiling_CAFs] GSE22863 [expression profiling_NSCLC stroma]每个数据集都是30个样品,这里面的分析可以有很多种花样,但是我看了看文献里面的差异基因的热图,有点像是强行找差异。
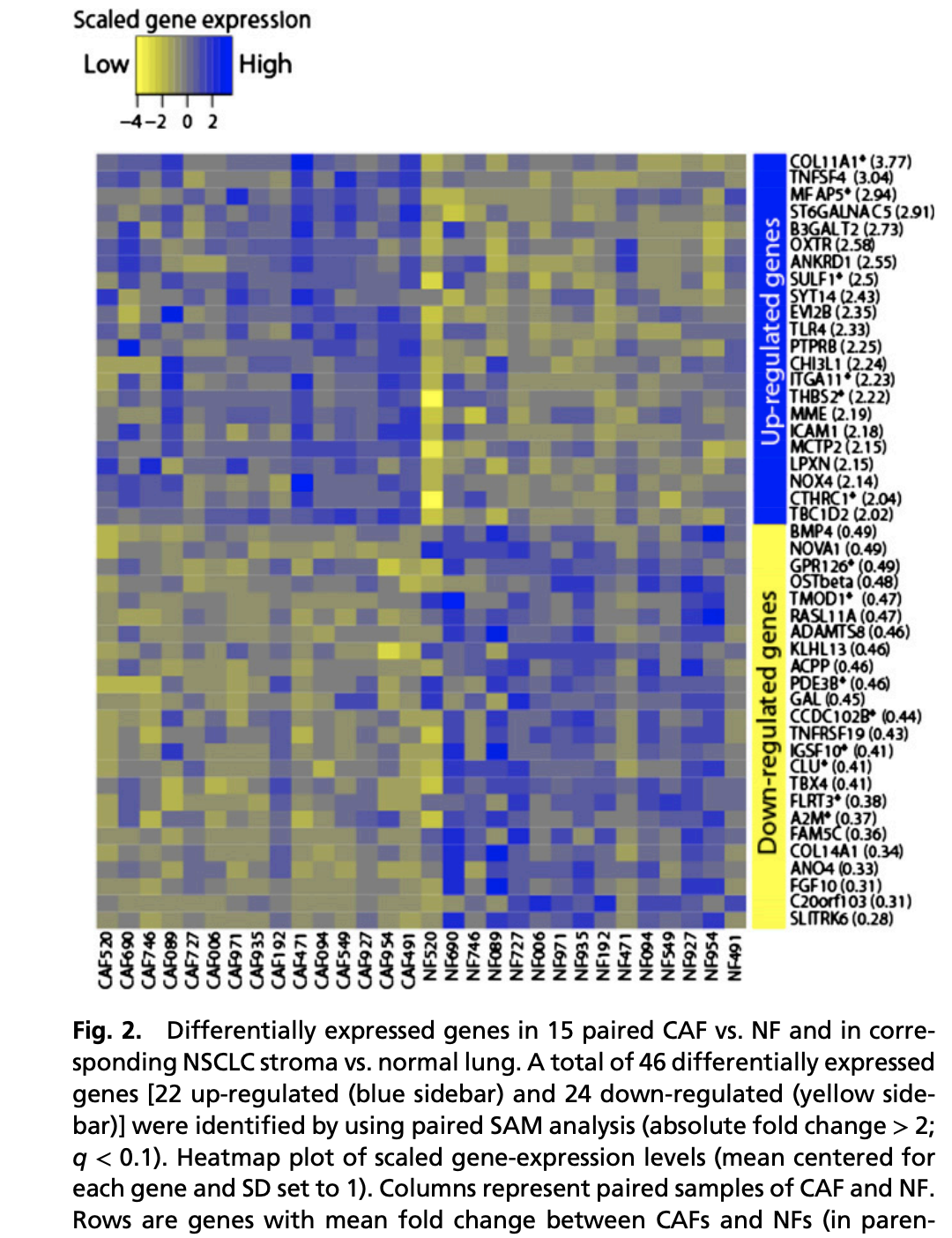
学徒作业
差异分析相信大家都不陌生了,基本上看我六年前的表达芯片的公共数据库挖掘系列推文即可;
- 解读GEO数据存放规律及下载,一文就够
- 解读SRA数据库规律一文就够
- 从GEO数据库下载得到表达矩阵 一文就够
- GSEA分析一文就够(单机版+R语言版)
- 根据分组信息做差异分析- 这个一文不够的
- 差异分析得到的结果注释一文就够
首先你需要完成前面提到的GSE22874 里面的2个表达量芯片数据集各自的差异分析,然后呢,2019的这个单细胞转录组数据集GSE117570就是4个NSCLC病人的N-T配对测序,理论上,前面提到的GSE22874数据集里面的分析,应该是可以在GSE117570这个单细胞转录组数据集里面验证一下。
这个难度有点大!