总是有粉丝询问3个分组的两两之间差异分析如何弄这样的问题,其实“师傅领进门,修行在个人”,我们讲解了方法并且给予的全部的学习资源,理论上后续应该是大家各凭本事,做好自己的科研。
limma包的说明书有200多页,它是有值得花时间的必要性!
首先创造一个有着3分组表达量矩阵
代码如下:
library(limma)
y <- matrix(rnorm(10000*9),10000,9)
fivenum(y)
(group=rep(LETTERS[1:3],each=3) )
y[sample(1:10000,1000),1:3]=y[sample(1:10000,1000),1:3]+2
y[sample(1:10000,1000),4:6]=y[sample(1:10000,1000),4:6]+2
y[sample(1:10000,1000),7:9]=y[sample(1:10000,1000),7:9]+2
library(pheatmap)
# pheatmap(y )
可以看到,我给这3个分组的表达量各自随机挑选了1000个基因进行系统性的提高,如下所示:
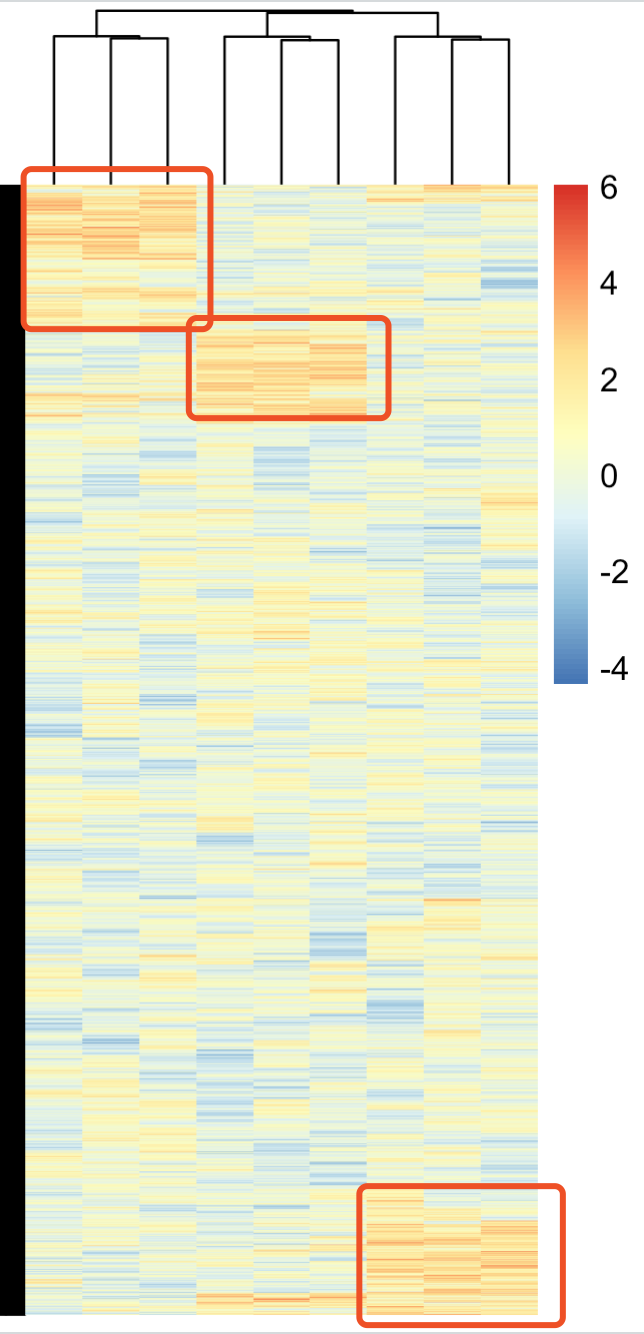
接下来进行3分组的两两之间差异分析
直接使用limma包即可:
design <- model.matrix(~0+group)
colnames(design) <- gsub("group", "", colnames(design))
design
contr.matrix <- makeContrasts(
AVSB = A-B,
AVSC = A-C,
BVSC = B-C,
levels = colnames(design))
contr.matrix
vfit <- lmFit(y, design)
vfit <- contrasts.fit(vfit, contrasts=contr.matrix)
efit <- eBayes(vfit)
plotSA(efit)
colnames(efit)
summary(decideTests(efit))
可以看到, 各自的差异基因数量;
> summary(decideTests(efit))
AVSB AVSC BVSC
Down 311 250 278
NotSig 9418 9517 9419
Up 271 233 303
虽然我前面是统一给每个分组各自1000个基因的干扰,但是实际上,它的效应会被这个表达量矩阵本来的随机性给叠加,所以这样的结果是可以理解的。
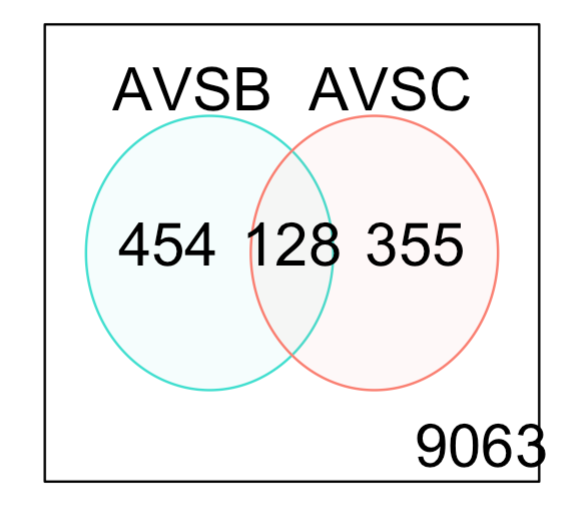
也可以随便挑选两个差异分析结果取交集看看:
dt <- decideTests(efit)
summary(dt)
de.common <- which(dt[,1]!=0 & dt[,2]!=0)
length(de.common)
vennDiagram(dt[,1:2], circle.col=c("turquoise", "salmon"))
如下所示:
这3次差异分析的结果都是可以独立取出来了:
colnames(efit)
# [1] "AVSB" "AVSC" "BVSC"
AVSB <- topTreat(efit, coef=1, n=Inf)
AVSC <- topTreat(efit, coef=2, n=Inf)
BVSC <- topTreat(efit, coef=3, n=Inf)
head(AVSB)
head(AVSC)
是不是很方便?
思考题
难道3个分组,仅仅是有两两组合的这样的3种形式的差异分析吗?
上面的代码大量涉及到R基础知识:
需要把R的知识点路线图搞定,如下:
- 了解常量和变量概念
- 加减乘除等运算(计算器)
- 多种数据类型(数值,字符,逻辑,因子)
- 多种数据结构(向量,矩阵,数组,数据框,列表)
- 文件读取和写出
- 简单统计可视化
- 无限量函数学习