最近看到了很多借助 单细胞水平的不同细胞亚群的差异来解释以前的传统转录组混合各种细胞亚群的样品差异的文章, 如下所示:
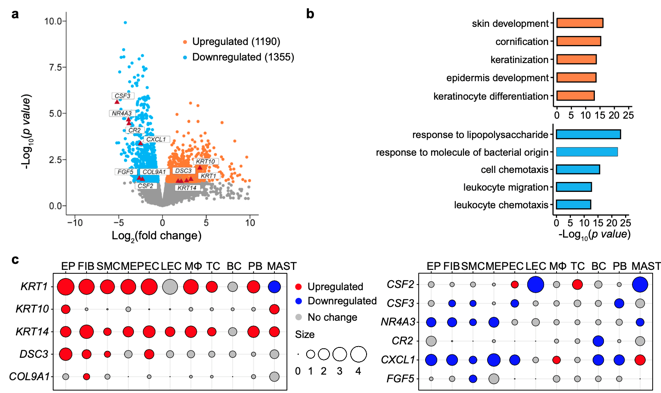
研究者首先做了一个bulk转录组,走了标准的差异分析,拿到了上下调基因以及注释它们的功能。然后把这些基因在自己的单细胞转录组各个亚群具体看其是否有表达差异,发现异质性很大,以前拿到的混合状态的差异其实是细胞亚群的比例差异而已。
图来源于文章:NATURE COMMUNICATIONS | (2021)12:87 | https://doi.org/10.1038/s41467-020-20358-y
我就在思考,这完全是颠覆了以前数以万计的芯片和转录组测序项目的结论啊!而且这样的数据挖掘思路,又可以成为一个风靡中国医生群体的生物信息学灌水策略了。
我特意看了看这样的策略是否有人采用,其中语言 一个对GSE88715数据集的挖掘,文章标题是;《Identification of Key Genes Potentially Related to Triple Receptor Negative Breast Cancer by Microarray Analysis》,链接是:https://www.biorxiv.org/content/10.1101/2020.12.21.423796v1.full 看起来就有点类似;
数据集链接是:https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE88715
这个数据本身使用的是 Agilent-028004 SurePrint G3 Human GE 8x60K Microarray (Probe Name Version) 芯片,发表的文章是 Spatially distinct tumor immune microenvironments stratify triple-negative breast cancers. J Clin Invest 2019 Apr 1;129(4):1785-1800. PMID: 30753167
并不是单细胞转录组,但是呢,它里面的样品也不是传统bulk转录组的样品,而是具体到了Epithelium 和 Stroma 这样的单细胞亚群。
图1:表达芯片的质量控制
一个很简单的箱线图,有意思的是它这个明明是有问题的图表!
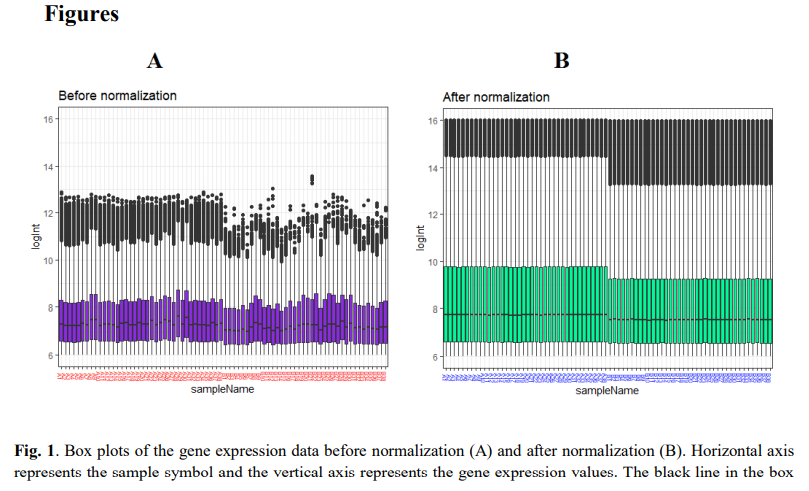
居然也好意思放出来,怪不得一直在预印本没办法发表呢, 两个分组的表达量分布范围天然就有差异,后面的差异分析其实就根本站不住脚!
图2,3,4 差异分析火山图和热图
虽然作者对表达量芯片矩阵的预处理并不到位,是值得批判的,但并不影响作者自顾自的走流程,这也是绝大部分生物信息学入门选手的弱点:
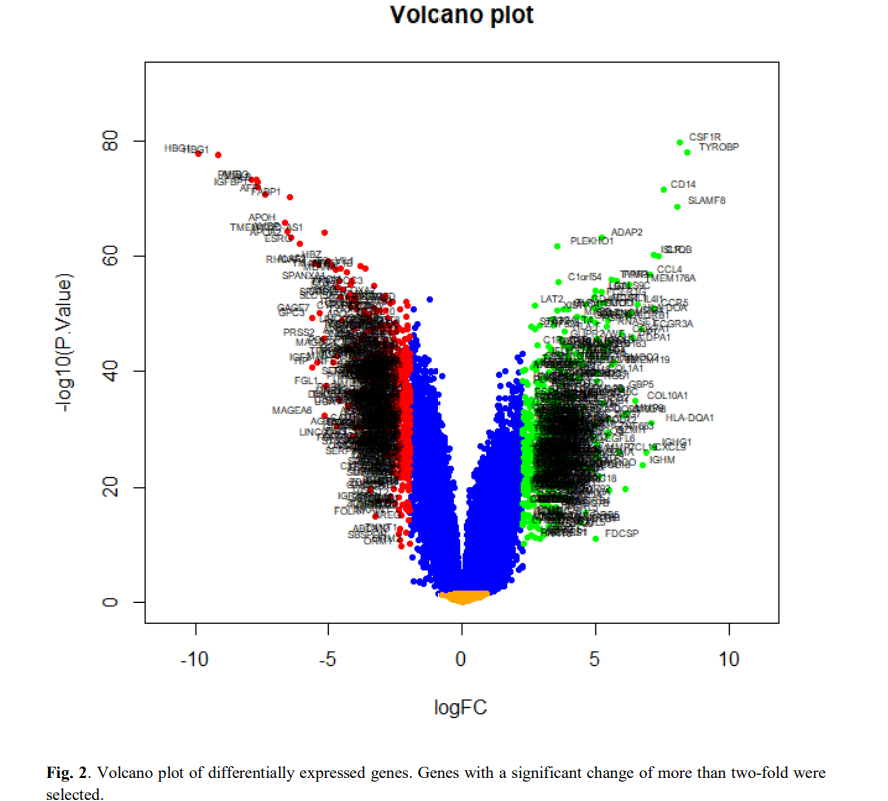
不管三七二十一,差异分析走起,上下调基因判断ok了,就火山图热图画出来了。
差异分析相信大家都不陌生了,基本上看我六年前的表达芯片的公共数据库挖掘系列推文即可;
- 解读GEO数据存放规律及下载,一文就够
- 解读SRA数据库规律一文就够
- 从GEO数据库下载得到表达矩阵 一文就够
- GSEA分析一文就够(单机版+R语言版)
- 根据分组信息做差异分析- 这个一文不够的
- 差异分析得到的结果注释一文就够
图5,6,7,8 挑选到的上下调基因各自的PPI网络图
之所以说它这个文章完全是在凑图,就是因为明明一个图就可以说明的事情它都可以扩充到4个图:
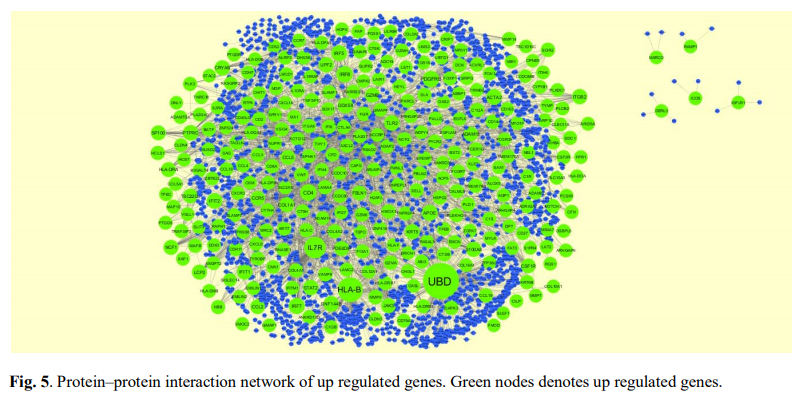
有了基因列表,做PPI网络图无非就是输入string数据库,然后简单的丢入cytoscape软件即可。图9,10 上下调PPI网络图的子网络
有了网络图,找网络图的子网络的cytoscape软件插件也有十多个,我们的b站课程:
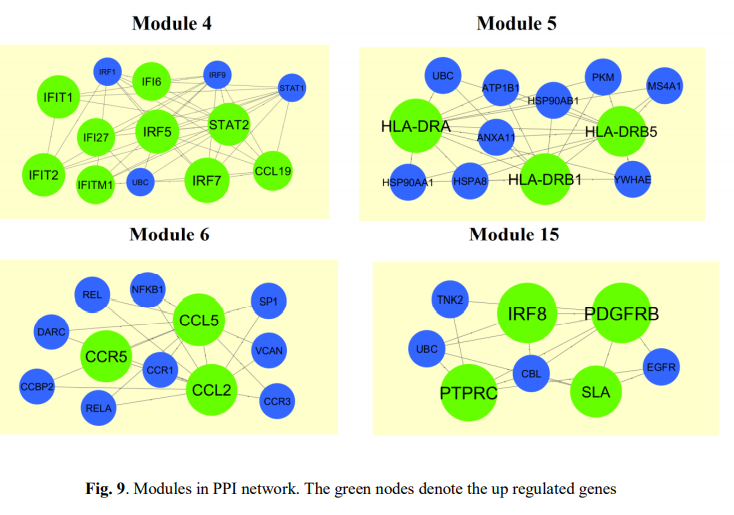
图11,12,13, 14 上下调基因与各自的MiRNA关系图和转录因子关系图
又是凑图,如下所示,一堆花花绿绿的点,除了唬人,就基本上没有其它作用了!

图15,16 上下调核心基因的生存分析
我在生信技能树多次分享过生存分析的细节;
- 人人都可以学会生存分析(学徒数据挖掘)
- 学徒数据挖掘之谁说生存分析一定要按照表达量中位值或者平均值分组呢?
- 基因表达量高低分组的cox和连续变量cox回归计算的HR值差异太大?
- 学徒作业-两个基因突变联合看生存效应
- TCGA数据库里面你的基因生存分析不显著那就TMA吧
- 对“不同数据来源的生存分析比较”的补充说明
- 批量cox生存分析结果也可以火山图可视化
- 既然可以看感兴趣基因的生存情况,当然就可以批量做完全部基因的生存分析
- 多测试几个数据集生存效应应该是可以找到统计学显著的!
- 我不相信kmplot这个网页工具的结果(生存分析免费做)
- 为什么不用TCGA数据库来看感兴趣基因的生存情况
- 200块的代码我的学徒免费送给你,GSVA和生存分析
- 集思广益-生存分析可以随心所欲根据表达量分组吗
- 生存分析时间点问题
- 寻找生存分析的最佳基因表达分组阈值
- apply家族函数和for循环还是有区别的(批量生存分析出图bug)
- TCGA数据库生存分析的网页工具哪家强
- KM生存曲线经logRNA检验后也可以计算HR值
生存分析是目前肿瘤等疾病研究领域的点睛之笔!图17,18 上下调核心基因的肿瘤和对照表达差异
表达差异无非就是箱线图啦,而且绝大部分都是网页工具代劳!
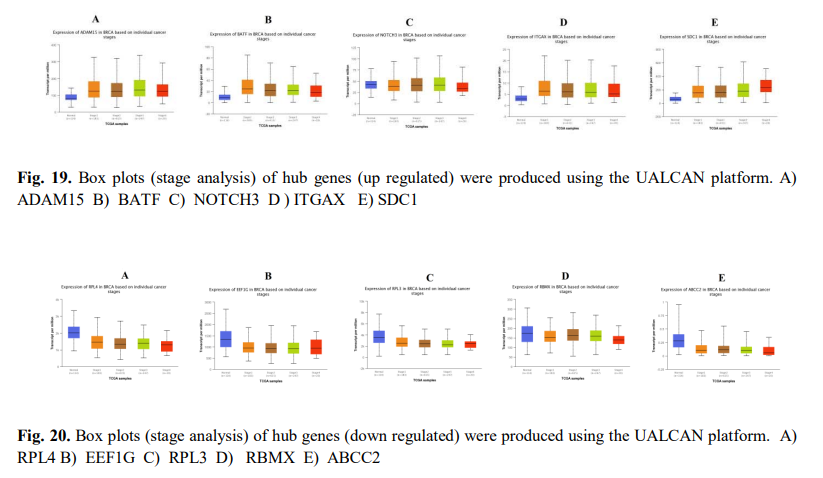
图19,20 上下调核心基因的肿乳腺癌亚型表达差异
仍然是丑爆了的图:
微弱的趋势,而且作者也没有给出统计量!图21 看核心基因的突变全景图 CbioPortal 网页工具
CbioPortal 网页工具可以看到多组学信息,不过这个研究仅仅是看了看突变情况:
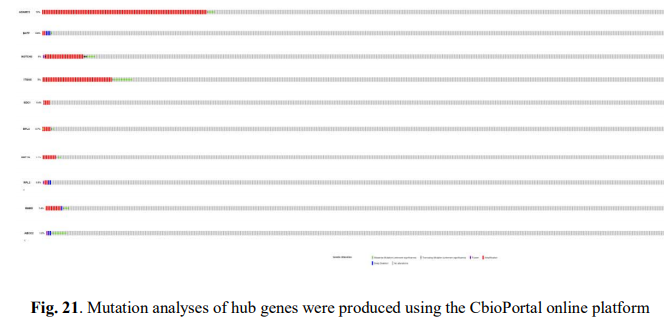
图22 看核心基因的蛋白表达情况 human protein atlas (HPA)网页工具
同样的,使用在线的 human protein atlas (HPA)网页工具即可
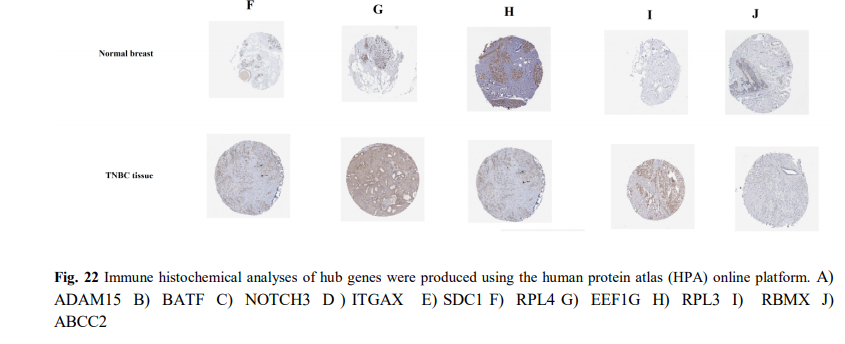
图23 看核心基因的TNBC亚型生存情况
生存分析已经无需过多介绍了,感兴趣可以具体看 我们的b站免费视频课程《临床生存分析》
图24 看核心基因的quantitative PCR实验验证
终于有一点点实验验证了,不容易啊!
图25 看核心基因与免疫浸润关系
仍然是网页工具,略