最近学员群又有人问到了 Agilent-012391 Whole Human Genome Oligo Microarray G4112A 这样的芯片数据,我让学生打包数据成为rdata发给我,我检查了一下,发现里面的基因ID其实是有问题的,如下所示:
> as.numeric(exprSet['GAPDH',])
[1] -0.251630 -0.155540 -0.273550 -0.380700 -0.428190 -0.392950
[7] -0.103190 -0.414220 -0.314840 -0.357170 -0.356710 -0.413600
[13] -0.255290 -0.411110 -0.309840 -0.393780 -0.350690 -0.141290
[19] -0.381040 -0.360000 -0.322420 -0.254410 -0.219950 -0.465110
[25] -0.463820 -0.151940 -0.389230 -0.412180 -0.215320 -0.395210
[31] -0.444730 -0.349110 -0.252500 -0.492400 -0.344810 -0.369310
[37] 0.066505 -0.291790 -0.280300 -0.189730 -0.386790 -0.263650
[43] -0.321960 -0.310220 -0.243800 -0.253200 -0.492870 -0.360890
[49] -0.418790 -0.356350 -0.158260 -0.364990 -0.376200 -0.148720
[55] -0.380690 -0.313450 -0.320720 -0.460920 -0.380320 -0.355930
[61] -0.326810 -0.408100 -0.402210
> as.numeric(exprSet['ACTB',])
[1] 0.021024 0.014020 0.030149 0.072446 0.125180 0.056335
[7] 0.111260 0.109760 0.029394 0.045947 0.053515 -0.020037
[13] -0.005441 -0.039747 0.114760 0.017469 0.041975 -0.003700
[19] 0.056623 -0.018326 0.050075 0.061230 0.119760 0.075487
[25] 0.070703 0.064862 0.114500 0.129870 0.058675 0.052601
[31] 0.143320 0.091340 0.068041 0.031905 0.166280 0.133070
[37] 0.078555 0.087181 0.060440 0.080932 0.125400 0.106220
[43] 0.075474 0.093877 0.048903 0.042316 0.072025 0.088744
[49] 0.054401 0.025626 0.075098 0.157990 0.121050 0.057900
[55] 0.100470 0.096716 0.083148 0.100050 0.116700 0.062778
[61] 0.041097 0.070754 0.065919
这个基本上不太可能啊,GAPDH和ACTB这两个都是比较常见的高表达量基因,不应该是这样的数值 !
简单的boxplot可以看到,这个表达量矩阵呢,其实是被zscore了,如果你不相信,也可以自己使用下面的代码进行查看:
library(GEOquery)
# 下载 (25.1 MB) ,耗时约一分钟,取决于网络速度
eSet <- getGEO( "GSE11223" ,
getGPL = F)
#(1)提取表达矩阵exp
exp <- exprs(eSet[[1]])
exp[1:4,1:4]
dim(exp)
boxplot(exp[,1:4])
被zscore了的表达量矩阵,其实并不会影响表达量高低划分,也不影响相关性计算。所以我们先弄清楚它的基因ID问题:
方法1:找到对应的包
#方法1 BioconductorR包
#http://www.bio-info-trainee.com/1399.html
if(!require(hgug4112a.db))BiocManager::install("hgug4112a.db")
library(hgug4112a.db)
ls("package:hgug4112a.db")
ids <- toTable(hgug4112aSYMBOL)
head(ids)
dim(ids)
看起来非常正确:
> head(ids)
probe_id symbol
1 A_23_P100001 FAM174B
2 A_23_P100011 AP3S2
3 A_23_P100022 SV2B
4 A_23_P100056 RBPMS2
5 A_23_P100074 AVEN
6 A_23_P100092 ZSCAN29
> dim(ids)
[1] 29693 2
因为确实是探针对应基因名字,很棒!
但是呢,麻烦的是这个数据集的作者并没有按照套路出牌,它上传的表达矩阵并不是探针为行名啊!
> head(exp[,1:4])
GSM282928 GSM282929 GSM282930 GSM282932
3 -0.069101 -0.143870 -0.033719 -0.070925
4 -0.163590 -0.261720 -0.230420 -0.307600
5 0.122570 -0.040508 0.049118 -0.061573
6 -0.285550 -0.200940 -0.248170 -0.532750
8 -0.491660 -0.425710 -0.113030 -0.409210
9 -0.168870 -0.138650 0.022177 -0.040322
> tail(exp[,1:4])
GSM282928 GSM282929 GSM282930 GSM282932
44283 -0.075932 -0.274350 0.292630 -0.21546
44284 0.090840 0.142150 0.196100 0.33035
44285 -0.129640 -0.093712 0.104620 -0.13917
44286 -0.047418 0.357040 -0.002053 -0.11540
44287 0.060758 0.051537 0.146950 0.17567
44290 -1.369200 -1.470200 -1.526100 -1.74300
所以这样的转换是不可能成功的啊,它首先表达量矩阵的行名字居然并不是以1开头。
方法2:下载GPL的soft文件 ,需要网络比较好
下载GPL的soft文件啊如果我是在中国大陆,基本上都是放弃这个方法,所以仅仅是给出示例代码:
# https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GPL1708
if(F){
library(GEOquery)
#Download GPL file, put it in the current directory, and load it:
gpl <- getGEO('GPL1708', destdir=".")
# 网络太慢了放弃
colnames(Table(gpl))
head(Table(gpl)[,c(1,15)]) ## you need to check this , which column do you need
probe2gene=Table(gpl)[,c(1,15)]
head(probe2gene)
library(stringr)
save(probe2gene,file='probe2gene.Rdata')
}
#
# load(file='probe2gene.Rdata')
# ids=probe2gene
方法3:直接读取txt文件
在 https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GPL1708 可以看到:
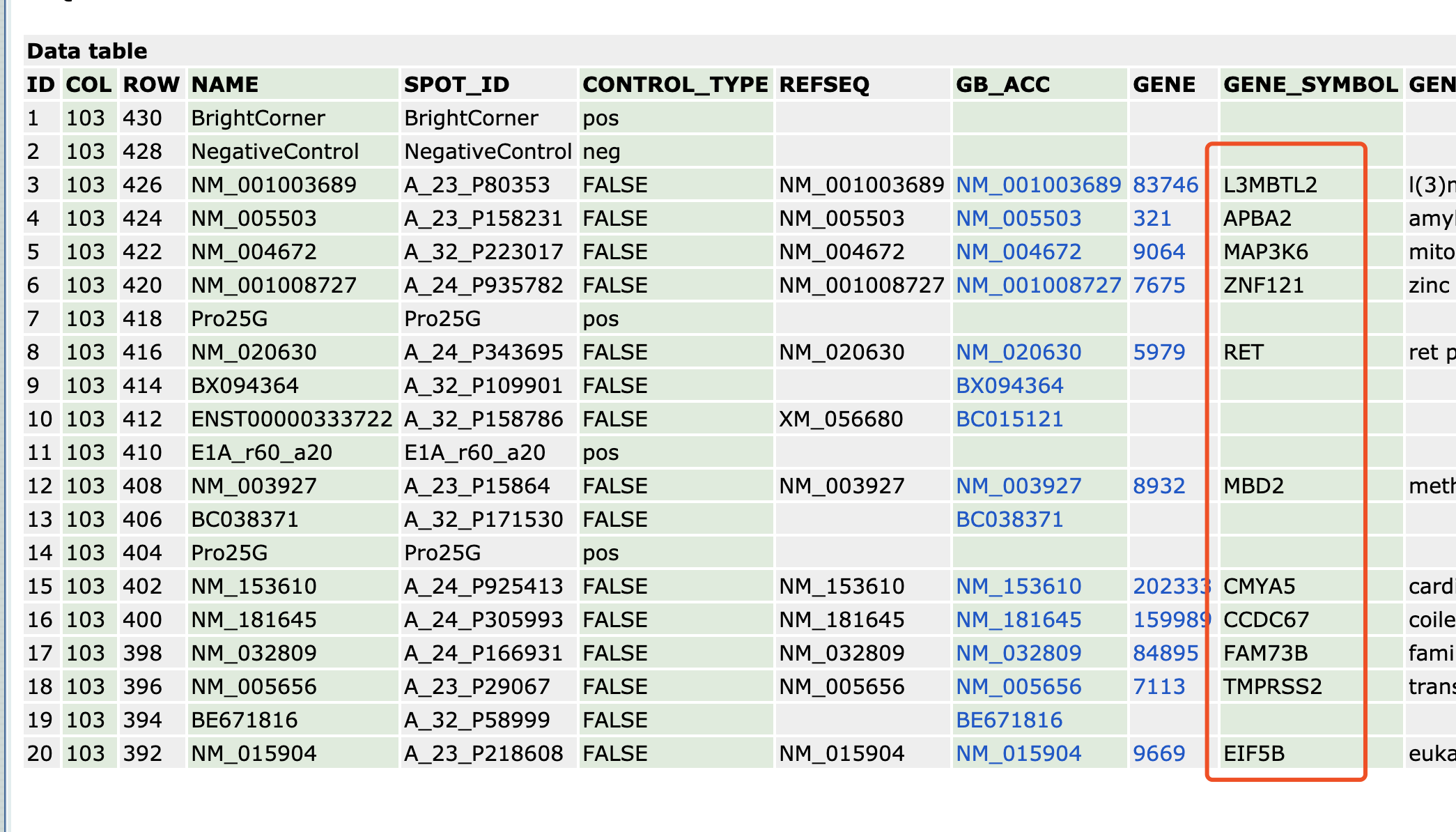
可以看到,第1,2,7行其实并不是传统意义的针对基因表达量设计的探针,这就是为什么前面下载了作者的表达量矩阵,居然是缺失1,2,7行名字的表达量的。
下载这个表格文件存储为txt文件,文件名是:GPL1708-20418.txt ,然后就可以直接读取啦!
> a=fread('GPL1708-20418.txt',data.table = F)
> a[1:4,1:4]
ID COL ROW NAME
1 1 103 430 BrightCorner
2 2 103 428 NegativeControl
3 3 103 426 NM_001003689
4 4 103 424 NM_005503
> colnames(a)
[1] "ID" "COL"
[3] "ROW" "NAME"
[5] "SPOT_ID" "CONTROL_TYPE"
[7] "REFSEQ" "GB_ACC"
[9] "GENE" "GENE_SYMBOL"
[11] "GENE_NAME" "UNIGENE_ID"
[13] "ENSEMBL_ID" "TIGR_ID"
[15] "ACCESSION_STRING" "CHROMOSOMAL_LOCATION"
[17] "CYTOBAND" "DESCRIPTION"
[19] "GO_ID" "SEQUENCE"
这个时候的转换,就容易多了,其实跟这个芯片的探针ID已经没有关系了,因为作者表达量矩阵就并不是以探针为名字,而是GPL1708-20418.txt 文件里面的行号,就是这么的诡异!