我们做肿瘤研究的单细胞数据,一般来说会选择初步很粗狂的定义大的细胞亚群,比如我常用的 第一次分群是通用规则是:
- immune (CD45+,PTPRC),
- epithelial/cancer (EpCAM+,EPCAM),
- stromal (CD10+,MME,fibo or CD31+,PECAM1,endo)
然后绝大部分文章都是抓住免疫细胞亚群进行细分,包括淋巴系(T,B,NK细胞)和髓系(单核,树突,巨噬,粒细胞)的两大类作为第二次细分亚群。
但是也有不少文章是抓住stromal 里面的fibo 和endo进行细分,并且编造生物学故事的。但是,在真实单细胞数据分析里面,你会惊讶的发现,stromal 里面并不是只有fibo 和endo哦,还可以有smooth muscle cells和percite这两个细胞亚群。
比如2018的文章:《Single-Cell Transcriptional Profiling Reveals CellularDiversity and Intercommunication in the Mouse Heart》 ,它的数据在 E-MTAB-6173 ,可以下载后进行深度分析!
文章是三年前发表的,那个时候的单细胞亚群的生物学命名还是比较原始的手段,需要大量阅读文章,比如研究者:We identified 12 distinct cell clusters expressing known markers of major cell types (Figures 1B and 1C).

可以看到,是大量参考文献,拿到了常见的单细胞亚群的标记基因,所以有如下所示的常规细胞亚群:
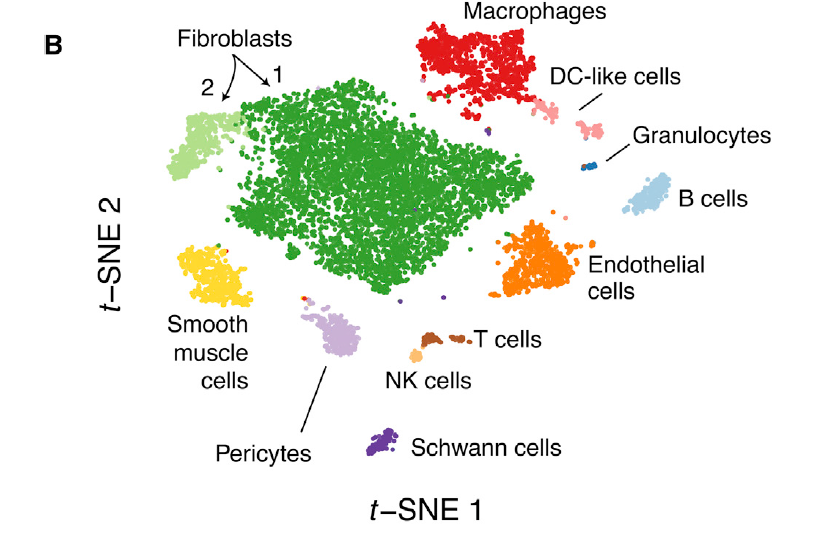
可以看到各个细胞亚群的非常特异的基因,如下所示的展示:
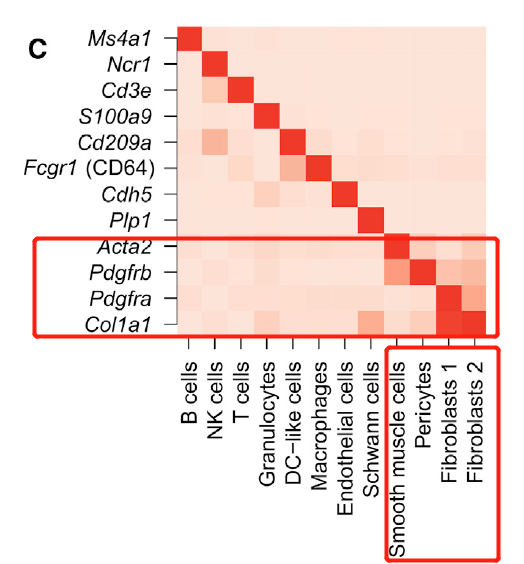
这样的展示方式算是比较常规的啦,属于以前我们做的投票:可视化单细胞亚群的标记基因的5个方法,下面的5个基础函数相信大家都是已经烂熟于心了: - VlnPlot(pbmc, features = c(“MS4A1”, “CD79A”))
- FeaturePlot(pbmc, features = c(“MS4A1”, “CD79A”))
- RidgePlot(pbmc, features = c(“MS4A1”, “CD79A”), ncol = 1)
- DotPlot(pbmc, features = unique(features)) + RotatedAxis()
- DoHeatmap(subset(pbmc, downsample = 100), features = features, size = 3)