很多人都说传统的bulk转录组测序“廉颇老矣”,急急忙忙转向了单细胞转录组这样的热点技术。
诚然,单细胞CNS好文层出不穷,不过最近无意中看到了一个传统的bulk转录组测序的science,还是蛮值得分享的。本次要介绍的文章也不旧,发表于2019年2月,在science杂志,标题是;《Tumor metastasis to lymph nodes requires YAP-dependent metabolic adaptation》,链接是 https://science.sciencemag.org/content/363/6427/644
通篇文章就一个转录组测序数据,如果没有背景知识,拿到了这样的数据仅仅是分析的话基本上没有用,但是结合背景知识辅助大量的湿实验就是一个非常好的生物学故事,卖到了science杂志!
转录组数据介绍
样品分成如下所示的3组:
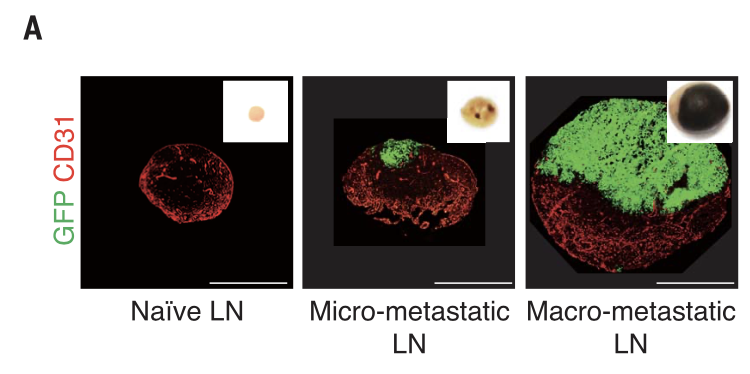
表达量矩阵文件并没有上传到GEO数据库,我只看到了这个链接:https://www.ebi.ac.uk/arrayexpress/experiments/E-MTAB-7621/
虽然没有表达量矩阵文件,但是有原始的测序数据在:https://www.ebi.ac.uk/ena/browser/view/PRJEB30960
ftp.sra.ebi.ac.uk/vol1/run/ERR309/ERR3090776/Macro3_GGCCAA_L001_R1_001.fastq.gz
ftp.sra.ebi.ac.uk/vol1/run/ERR309/ERR3090777/Macro5_AGAATC_L001_R1_001.fastq.gz
ftp.sra.ebi.ac.uk/vol1/run/ERR309/ERR3090778/Macro6_AGGCAT_L001_R1_001.fastq.gz
ftp.sra.ebi.ac.uk/vol1/run/ERR309/ERR3090779/Macro7_ACACGC_L001_R1_001.fastq.gz
ftp.sra.ebi.ac.uk/vol1/run/ERR309/ERR3090780/PT1_CAGGAC_L001_R1_001.fastq.gz
ftp.sra.ebi.ac.uk/vol1/run/ERR309/ERR3090781/PT2_CAGATG_L001_R1_001.fastq.gz
ftp.sra.ebi.ac.uk/vol1/run/ERR309/ERR3090782/PT3_GTAGAA_L001_R1_001.fastq.gz
ftp.sra.ebi.ac.uk/vol1/run/ERR309/ERR3090783/PT4_CGATCT_L001_R1_001.fastq.gz
ftp.sra.ebi.ac.uk/vol1/run/ERR309/ERR3090784/TDLN2_ATTGGT_L001_R1_001.fastq.gz
ftp.sra.ebi.ac.uk/vol1/run/ERR309/ERR3090785/TDLN4_GCAGCC_L001_R1_001.fastq.gz
ftp.sra.ebi.ac.uk/vol1/run/ERR309/ERR3090786/TDLN5_TGCTAT_L001_R1_001.fastq.gz
ftp.sra.ebi.ac.uk/vol1/run/ERR309/ERR3090787/TDLN6_CTCATA_L001_R1_001.fastq.gz
可以看到是12个样品,而且是单端测序数据文件。
完全不走寻常路的转录组测序数据分析
虽然这个数据集附带的文章发表在science杂志,并不能说明他们的转录组测序数据分析就是值得学习的!
我看了看文章附件:
- Mapping of RNA-Seq reads were performed using Bowtie2version 2.1.0.
- The alignment file was used to assemble transcripts, estimate their abundances,and detect differential expression of genes or isoforms using cufflinks.
- The Read Count datawere processed based on Quantile normalization method using the Genowiz ™ version 4.0.5.6(Ocimum Biosolutions, India).
- For bioinformatics analysis, each gene expression data wasannotated with official gene symbols, then normalized (log2). The principal component analysis(PCA) data were analyzed using Genowiz™ version 4.0.5.6 (Ocimum Biosolutions, India).
- Unsupervised hierarchically clustered heatmaps were generated by using GeneCluster 3.0(University of Tokyo, Human Genome Center).
- Gene set enrichment analysis (31) (GSEA) wasperformed with hallmark (h) or oncogenic (c6) gene set collections of the Molecular SignatureDatabase v6.1 (http://www.broadinstitute.org/gsea/msigdb).
- Gene classification analysis andupstream regulator analysis was performed with Ingenuity Pathway Analysis (IPA, Qiagen).
确实是完全不走寻常路啊!其实常规的差异分析呢,基本上看我六年前的表达芯片的公共数据库挖掘系列推文即可; - 解读GEO数据存放规律及下载,一文就够
- 解读SRA数据库规律一文就够
- 从GEO数据库下载得到表达矩阵 一文就够
- GSEA分析一文就够(单机版+R语言版)
- 根据分组信息做差异分析- 这个一文不够的
- 差异分析得到的结果注释一文就够
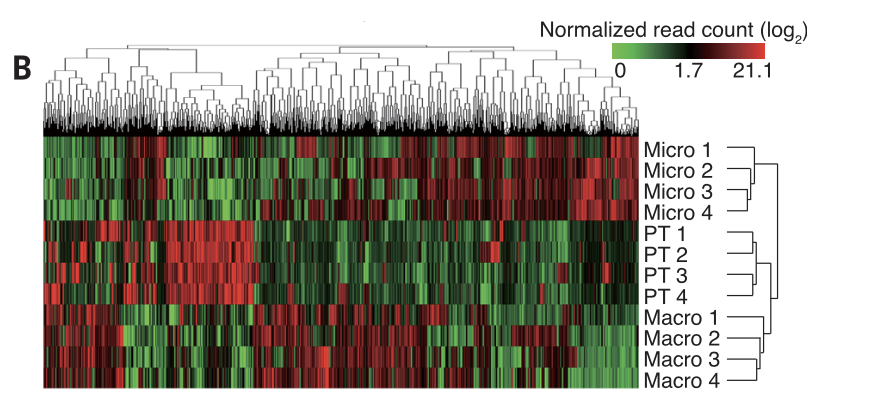
我强调的转录组质控3张图也是存在的
首先是表达量热图:
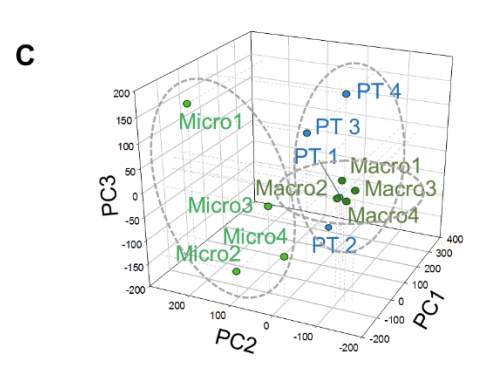
文章的主成分分析图选择了3维:
这些图表绘制我都有公开课: - 视频免费在B站:https://www.bilibili.com/video/BV12s41137HY 大家学习的时候记得发弹幕交流哈。
- 也有微云离线版本视频下载本地播放:
-
- 上游分析视频以及代码资料在:https://share.weiyun.com/5QwKGxi
- 下游主要是基于counts矩阵的标准分析的代码 https://share.weiyun.com/50hfuLi
- 同步查看视频配套代码 :https://www.jianshu.com/p/a84cd44bac67
- RNA-SEQ实战演练的素材:https://share.weiyun.com/5h1Z2QY ,包括一些公司PPT,综述以及文献以及测试数据
- RNA-SEQ 实战演练的思维导图:文档链接:https://mubu.com/doc/38y7pmgzLg 密码:p6fo
标准的差异分析和富集分析
首先是GSEA算法针对msigdb的结果:
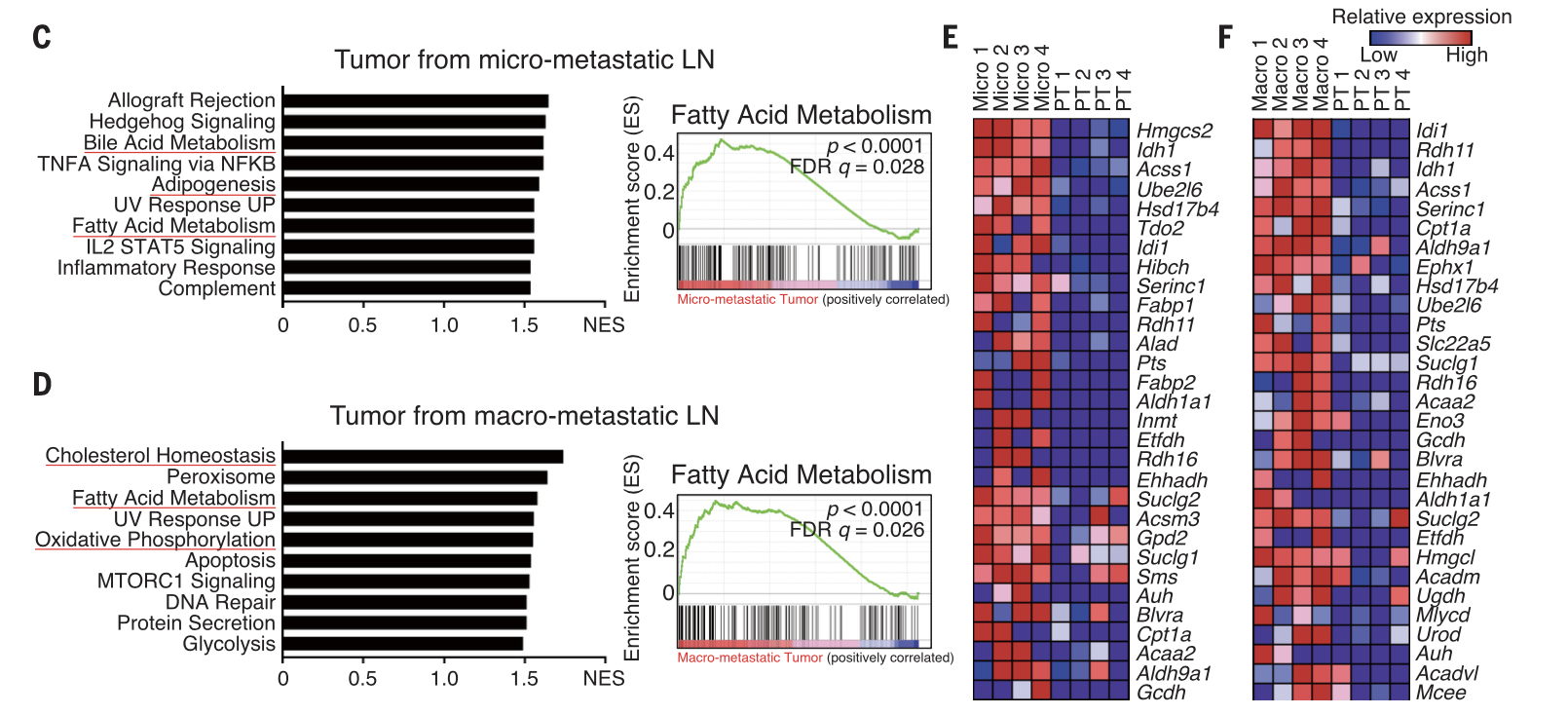
可以看到无论是micro- metastatic (Micro), 还是 macrometastatic (Macro) ,相比起 primary (PT), ,都有一个共同的 fatty acid oxidation (FAO). 通路在转移癌症里面是被激活的。
然后因为是大课题组,所以使用了土豪高配版 Ingenuity Pathway Analysis (IPA, Qiagen). 也是做生物学数据库注释:
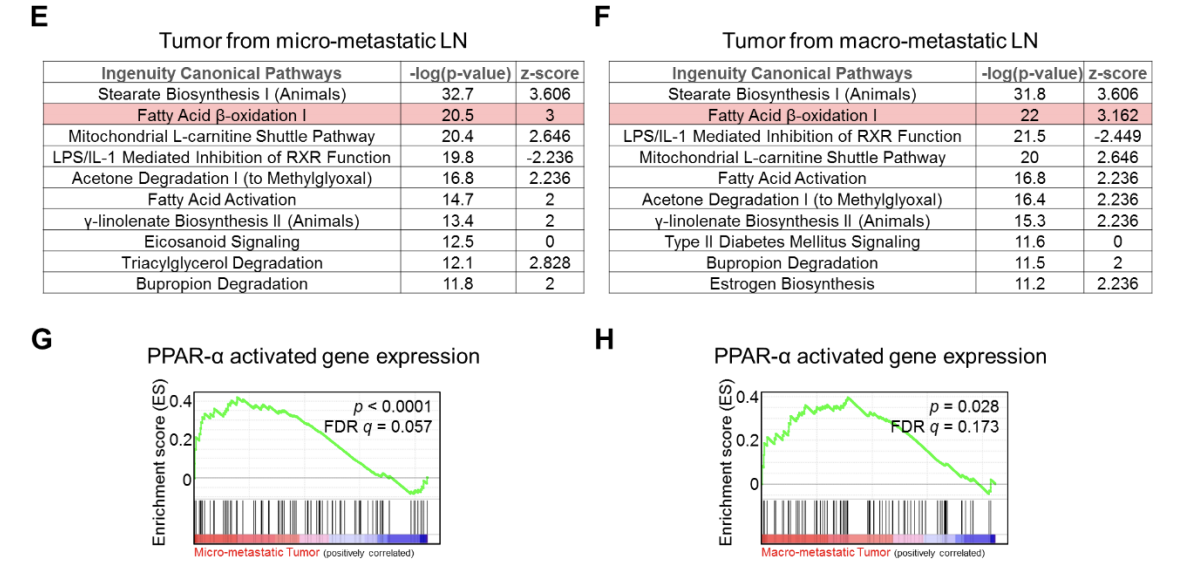
可以看到即使你没有经费购买土豪高配版 Ingenuity Pathway Analysis (IPA, Qiagen).,普通的GSEA算法针对msigdb也是可以拿到合理的结果,聚焦到 fatty acid oxidation (FAO).前面的分析定位到了FAO
然后就专门设计实验来证明 Fig. 2. Enhanced FAO is required for LN metastasis. 然后就需要弄清楚是什么激活了FAO,这个时候根据作者的背景知识选择性检查了 oncogenic signaling genes
Transcriptional coactivator yes-associated protein (YAP) is selectively activated in LN-metastatic tumors, leading to the up-regulation of genes in the FAO signaling pathway.
实验证明最显著的是;metastasis-adapted B16F10 cells transfected with small interfering RNAs (siRNAs) targeting the indicated genes (n = 4 samples for each group). siCtrl, control siRNA; siMYC, siEGFR, siKRAS, and siAKT, siRNAs for MYC, epidermal growth factor receptor, KRAS, and AKT, respectively.
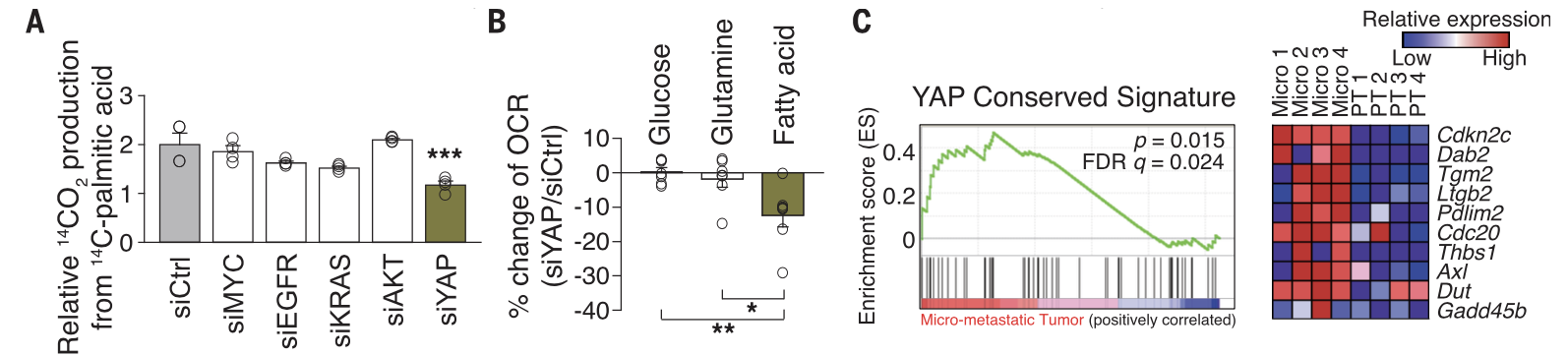
文章最后针对 FAO 的具体机制又看了看两个通路 ,所以有了; - Fig. 3. YAP activation is critical for enhanced FAO and tumor LN metastasis.
- Fig. 4. LN-metastatic tumors produce bile acids that can activate YAP, and YAP activation is correlated with melanoma LN metastasis and patient survival.
如果你拿GSEA算法针对msigdb的,其实哪怕是仅仅是挑选统计学显著的,也是几百个以上的通路被富集到,如果没有足够的背景知识去人为选择结果进行后续生物学故事的编纂,那只能说是简单的数据分析教程,永远不可能成为一个生命科学领域的science文章!如果你有浑厚的生物学背景知识和讲故事的能力
这样的一个传统bulk转录组测序,就12个样品,不到一万块钱,见:明码标价之转录组常规测序服务(仅需799每个样品),数据分析呢,两个差异分析加上GSE富集分析,也就是 800块钱,明码标价之转录组下游分析仅需800元 。
更多分析需求,见我们的明码标价专栏: - ATAC-seq项目的标准分析仅收费1600
- 单细胞转录组的质控降维聚类分群和生物学注释仅收费800
- 普通转录组上游分析仅收费800
- 公共数据库的WGCNA分析仅需800
- 公共数据库的生存分析进需800
测序1万块钱,分析800块钱,你也可以发science哦!