我去年多次分享过 人类和小鼠的基因名字其实是有规律的,见:人鼠基因转换之首字母大写,但其实我也指出来了,这个规律并不是绝对的。
应该是生命科学领域的研究绝大部分情况下都不是绝对的,很多情况下我们可以偷个懒进行普通的人鼠基因转换之首字母大写转换即可,比如单细胞水平下面的标记基因可视化。但是有一些时候,我们也需要认真,比如进行基于小鼠的基因集数据库资源生物学注释。
超几何分布检验和GSEA的差异
通常拿到了上下调差异基因列表,然后说的GO/KEGG数据库注释,指的是超几何分布检验。
但是如果我们并不是首先自定义阈值,确定上下调差异基因列表,而是根据某个指标(比如logFC)把全部的基因排序,再去进行GO/KEGG数据库注释,一般来说就是GSEA分析啦。
但是数据库不仅仅是GO/KEGG哦
MSigDB(Molecular Signatures Database)数据库中定义了已知的基因集合:http://software.broadinstitute.org/gsea/msigdb 包括H和C1-C7八个系列(Collection),每个系列分别是:
- H: hallmark gene sets (癌症)特征基因集合,共50组,最常用;
- C1: positional gene sets 位置基因集合,根据染色体位置,共326个,用的很少;
- C2: curated gene sets:(专家)校验基因集合,基于通路、文献等:
- C3: motif gene sets:模式基因集合,主要包括microRNA和转录因子靶基因两部分
- C4: computational gene sets:计算基因集合,通过挖掘癌症相关芯片数据定义的基因集合;
- C5: GO gene sets:Gene Ontology 基因本体论,包括BP(生物学过程biological process,细胞原件cellular component和分子功能molecular function三部分)
- C6: oncogenic signatures:癌症特征基因集合,大部分来源于NCBI GEO 发表芯片数据
- C7: immunologic signatures: 免疫相关基因集合。
可以看到,GO/KEGG是最出名的,但不是唯一的!
如果是小鼠或者其它物种研究呢
这时候进行普通的人鼠基因转换之首字母大写转换可能是有问题,很久以前看到了一个学术单位做过类似的整理,在 http://bioinf.wehi.edu.au/software/MSigDB/
Gene Sets for Human:
H hallmark gene sets (rdata file)
C1 positional gene sets (rdata file)
C2 curated gene sets (rdata file)
C3 motif gene sets (rdata file)
C4 computational gene sets (rdata file)
C5 GO gene sets (rdata file)
C6 oncogenic signatures (rdata file)
C7 immunologic signatures (rdata file)
Gene Sets for Mouse:
H hallmark gene sets (rdata file)
C2 curated gene sets (rdata file)
C3 motif gene sets (rdata file)
C4 computational gene sets (rdata file)
C5 GO gene sets (rdata file)
C6 oncogenic signatures (rdata file)
C7 immunologic signatures (rdata file)
他们提供了 MSigDB(Molecular Signatures Database)数据库 的全部基因集的下载。
- http://bioinf.wehi.edu.au/software/MSigDB/human_H_v5p2.rdata
- http://bioinf.wehi.edu.au/software/MSigDB/mouse_H_v5p2.rdata
为了说明这个情况,我拿hallmark gene sets 对人类和小鼠,进行了简单的比较:
rm(list = ls())
# http://bioinf.wehi.edu.au/software/MSigDB/human_H_v5p2.rdata
# http://bioinf.wehi.edu.au/software/MSigDB/mouse_H_v5p2.rdata
load("human_H_v5p2.rdata")
load("mouse_H_v5p2.rdata")
ls(Hs.H)
ls(Mm.H)
library(clusterProfiler)
library(org.Hs.eg.db)
library(org.Mm.eg.db)
d1 <- lapply(Hs.H, function(x){
bitr(x,
fromType = "ENTREZID",
toType = "SYMBOL",
OrgDb = org.Hs.eg.db)[,2]
})
d2 <- lapply(Mm.H, function(x){
bitr(x,
fromType = "ENTREZID",
toType = "SYMBOL",
OrgDb = org.Mm.eg.db)[,2]
})
identical(names(d1),names(d2))
names(d2)
首先看看人类特殊的基因
代码如下:
shu=lapply(names(d1), function(x){
h=d1[[x]]
m=toupper(d2[[x]])
hu=setdiff(h,m)
hu
})
shu
得到的基因如下:
[[1]]
[1] "BCL2A1" "CXCL6" "ICOSLG"
[[2]]
[1] "GPI" "ERO1A" "ZNF292" "CA12" "MT1E" "MT2A"
[[3]]
[1] "CYP51A1" "SCD" "TP53INP1" "STX5" "NIBAN1"
[[4]]
[1] "KIF4A" "SEPTIN9"
[[5]]
[1] "TP53"
这个时候有两个可能性,首先,可能是在小鼠里面,这个 hallmark gene sets 就并不包含这个基因,另外一个可能是,简单的基因大小写转换并不能对应这个基因,比如我们看看第五个基因集里面的人类特有的TP53。
仔细看了看在小鼠里面,这个基因是 “Trp53” , 这就很尴尬了,如果是简单的人鼠基因转换之首字母大写转换,就贻笑大方了。
再看看小鼠特殊的基因
代码如下:
smu=lapply(names(d1), function(x){
h=d1[[x]]
m=toupper(d2[[x]])
mu=setdiff(m,h)
mu
})
smu
得到的基因如下:
[[50]]
[1] "AKT1" "AKT2" "FAP" "INS1" "INS2" "ABCC1" "PAK1" "STXBP2" "ABCC9" "HNF1B" "PAK2" "ABCC4"
[13] "ABCC12" "ABCC5" "SEC11C"
我们深入看看 第50个基因集:
> d1[[50]]
[1] "PAX6" "NEUROD1" "ISL1" "NKX2-2" "PCSK1" "NKX6-1"
[7] "SLC2A2" "SEC11A" "DCX" "SPCS1" "FOXA2" "GCK"
[13] "MAFB" "INS" "PDX1" "ABCC8" "IAPP" "SRP9"
[19] "NEUROG3" "FOXO1" "AKT3" "GCG" "DPP4" "PAX4"
[25] "SYT13" "SCGN" "HNF1A" "STXBP1" "CHGA" "VDR"
[31] "PCSK2" "INSM1" "SST" "ELP4" "SRPRB" "PAK3"
[37] "G6PC2" "PKLR" "LMO2" "SRP14"
> d2[[50]]
[1] "Gck" "Akt1" "Akt2" "Neurog3" "Chga" "Dcx"
[7] "Dpp4" "Fap" "G6pc2" "Gcg" "Foxa2" "Iapp"
[13] "Ins1" "Ins2" "Isl1" "Mafb" "Lmo2" "Abcc1"
[19] "Neurod1" "Nkx2-2" "Nkx6-1" "Pak1" "Pak3" "Pax4"
[25] "Pax6" "Pcsk1" "Pcsk2" "Pdx1" "Pklr" "Slc2a2"
[31] "Sst" "Srp14" "Srprb" "Stxbp1" "Stxbp2" "Abcc8"
[37] "Abcc9" "Hnf1a" "Hnf1b" "Scgn" "Vdr" "Pak2"
[43] "Akt3" "Abcc4" "Abcc12" "Srp9" "Abcc5" "Insm1"
[49] "Foxo1" "Sec11a" "Sec11c" "Spcs1" "Elp4" "Syt13"
全部的50个基因集差异情况
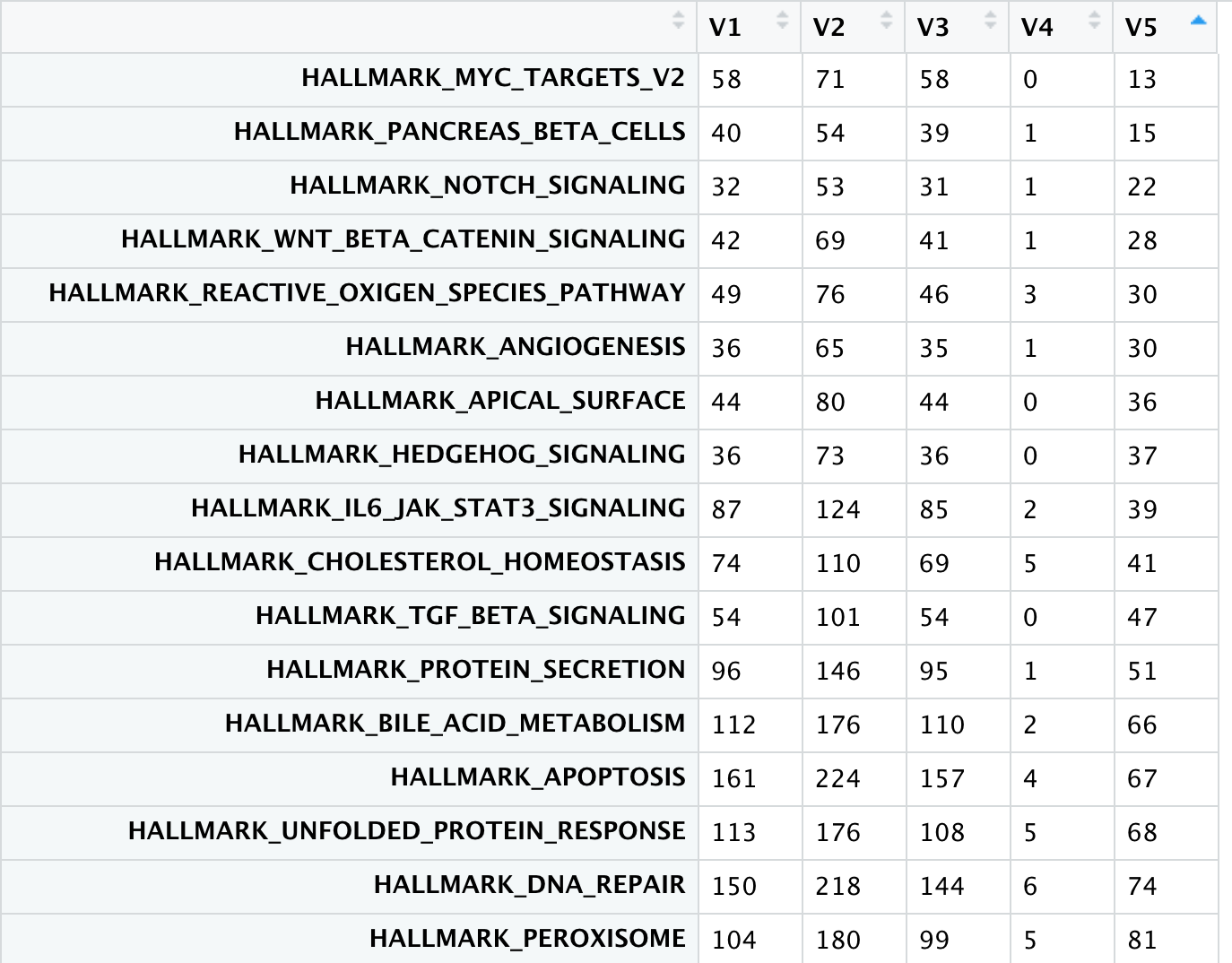
下面的表格里面的表头分别是:
- 人基因集的基因数量
- 小鼠基因集的基因数量
- 两个基因集overlap数量
- 人特有的基因数量
- 小鼠特有的基因数量:
这个代码我就不发出来了,作为一个学徒作业吧!
挺有意思的,生命科学就是这样的任性。