最近有粉丝在我们《生信技能树》公众号后台付费求助,想follow一个文章看两个基因组合起来在一个数据集的生存分析。
因为她的课题是保密的,我这里不方便提基因名字和数据集,就show她想follow的例子吧,就是文章 https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5021195/ 中的 Extended Figure 2 的图 e。因为对方让我们先秀一秀肌肉,所以我把这个 Extended Figure 2 的图 e安排给了学徒,感谢学徒在这个春节假期还兢兢业业完成任务!
下面是学徒的探索
我们先来看看关于Extended Figure 2 的图 e 的 原文描述:

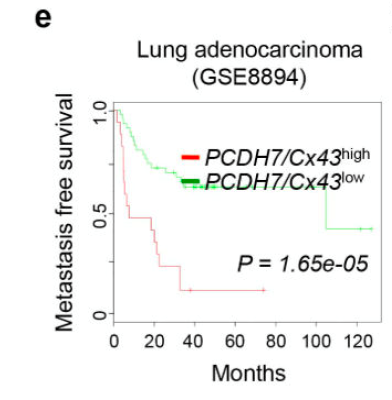
e, Kaplan-Meier plot illustrates the probability of cumulative metastasis free survival in 63 cases (GSE8893) of lung adenocarcinoma based on Cx43/PCDH7 expression in the primary tumour.
其实就是KM 显示了63例肺腺癌(GSE8893)基于原发肿瘤中Cx43/PCDH7表达的累积无转移生存概率
有意思的是注释里写的 GSE8893 ,图中画的是 GSE8894 ?
去GEO里查看了数据,发现实际上是 GSE8894,和文中的 63 例 lung adenocarcinoma 对应

首先下载GSE8894表达量矩阵和临床新
全部代码如下:
rm(list = ls())
options(stringsAsFactors = F)
library(AnnoProbe)
# 这个包AnnoProbe,在GitHub上面,安装起来稍微有一点点麻烦。
library(GEOquery)
gse=geoChina('GSE8894')
eSet=gse[[1]]
probes_expr <- exprs(eSet);dim(probes_expr)
head(probes_expr[,1:4])
#boxplot(probes_expr,las=2)
probes_expr=log2(probes_expr+1)
phenoDat <- pData(eSet)
head(phenoDat[,1:4])
table(phenoDat$`cell type:ch1`)
pd <- phenoDat[phenoDat$`cell type:ch1` =='Adenocarcinoma',]
colnames(pd)
pd = pd[,c(2,40,41)]
colnames(pd) = c('id','time','event')
meta = pd
head(meta)
probes_expr = as.data.frame(probes_expr)
exp = probes_expr[,colnames(probes_expr) %in% pd$id]
GPL=eSet@annotation
if(F){
if(!require(hgu133plus2.db))BiocManager::install("hgu133plus2.db")
library(hgu133plus2.db)
ls("package:hgu133plus2.db")
ids <- toTable(hgu133plus2SYMBOL)
head(ids)
}else if(F){
getGEO(GPL)
ids = data.table::fread(paste0(GPL,".soft"),header = T,skip = "ID",data.table = F)
ids = ids[c("ID","Gene Symbol"),]
colnames(ids) = c("probe_id")
}else if(T){
ids = idmap(GPL,type = "bioc")
}
probes_anno=ids
head(probes_anno)
genes_exp <- filterEM(exp,probes_anno )
因为文章关注点是两个基因,所以我仅仅是保存这两个基因的表达量信息和这些病人的临床信息而已,代码如下:
head(genes_exp)
# 'PCDH7'
# GJA1
genes_exp['PCDH7',]
genes_exp['GJA1',]
mat=genes_exp[c('GJA1','PCDH7'),]
save(mat,meta,file = 'input_for_survial.Rdata')
这里有一个小插曲,起初呢,学徒查询的时候发现,GPL570中竟然没有 Cx43?但是 PCDH7是没有问题,如下所示:
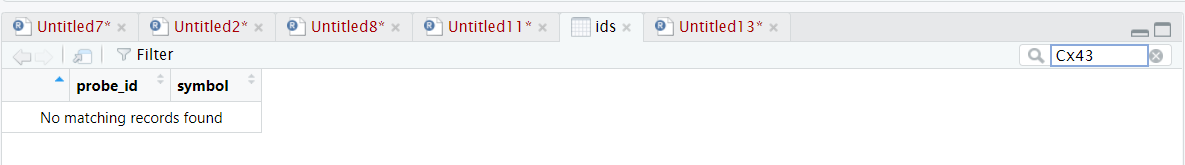
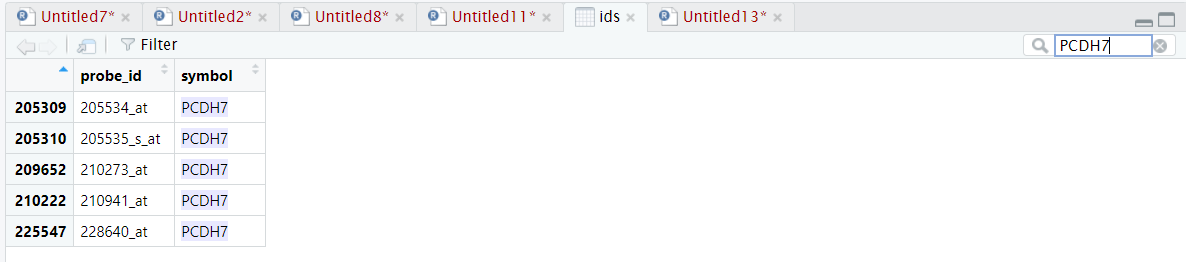
然后她特地查找了一下 Cx43 会不会有其他的别名,发现没有,于是求助了Jimmy老师,发给了她链接
这个基因是:https://www.genecards.org/cgi-bin/carddisp.pl?gene=GJA1
Gap Junction Protein, Alpha 1, 43kDa
Gap junction alpha-1 protein (GJA1), also known as connexin 43 (Cx43), is a protein that in humans is encoded by the GJA1 gene on chromosome 6.
呃,原来她只是在 NCBI 的gene里看了一下,所以查无此基因。不过好歹是问题解决了,继续看下去。
做生存分析
代码如下;
rm(list = ls())
options(stringsAsFactors = F)
library(survival)
library(survminer)
load(file = 'input_for_survial.Rdata')
identical(colnames(mat),rownames(meta))
e=cbind(meta,t(mat))
head(e)
e$time = as.numeric(e$time)
e$event = as.numeric(e$event)
str(e)
colnames(e)
# 找到合适的“高低表达量”的定义,高于cut,就是高表达
tmp.cut <- surv_cutpoint(
e,
time = "time",
event = "event",
variables = c('GJA1','PCDH7')
)
summary(tmp.cut)
plot(tmp.cut, "GJA1", palette = "npg")
# 将数字的表达信息根据粗体转化为high or low的二元信息
tmp.cat <- surv_categorize(tmp.cut)
head(tmp.cat)
table(tmp.cat[,3:4])
####################这里的.
kp=tmp.cat$GJA1 == tmp.cat$PCDH7
table(kp)
tmp.cat=tmp.cat[kp,]
library(survival)
# 常规的画法
?survfit
fit <- survfit(Surv(time, event) ~ GJA1+PCDH7,data = tmp.cat)
ggsurvplot(
fit,
pval = TRUE,
conf.int = FALSE,
xlim = c(0,120),
break.time.by = 20,
legend=c(0.5, 0.9),
legend.title = '',
risk.table.y.text.col = T,
risk.table.y.text = FALSE
)+
labs(x = "Months", y = "Metastasis free survival",
subtitle = "GSE8894") +
ggtitle("lung adenocarcinoma")
也是超级简单,得到了如下所示的跟原文基本上一模一样的图:
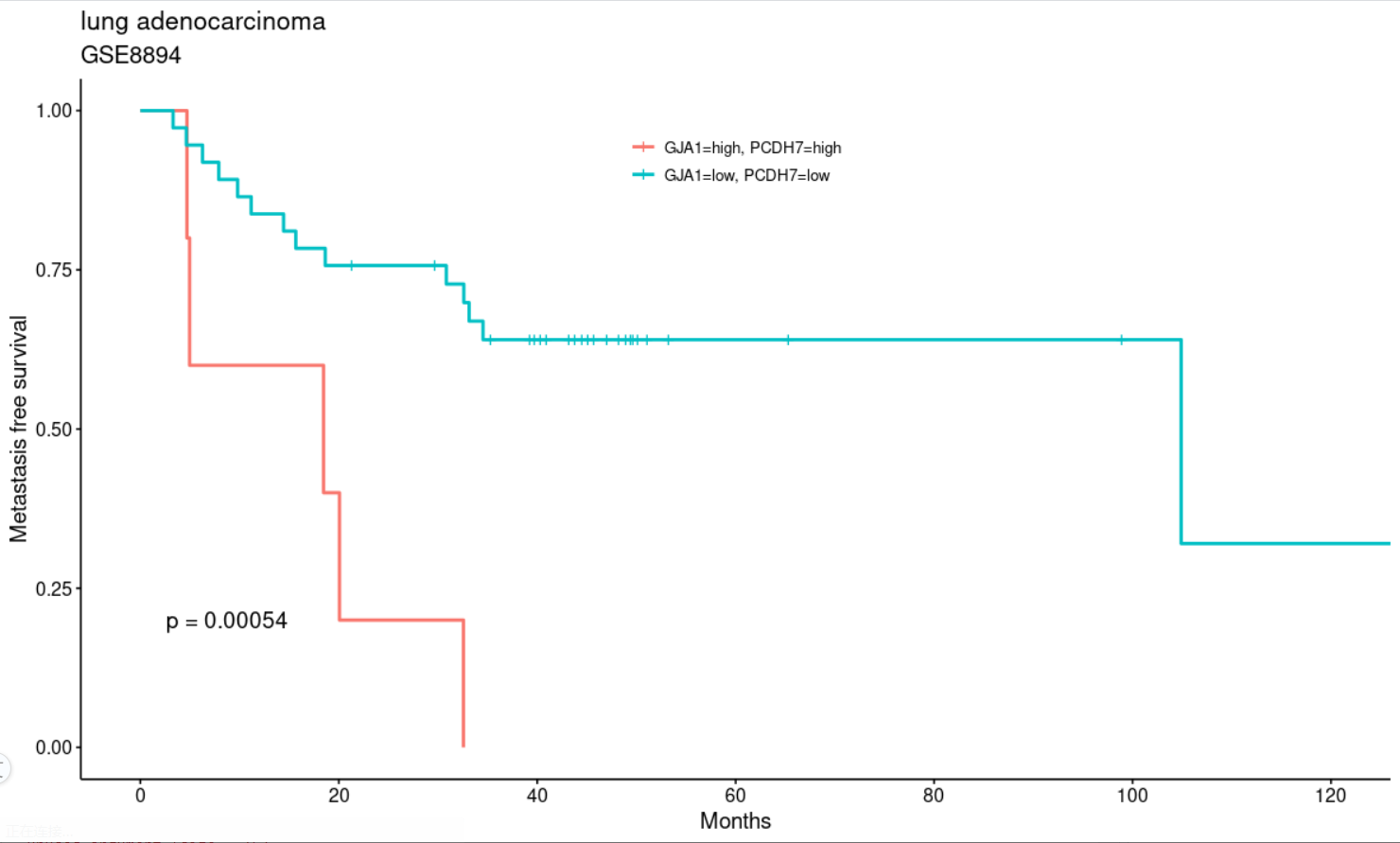
网页工具是不可能做到如此的个性化
如果你有时间请务必学习R语言,自由自在的探索海量的公共数据,辅助你的科研,那么:
如果你没有时间从头开始学编程,也可以委托专业的团队付费拿到同样的数据分析,这样的生存分析费用是800元,可以拿到全部的数据和代码。
- 需要自己读文献筛选合适的数据集
- 提供30分钟左右的生存分析背景知识讲解
如果需要委托,直接在我们《生信技能树》公众号留言即可,我们会安排合适的生信工程师对接具体的项目。