最近有粉丝咨询我多个gsea数据集整合时候的批次效应的处理,我看了看,有affymetrix,agilent,illumina的芯片数据,还有测序的转录组,我勒个去,感觉是在集邮一样,然后邮件附上了一大堆热图,层次聚类图,PCA图,相关性图,看得我眼花缭乱。
为什么一定要纠结批次效应呢,robust rank aggregation (RRA) 算法不香吗?
在 RobustRankAggreg 包就有实现robust rank aggregation (RRA) 算法的函数,而且很早以前我就系统性介绍过:多个数据集整合神器-RobustRankAggreg包,是我的付费推文频次遥遥领先的:
随手查一下 robust rank aggregation (RRA) 的引用情况,就可以看到发表在 Front. Oncol., 27 September 2019的文章《Identification of a Nine-Gene Signature and Establishment of a Prognostic Nomogram Predicting Overall Survival of Pancreatic Cancer》走的就是robust rank aggregation (RRA) 算法整合多个gse数据集的上下调差异基因,文章链接是:https://doi.org/10.3389/fonc.2019.00996
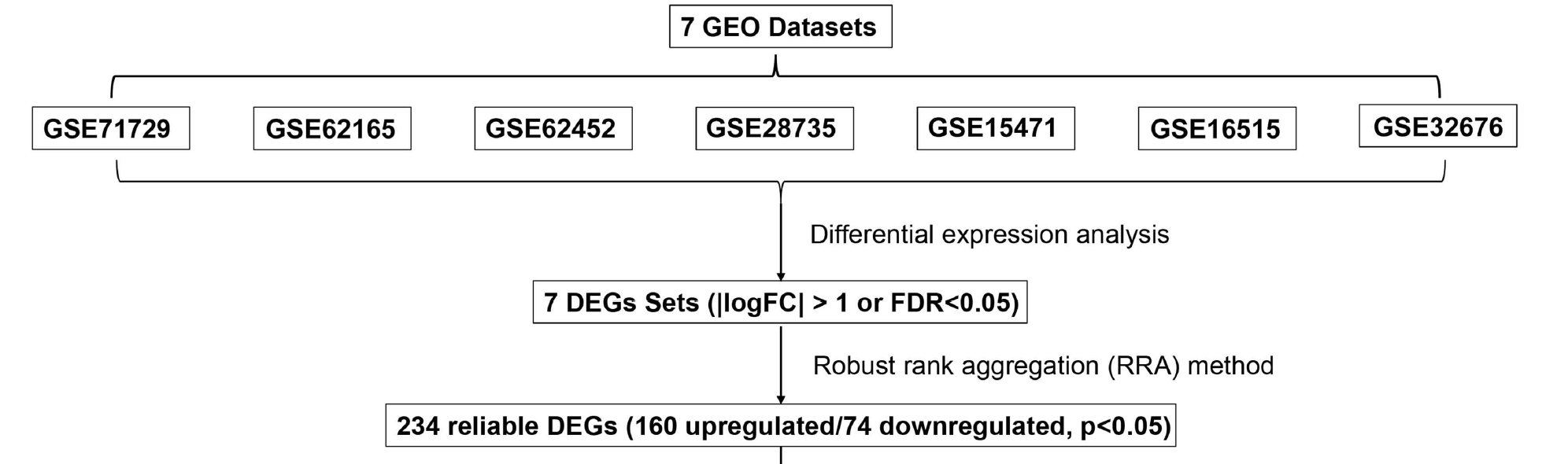
这些数据集(GSE71729, GSE62165, GSE62452, GSE28735, GSE15471, GSE16515, and GSE32676)都是独立的差异分析,然后使用robust rank aggregation (RRA) 算法整合即可,流程示意图如下所示:
这些差异分析呢,可以看到我五年前的教程,推文在:
- 解读GEO数据存放规律及下载,一文就够
- 解读SRA数据库规律一文就够
- 从GEO数据库下载得到表达矩阵 一文就够
- GSEA分析一文就够(单机版+R语言版)
- 根据分组信息做差异分析- 这个一文不够的
- 差异分析得到的结果注释一文就够
反正这些芯片技术都是十几年前的了,大家不要觉得我五年前的教程有什么过时的地方哈。
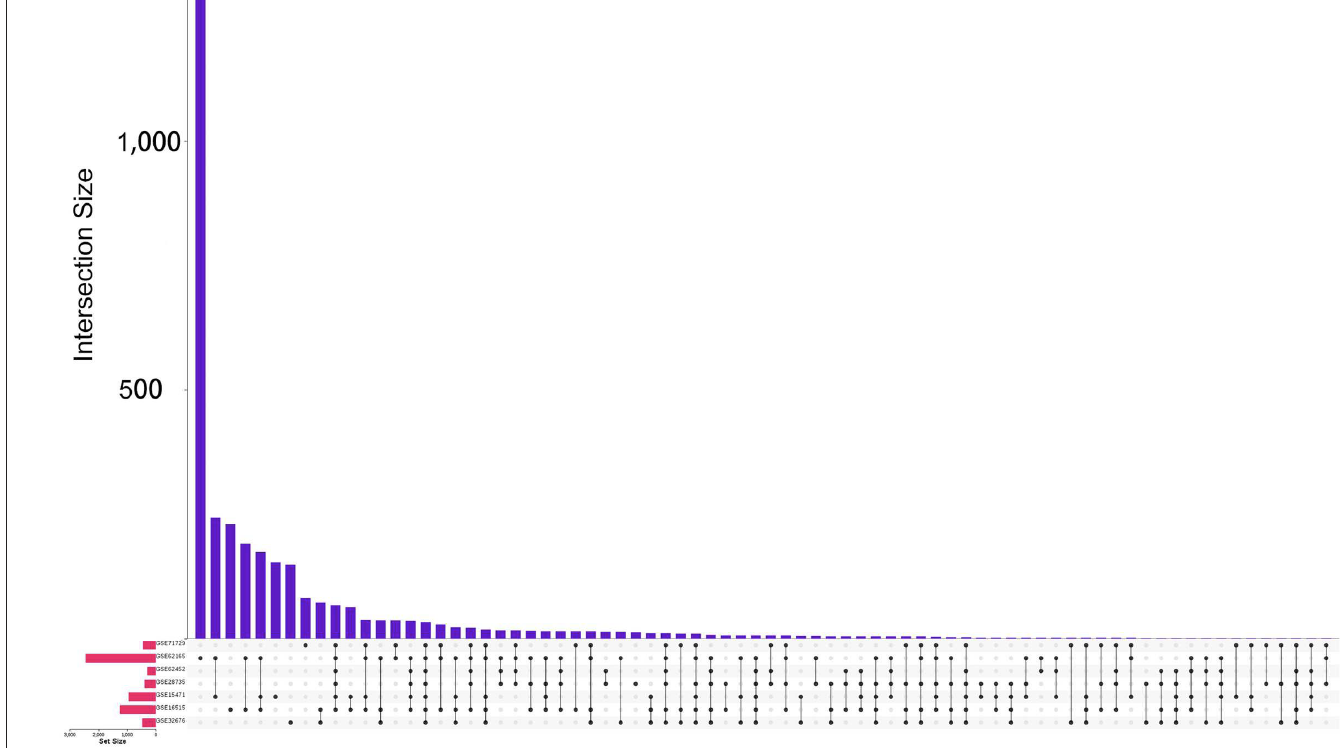
拿到数据集的上下调差异基因后,因为数据集是7个,所以韦恩图不方便展示,这个时候使用upset图,可以看到不同数据集 的 各自独立定位到的上下调基因差别很大:
但是使用robust rank aggregation (RRA) 算法整合,仍然是可以发现那些表现一致的基因,如下所示:
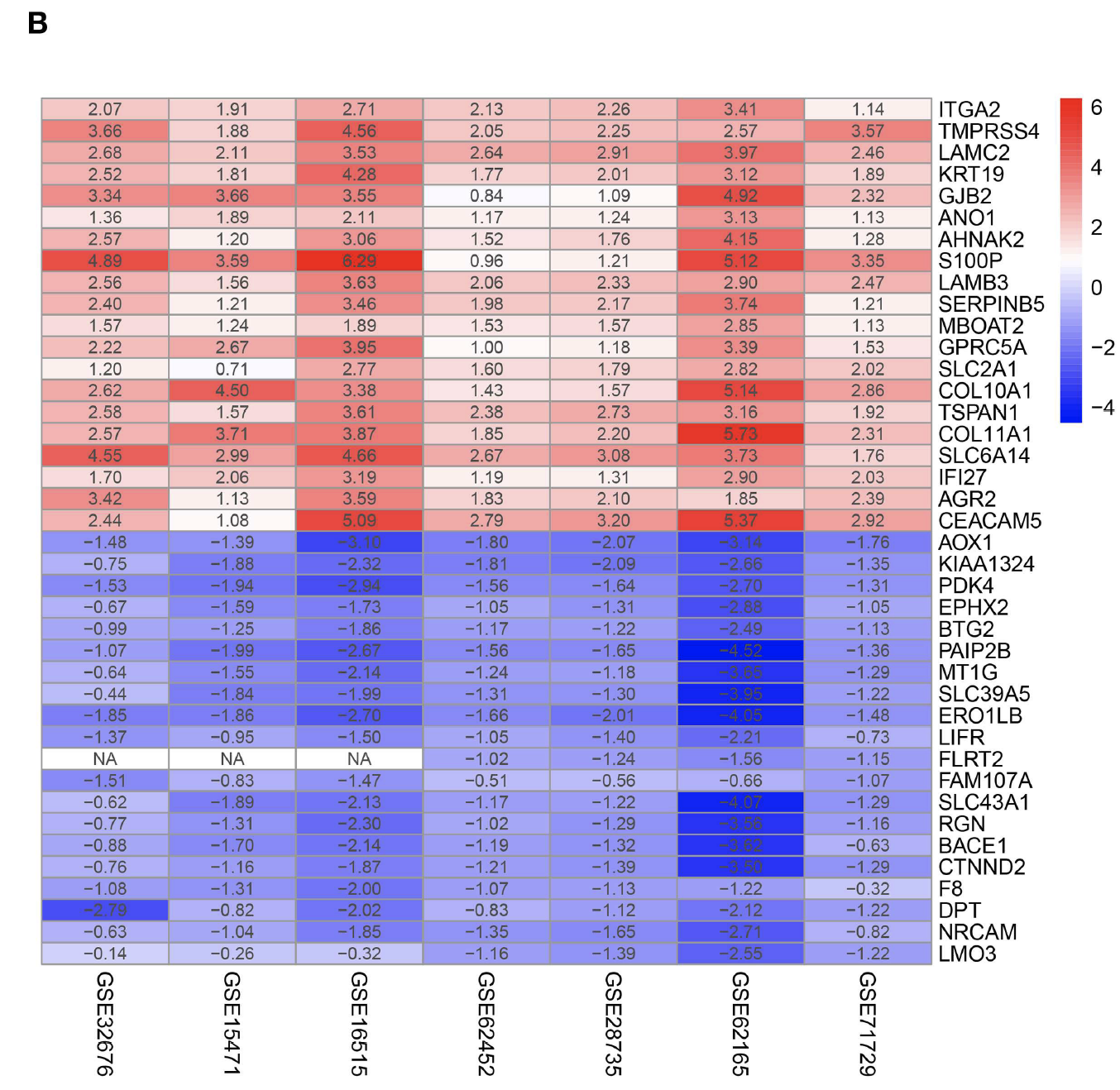
虽然使用robust rank aggregation (RRA) 算法整合拿到的这些上下调基因在不同数据集上下调的程度不一样,但其实各个数据集是无需跨越比较变化倍数的, 因为数据集有自己的独特性,尽管说去除批次效应的算法可以一定程度的抹除掉数据集批次效应。
后面的分析就非常常规了,去有临床信息的病人队列里面检测这些基因的生存预后情况,比如 The Cancer Genome Atlas pancreatic ductal adenocarcinoma (TCGA-PAAD) 队列,就是Univariate and Lasso-Cox regression 啦,基于TCGA构建好预后基因集,再去外部数据集里面验证,这个研究就选择了 GSE62452 and GSE57495 。很明显,必须是统计学选择的哦,这样才能发表嘛。
最后就成为了一个 A nine-gene signature comprising MET, KLK10, COL17A1, CEP55, ANKRD22, ITGB6, ARNTL2, MCOLN3, and SLC25A45 ,虽然说真实的临床价值待商榷,但是该说的场面话可不能少哦: - The nine-gene signature was closely associated with the progression, aggressiveness, and prognosis of pancreatic cancer and its constituents are potential therapeutic targets.
- The prognostic nomogram reliably predicted overall survival in pancreatic cancer and may facilitate individualized treatment and making of medical decisions.
学徒作业
那就复现出上面的差异基因热图吧!自己去下载这些数据集(GSE71729, GSE62165, GSE62452, GSE28735, GSE15471, GSE16515, and GSE32676),走独立的差异分析后,然后使用robust rank aggregation (RRA) 算法整合即可。可以参考我以前的教程:多个数据集整合神器-RobustRankAggreg包。