首先你需要认识node数据库哦,见:https://mp.weixin.qq.com/s/ygOMbwm4tMi2AitguXmcwQ
然后呢,以前jimmy老师分享过 SRA和GEO数据库的规律,见:
和geo以及SRA 数据库一样,node 数据有没有存放规律呢?
通常NODE 中的高通量项目数据分为五类:
参考页面:https://www.biosino.org/node/project/detail/OEP000155
- Project ID,OEP000155,OEP 开头,为项目ID;
- Experiment ID,OEX001583,OEX 开头,为实验ID,对应实验类型,如rna-seq、microarray、wes等;
- Sample ID,OES014891,OES 开头,为样本ID,对应临床样本,是和实验使用的临床样本中记录的名称对应的;
- Run ID,OER028237,OER 开头,为具体的样本在某实验的数据,如测序数据、芯片数据等。
- Data ID,OED065488,OED 开头,为具体数据的下载ID,我们可以通过该ID 直接下载数据,如https://www.biosino.org/download/node/data/public/OED064632
- Analysis ID,OEZ000398,OEZ 开头,为作者提供的一些额外的分析文件,如FPKM 矩阵、拷贝数或变异位点的分析结果,同样也是对应一个或多个OED(如一个FPKM 对应log2 前后的矩阵)。
一篇文章一般对应一个OEP 项目,而一个OEP 项目由多个OEX 实验,而每个OEX 实验中包含了全部参与该实验sample 的OER 信息,而OES 的样本中也可以获取该临床样本所参与的全部OEX 实验中的OER 信息。而一个OER 数据可能包含一个或多个下载数据(如双端测序fq文件,则该OER 下对应R1 和R2 的OED 信息)。
令人难过的是,我发现OED 的排列和前面的其他数据排列并没有任何的规律:
OEP000155 OEX001584 OES014455 OER026801 OED064632
OEP000155 OEX001583 OES014428 OER026252 OED063807
OEP000077 OEX000085 OES002007 OER008964 OED006656
OEP000077 OEX000085 OES002015 OER008965 OED006634
比如上面来自不同项目的数据,是完全没有规律的。
但是node 数据库提供了一个导出所有元数据的接口:
通过该接口,我们可以将所选的类型下数据的元数据下载下来,比如我选择了外显子的OEX 数据:
包含关于OEX 的全部RUN:
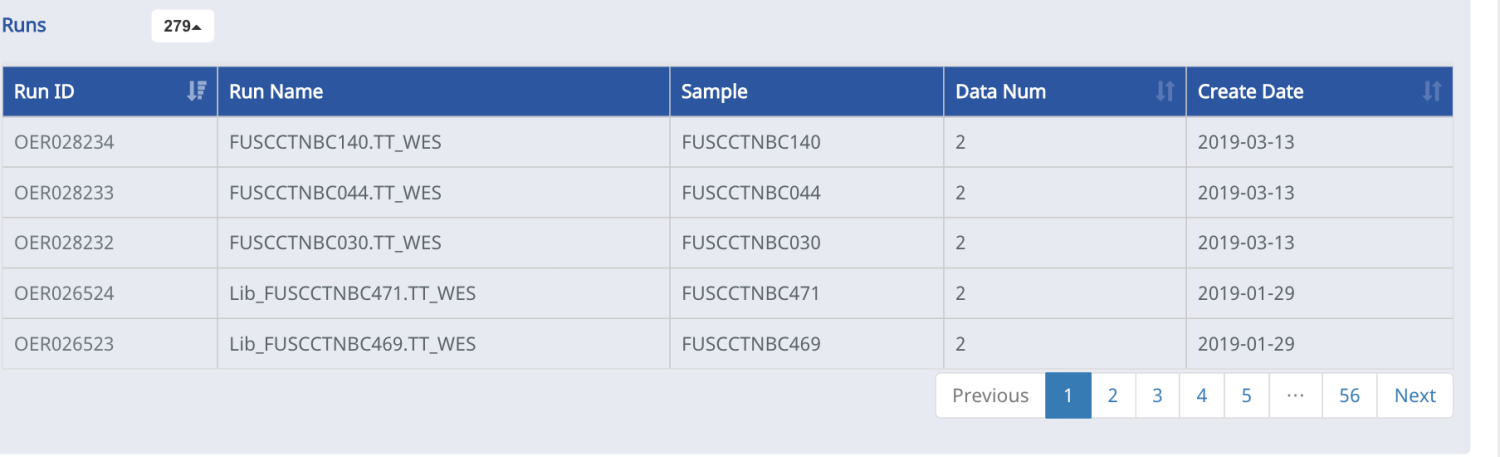
可以通过正上方的按钮获取元数据:

其中包括:
project_id experiment_id sample_id run_id data_id security fileName url MD5
OEP000155 OEX001583 OES014483 OER026249 OED063528 Public FUSCCTNBC001.TT_WES_R1.fastq.gz https://www.biosino.org/download/node/data/public/OED063528 d8681c1944d882f5d86ac7f22867f95c
OEP000155 OEX001583 OES014483 OER026249 OED063804 Public FUSCCTNBC001.TT_WES_R2.fastq.gz https://www.biosino.org/download/node/data/public/OED063804 6d01270fa509f2af6b457253cb35e02f
OEP000155 OEX001583 OES014784 OER026250 OED063529 Public FUSCCTNBC002.TT_WES_R1.fastq.gz https://www.biosino.org/download/node/data/public/OED063529 e020d2b79d165c18ea921cd85f66861e
OEP000155 OEX001583 OES014784 OER026250 OED063805 Public FUSCCTNBC002.TT_WES_R2.fastq.gz https://www.biosino.org/download/node/data/public/OED063805 498830f375f72a414c26fbc7241b15e2
OEP000155 OEX001583 OES014467 OER026251 OED063530 Public FUSCCTNBC005.TT_WES_R1.fastq.gz https://www.biosino.org/download/node/data/public/OED063530 fc91b5c36a62f5e93b39d2a95aab9868
OEP000155 OEX001583 OES014467 OER026251 OED063806 Public FUSCCTNBC005.TT_WES_R2.fastq.gz https://www.biosino.org/download/node/data/public/OED063806 bf37bc65c624877b0c8df9a348ba6ea7
OEP000155 OEX001583 OES014428 OER026252 OED063531 Public FUSCCTNBC006.TT_WES_R1.fastq.gz https://www.biosino.org/download/node/data/public/OED063531 42a6d73596f908f68a87532660bf3535
OEP000155 OEX001583 OES014428 OER026252 OED063807 Public FUSCCTNBC006.TT_WES_R2.fastq.gz https://www.biosino.org/download/node/data/public/OED063807 99ae023111d64b50ffafb3e27cfaa917
OEP000155 OEX001583 OES014628 OER026253 OED063532 Public FUSCCTNBC007.TT_WES_R1.fastq.gz https://www.biosino.org/download/node/data/public/OED063532 d3dbd153b524eebae6aa04c90145b017
这里我截取了一部分的内容。
这时候如果你会linux 的话,就可以尝试直接获取一下上面的下载链接来使用wget 的方式批量下载了,先提取一下下载链接:
$ cat download.txt | cut -f8 | sed '1d' | head -3
https://www.biosino.org/download/node/data/public/OED063528
https://www.biosino.org/download/node/data/public/OED063804
https://www.biosino.org/download/node/data/public/OED063529
这里我只选择了三个,用wget:
nohup cat dl_link | xargs wget &
速度还是很快的,i了i了:
OED063528 2%[ ] 146.23M 16.9MB/s eta 4m 29s
-rw-rw-r-- 1 yzpeng yzpeng 5.1G 1月 18 11:22 OED063528
-rw-rw-r-- 1 yzpeng yzpeng 5.3G 1月 18 11:32 OED063529
-rw-rw-r-- 1 yzpeng yzpeng 5.5G 1月 18 11:27 OED063804
因为OED 只是一串ID,接着我们还需要获取OED 和对应fileName 的文件,将下载的OED 名称修改为对应的fq 文件名:
$ cat download.txt | cut -f5,7 | sed '1d' | head -3
OED063528 FUSCCTNBC001.TT_WES_R1.fastq.gz
OED063804 FUSCCTNBC001.TT_WES_R2.fastq.gz
OED063529 FUSCCTNBC002.TT_WES_R1.fastq.gz
批量修改一下:
cat download.txt | cut -f5,7 | sed '1d' | head -3 > oed2fq_name
我linux 还是能力有限,尝试R 修改一下:
> var = read.table(file = 'oed2fq_name', header = F)
> var
V1 V2
1 OED063528 FUSCCTNBC001.TT_WES_R1.fastq.gz
2 OED063804 FUSCCTNBC001.TT_WES_R2.fastq.gz
3 OED063529 FUSCCTNBC002.TT_WES_R1.fastq.gz
> for (i in 1:nrow(var)){ file.rename(var[i,1], var[i,2]) }
$ ls *.fastq.gz
FUSCCTNBC001.TT_WES_R1.fastq.gz FUSCCTNBC002.TT_WES_R1.fastq.gz
FUSCCTNBC001.TT_WES_R2.fastq.gz
我们还可以获取一下元数据中的md5 信息,进行下载后的文件完整性验证:
$ cat download.txt | awk '{print $9,$7}' | tr '\t' ' ' | sed 's/[ ]/ /' | sed '1d'
d8681c1944d882f5d86ac7f22867f95c FUSCCTNBC001.TT_WES_R1.fastq.gz
6d01270fa509f2af6b457253cb35e02f FUSCCTNBC001.TT_WES_R2.fastq.gz
e020d2b79d165c18ea921cd85f66861e FUSCCTNBC002.TT_WES_R1.fastq.gz
验证一下:
$ cat download.txt | awk '{print $9,$7}' | tr '\t' ' ' | sed 's/[ ]/ /' | sed '1d' > md5.txt
$ md5sum -c md5.txt
FUSCCTNBC001.TT_WES_R1.fastq.gz: OK
FUSCCTNBC001.TT_WES_R2.fastq.gz: OK
我们也可以和我们从EBI 上下载的数据验证一下md5,查阅一下FUSCCTNBC001 对应的SRR 号为SRR8517928: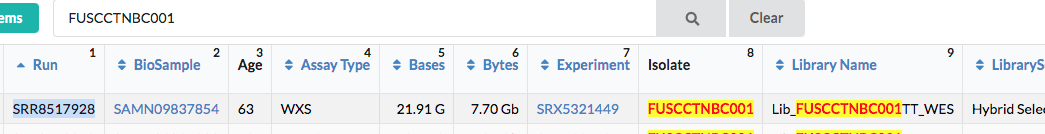
它们的md5sum 竟然是不同的。
$ md5sum FUSCCTNBC001.TT_WES_R1.fastq.gz
d8681c1944d882f5d86ac7f22867f95c FUSCCTNBC001.TT_WES_R1.fastq.gz
$ md5sum /home/data/tnbc/public_data/FUSCCTNBC/SRR8517928_1.fastq.gz
c6b7aa4eaeb0d9ac31ed90759076d638 /home/data/tnbc/public_data/FUSCCTNBC/SRR8517928_1.fastq.gz
大小也不同:
-rw-r--r-- 1 tnbc tnbc 5.2G 12月 29 10:18 SRR8517928_1.fastq.gz
-rw-rw-r-- 1 yzpeng yzpeng 5.1G 1月 18 11:22 FUSCCTNBC001.TT_WES_R1.fastq.gz
node 下载的数据要偏小一些。
会不会两个数据是分别存放的是肿瘤和正常对照的样本呢?
看了一下前面几行:
$ zcat FUSCCTNBC001.TT_WES_R1.fastq.gz | head
@ST-E00167:149:HLFWNCCXX:3:1101:5477:1625 1:N:0:CAGATCTG
NATATACTCAAAAGAACTGAAAGCAATGCAGGCTGGCTGTGGTAGCTCACACCTGTAATCCTAGCATGTTGGGAGGCTGAGGAGGGTAGATCGGAAGAGCACACGTCTGAACTCCAGTCACCAGATCTGATCTCGTATGCCGTCTTCTGC
+
#AAFFAKFK<FFKKKKKFKKKFKKKK,FA<KFA<<<(,AKK<F,FKAAFAKK,7<7KK7FKFKKKKKKKAFKKFKFA<,AKKA7F7,A7K7FAFFK7FF,FAKFKF,7FAFKFK7FFAFFKKFKKA<7AK7,,,AFKKF,,AA(,,7,,A
$ zcat /home/data/tnbc/public_data/FUSCCTNBC/SRR8517928_1.fastq.gz | head
@SRR8517928.1 1/1
NATATACTCAAAAGAACTGAAAGCAATGCAGGCTGGCTGTGGTAGCTCACACCTGTAATCCTAGCATGTTGGGAGGCTGAGGAGGGTAGATCGGAAGAGCACACGTCTGAACTCCAGTCACCAGATCTGATCTCGTATGCCGTCTTCTGC
+
#AAFFAKFK<FFKKKKKFKKKFKKKK,FA<KFA<<<(,AKK<F,FKAAFAKK,7<7KK7FKFKKKKKKKAFKKFKFA<,AKKA7F7,A7K7FAFFK7FF,FAKFKF,7FAFKFK7FFAFFKKFKKA<7AK7,,,AFKKF,,AA(,,7,,A
再看看最后一段:
@SRR8517928.73046074 73046074/1
CCNGGGCTCGGACGAGACCGACTTAATCCTCCTAGTTTGGCAAGTGGTTGAGCCGCGGAGCCGCCAGGTAGGGTGGAAAAGAGGCGGTGAGGTCCCCAGGGAGCTTCCGGGGTCGAGGGGCGCGGCTNATCGGTTTCCCNCGCCGCTCAC
+
AA#FFKKKKKKKKKKKKKKKKKFKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKFKKKKKKKKKKKKKKKKK(FFKKKKFKKKKKKKKKKKKAKKKKKKKKKKKKKKKKKKKKF#KFKFKKKKKKK#K<KKFKFKKK
@ST-E00167:149:HLFWNCCXX:3:2224:24515:73036 1:N:0:CAGATCTG
CCNGGGCTCGGACGAGACCGACTTAATCCTCCTAGTTTGGCAAGTGGTTGAGCCGCGGAGCCGCCAGGTAGGGTGGAAAAGAGGCGGTGAGGTCCCCAGGGAGCTTCCGGGGTCGAGGGGCGCGGCTNATCGGTTTCCCNCGCCGCTCAC
+
AA#FFKKKKKKKKKKKKKKKKKFKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKKFKKKKKKKKKKKKKKKKK(FFKKKKFKKKKKKKKKKKKAKKKKKKKKKKKKKKKKKKKKF#KFKFKKKKKKK#K<KKFKFKKK
数据是一模一样的。可能真的只是第一行记录的read 标识符不同造成的差别吧。
总的来说,感觉node 的使用体验还是真不戳的。
需要注意的是,虽然大部分上传的NGS 数据都是打着public 的招牌:

可是有很多的内容还是需要获取作者的许可的: