看到于2017年发表在Cancer Res 杂志的文章;E3 Ubiquitin Ligase UBR5 Drives the Growth and Metastasis of Triple-Negative Breast Cancer. 做的是肿瘤外显子数据,最后是: An analysis of primary TNBC specimen by whole-exon sequencing revealed strong gene amplifications of UBR5 associated with the disease.
但是居然莫名其妙的就定位到了UBR5基因,而且这就是它全文的亮点: Here, we report for the first time a distinctive and profound role of the E3 ubiquitin ligase UBR5 in the growth and metastasis of TNBC. 这样的结论基本上等于没有结论,因为找到一个在三阴性乳腺癌里面有重要作用的基因简直是小菜一碟,人类可是有2万多个基因啊,仅仅是抑癌基因和原癌基因都超过两千个了。虽然是WES数据,但是主要关注点是CNV 情况
文章写明了病人来自于中南大学的湘雅医院,数据如下:
- A total of 21 pairs of TNBC samples and adjacent noncancer tissue samples were obtained from patients
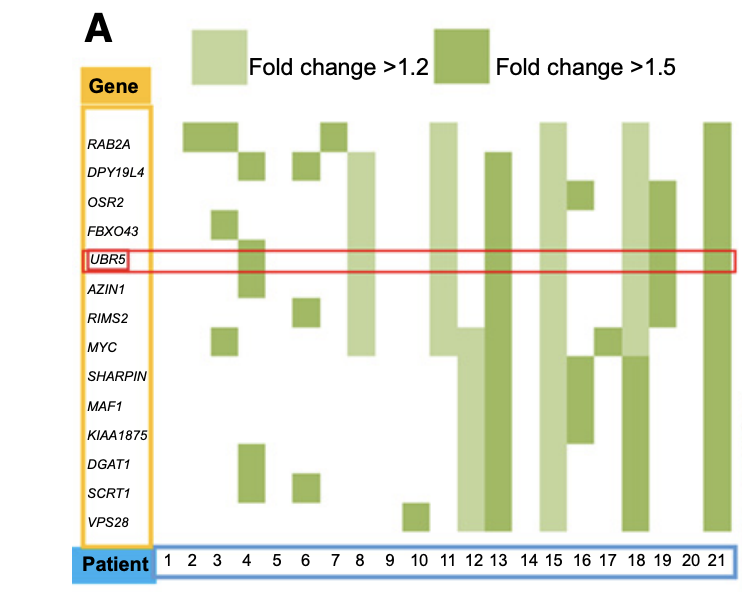
- Many genes showing significant amplifications (fold change >1.2) in these samples across multiple patients
文章提到了: Next generation of whole-exon sequencing was performed by Genomics Core Facilities at Weill Cornell Medicine. 但是并没有给出测序数据,甚至没有数据分析的描述部分。不给出maf文件格式的somatic突变位点列表,也不给segment格式的拷贝数情况文件。
关于肿瘤外显子数据,研究者们并没有给出来突变全景图,在我看来是一个很大的败笔。另外,主要是分析居然是集中在CNV,但是也不是拷贝数全景图,是一个有意思的图表:
在拷贝数分析里面,fold change 是一个并不算是规范的表达方式,因为正常人的拷贝数肯定是2,所以其实应该是把基因的拷贝数区分成为 :- -2 全部缺失
- -1 缺失一个拷贝
- 0 正常的
- 1 增加一个拷贝
- 2 增加多个拷贝
更重要的,基因UBR5只不过是拷贝数变化的几千个基因的一个而已,凭什么后续就集中火力拿它讲生物学故事呢?不过大家更认可实验结果
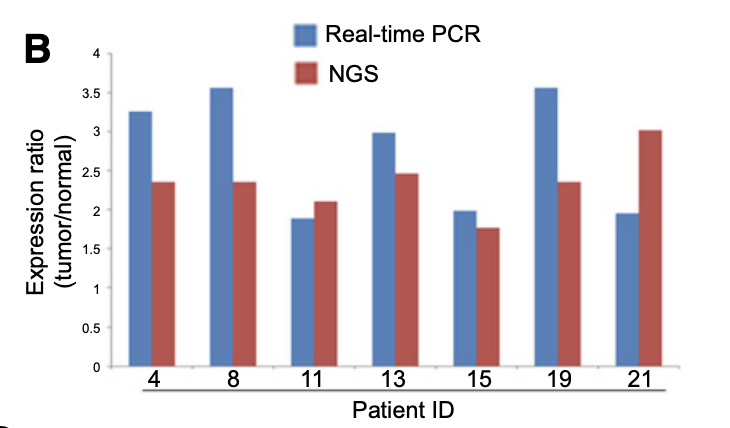
前面显示出病人4,8,11,13,15,19,21的基因UBR5出现了拷贝数增加,所以研究者使用了 RT-PCR实验验证。 而且也描述的很正规:Target gene primers were synthesized by Integrated DNA Technology, and amplification of endogenous b-actin was used as an internal control.
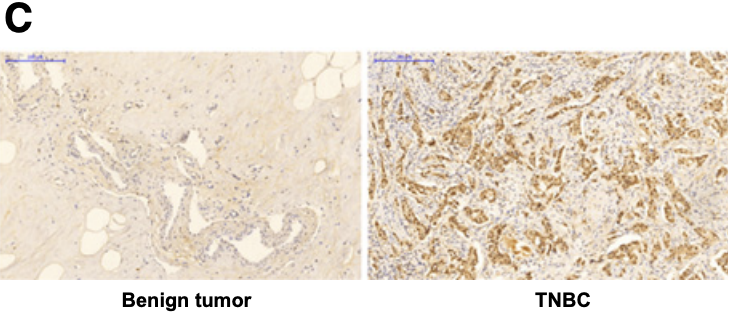
还做了IHC染色
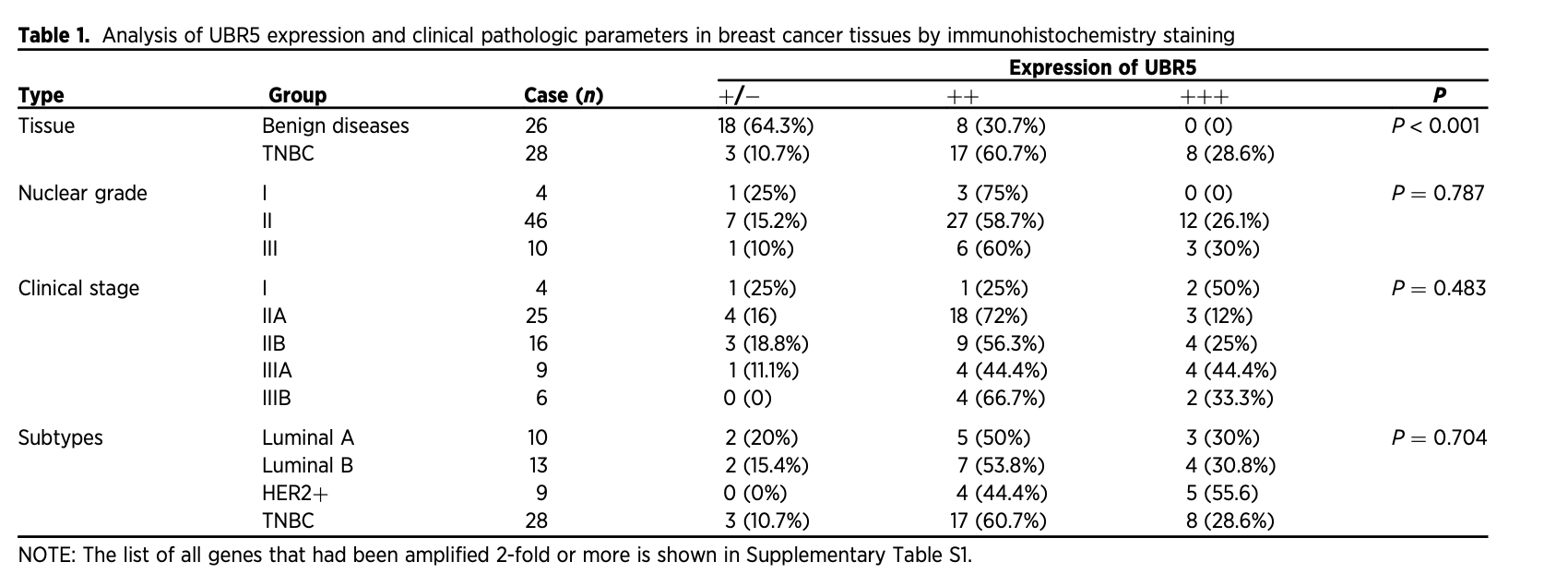
还有更详尽的IHC队列:
后面的篇幅全部是关于基因UBR5的湿实验了
主要是细胞系实验,包括:
- Sulforhodamine B assay
- Wound-healing assay
- Migration and invasion Transwell assay
- Cell adhesion assay
- Clonogenic assays
- Hanging drop assay
- Tumorigenesis studies
小鼠实验:- Quantification of number of macro- and micrometastases in lungs
- Histology, immunohistochemical, and TUNEL staining
- n a syngeneic murine model of transplanted TNBC in lieu of Ubr5-null mice
这些实验但凡是做肿瘤研究的朋友圈都了如指掌,我就不赘述了。学徒作业
TCGA数据库里面的乳腺癌患者有一千多个,即使是TNBC也有100多个,可以很容易下载到其maf文件格式的somatic突变位点列表以及segment格式的拷贝数情况文件。
我需要你仔细查看基因UBR5出现了拷贝数增加的病人在全部的TNBC病人队列的比例!以及给出其它跟基因UBR5拷贝数变化非常类似的基因列表。假如你不会探索TCGA数据库
我在B站有一个免费视频课程哈:
视频观看方式
- 课程B站地址:https://www.bilibili.com/video/av49363776
- TCGA教学视频售后文档记录 https://docs.qq.com/doc/DYkVzUmZLWlhRRXVz 请先通读文档后再发问
- 我这边备份的TCGA数据来源于xena,ucsc的,都在,https://share.weiyun.com/5zLnKmO
我喜欢把TCGA数据库的应用划分为8个领域:- 1、探索各类肿瘤不同临床特征(性别、年龄、种族、临床分期)的预后(生存曲线)
- 2、探索各类肿瘤与对照的单个分子(mRNA,lncRNA,miRNA,甲基化,蛋白)水平的差异情况(箱线图)
- 3、探索各类肿瘤与对照的全局(mRNA,lncRNA,miRNA,甲基化,蛋白)水平的差异情况(差异分析流程)
- 4、探索各类肿瘤中两个分子(mRNA,lncRNA,miRNA,甲基化,蛋白)水平相关性(散点图)
- 5、探索各类肿瘤中多个分子(mRNA,lncRNA,miRNA,甲基化,蛋白)水平总结(热图)
- 6、探索各类肿瘤中单个分子(mRNA,lncRNA,miRNA,甲基化,蛋白)与所有其它分子相关性并且排序
- 7、探索各类肿瘤中单个基因突变或者单个分子(mRNA,lncRNA,miRNA,甲基化,蛋白)水平的预后(生存曲线)
- 8、探索各类肿瘤不同临床特征(性别、年龄、种族、临床分期)分组后的单个分子(mRNA,lncRNA,miRNA,甲基化,蛋白)特性的分布
希望大家多交流最新TCGA数据应用,最新文献,最新技术哦!我的视频,仅仅是一个抛砖引玉哈!