看到于2020年11月发表在杂志《nature cancer》的文章:《Mutations in BRCA1 and BRCA2 differentially affect the tumor microenvironment and response to checkpoint blockade immunotherapy》里面有全基因组测序数据,文献链接是:https://www.nature.com/articles/s43018-020-00139-8
全基因组测序数据可以下载
7个WGS数据,怎么着也得人民币5万块钱!数据在 https://www.ncbi.nlm.nih.gov/bioproject/PRJNA632854 :
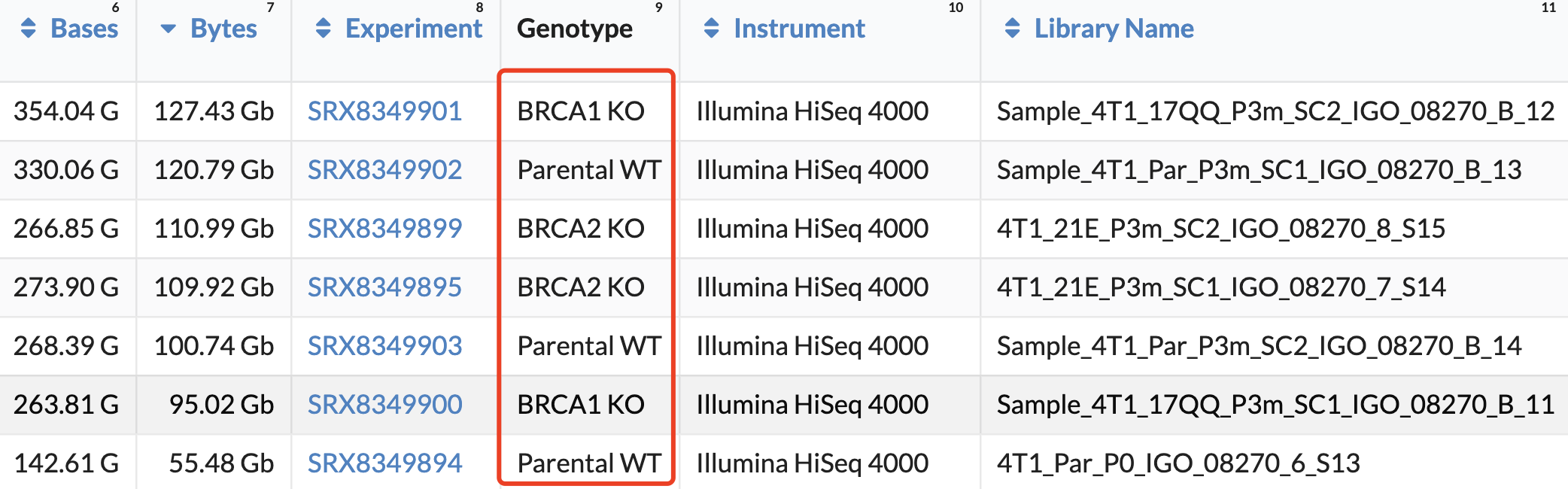
可以看到,我们能够下载这些数据,然后进行二次分析的!作为一个学徒作业吧,大家下载这些数据,走流程拿到snv和indel的vcf文件!
全基因组测序数据处理
文献的描述如下;

涉及到的软件如下:
- DNA was extracted and sequenced at 74–106× (median 80×) coverage.
- FASTQ files were aligned to MM10 mouse reference with bwamem v.0.7.15.
- Somatic mutations were called with VarScan v.2.4.3, Strelka v.2.9.10, Platypus 0.8.1, Mutect2 (part of GATK 4.1.4.1) and SomaticSniper 1.0.5.0 (for SNVs only).
- DNA from a 4T1 parental single-cell clone at time 0 was used as normal.
这些软件大多都有教程及文档,大家可以去《生信技能树》学习它们。我在B站分享了 免费视频课程《WES数据分析》 - 视频免费在B站:https://www.bilibili.com/video/BV15s411P7ay 大家学习的时候记得发弹幕交流哈
- 同步查看视频配套代码:https://www.jianshu.com/p/49d035b121b8
- WES 实战演练的素材:链接:https://share.weiyun.com/56MQHYG 密码:8jxwba 包括一些公司PPT,综述以及文献。
- WES 实战演练的思维导图:文档链接:https://mubu.com/doc/1iDucLlG5g 密码:7uch
拿到变异文件后
文献里面的关于变异文件的数据分析超级简陋,仅仅是看了看数量:
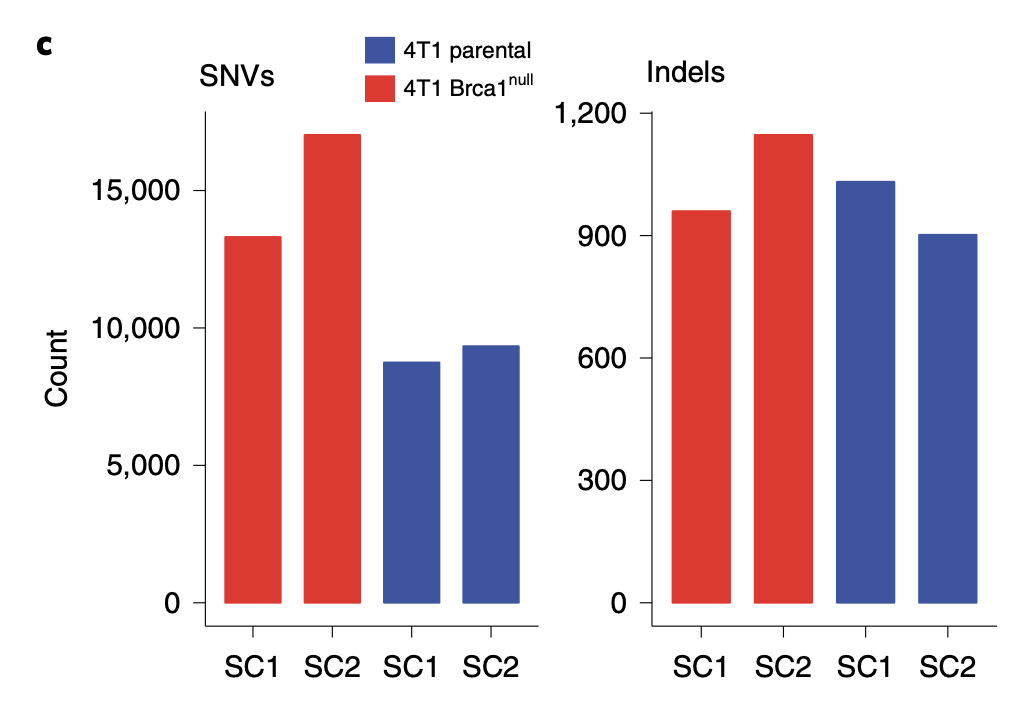
这个其实都不需要他们做小鼠数据的,因为TCGA数据库里面本来就是有BRCA1突变的人类和BRCA2突变的,也可以进行比较。
另外一个关于这些变异位点图表,也仅仅是超级简单,仅仅是看indel的长度
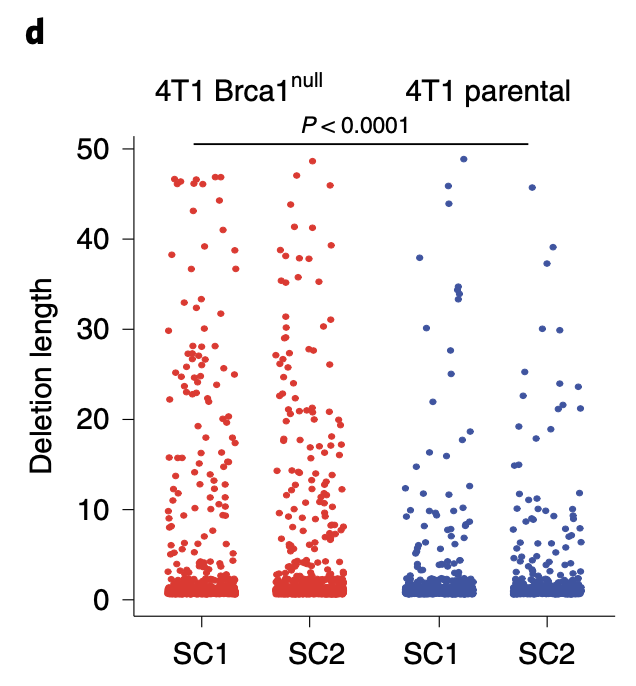
关于变异分类
主要可分为单个碱基对的变异(SNVs/SNPs)、小的插入或缺失(InDels≤50bp)以及结构变异(SVs>50bp)。 这里我直接摘抄:基因组变异类型详解及区分吧,免得浪费时间自己去整理了。
No.1 | 单个碱基对的变异
SNPs 与 SNVs,二者都是单核苷酸的改变,如果细究起来,还是有些区别的。SNPs一般是针对“群体”而言,且在群体中占据一定比例(well characterized),而SNVs一般是针对“个体”而言,发生频率非常低,不常见 (not well characterized)。
- 单核苷酸多态性(Single Nucleotide Polymorphisms,SNPs),单个核苷酸碱基的改变,包括置换、颠换、缺失和插入,导致的核酸序列的多态性,是人类最常见的遗传变异类型。由于具有遗传稳定性强、数量多、分布广等特点,SNP被广泛应用于群体遗传学以及疾病相关基因定位等研究中。
- 单核苷酸变异(Single Nucleotide Variants,SNVs)是DNA序列中单一核苷酸的变异。有单位点核苷置换,单位点核苷缺失,单位点核苷插入三种常见模式。其中置换模式为基因组上某一单位点核苷变异成另一核苷,缺失模式为基因组上某一位点的核苷缺失,插入模式为基因组上某一单位点核苷重复表达。
No.2 | 小的插入或缺失
插入和缺失( insertion-deletion,InDel),指的是在基因组的某个位置上所发生的小片段序列的插入或者缺失,其长度通常在50bp以下。与SNP不同的是,它并不是单个碱基的变化,而是在基因组中发生不同大小的DNA片段的插入或者缺失。它在基因组中的分布频率也是仅次于SNP,且很多都发生在基因内部甚至是外显子区域、启动子区域等重要位置。这种变异往往能够引起基因功能产生重大变化,同时InDel也是非常重要的一种基因组结构变异。
No.3 | 结构变异
结构变异(Structural Variation,SV)这种类型比较多,根据结构变异的不同类型,结构变异可以进一步分为50bp以上的长片段序列插入(Insertion) 、缺失(Deletion)、反转(Inversion)、染色体内易位(Intra-chromosomal Translocation)、染色体间易位 (Inter-chromosomal Translocation)、拷贝数变异(Copy Number Variation)以及一些形式更为复杂的变异。