最近因为课题需要,在分析数据集:GSE65212,我看了看平台是:GPL148777,写着 Affymetrix Human Genome U133 Plus 2.0 Array [Brainarray Version 13, HGU133Plus2_Hs_ENTREZG],这不就是jimmy授课讲解的那个应用最广泛的芯片嘛,这次分析,妥妥的!!!如下:
- https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE65212
- https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GPL14877
但是,我在在利用hgu133plus2.db进行探针名转换为基因名时出现问题 ,代码如下:library(hgu133plus2.db) ids=toTable(hgu133plus2SYMBOL) #toTable这个函数:通过看hgu133plus2.db这个包的说明书知道提取probe_id(探针名)和symbol(基因名)的对应关系的表达矩阵的函数为toTable head(ids) #head为查看前六行 colnames(ids)=c('probe_id','symbol') ids=ids[ids$symbol != '',] dim(ids) # 41922 2 ids=ids[ids$probe_id %in% rownames(dat),] dim(ids) # 165 2转换完成后数据只剩162行 ???顿时不淡定了,然后去仔细看了一下平台信息:
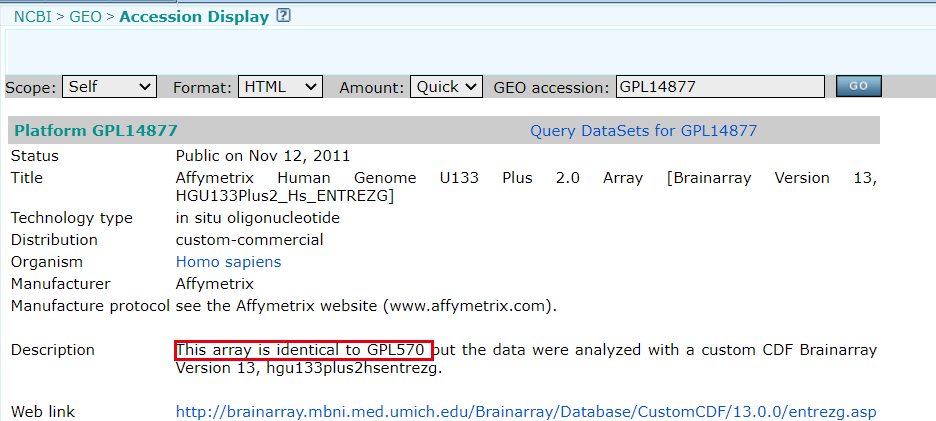
这个 Affymetrix Human Genome U133 Plus 2.0 Array [Brainarray Version 13, HGU133Plus2_Hs_ENTREZG],,上面写的是 芯片与GPL570相同,我怀疑是R包hgu133plus2.db的问题,所以使用jimmy老师的 AnnoProberm(list = ls()) ## 魔幻操作,一键清空~ options(stringsAsFactors = F) library(AnnoProbe) GPL570 <- idmap("GPL570") GPL14877 <- idmap("GPL14877") # Error in idmap("GPL14877") : # This platform is not in our list, please use our shinyAPP to custom annotate your probe sequences, or ask us to process and then update the R package!尴尬了,发现这个 GPL14877 根本就不在常见的160个芯片里面,唉!!!
最后最好是,直接网页下载读取,在https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GPL14877 找到如下信息:
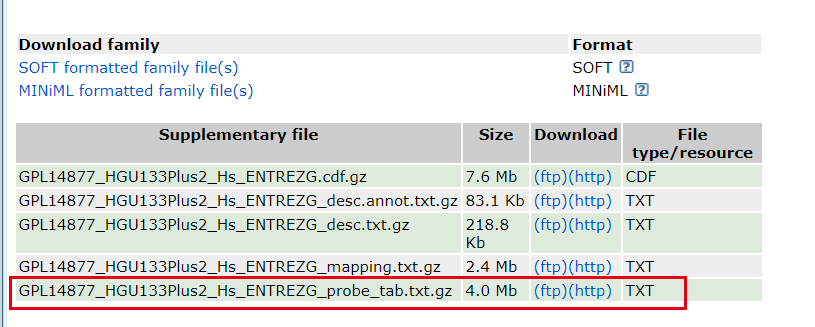
GPL14877 <-read.table("GPL14877_HGU133Plus2_Hs_ENTREZG_probe_tab.txt.gz",sep = "\t",header = T ) # 看一下数据 head(GPL570) head(GPL14877) library(hgu133plus2.db) ids=toTable(hgu133plus2SYMBOL) head(ids) #head为查看前六行 table(GPL14877$Probe.Set.Name %in% GPL570$probe_id) table(GPL14877$Probe.Set.Name %in% ids$probe_id) table(GPL570$probe_id %in% ids$probe_id)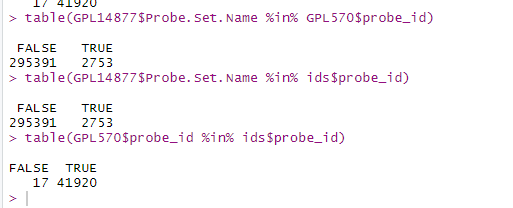
真的非常的意外啊!!!GPL14877 与 GPL570 不一样 ?
我再次看:
This array is identical to GPL570 but the data were analyzed with a custom CDF Brainarray Version 13, hgu133plus2hsentrezg.
所以这句话我的理解有问题?还是下载的文件有问题?
接着尝试下了下图mapping文件,拼老命也得干掉这个疑问!
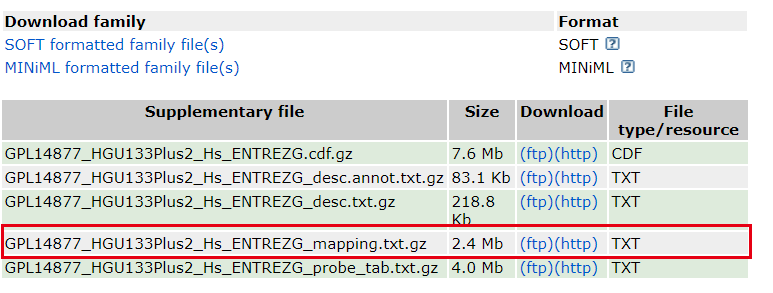
下载读取后发现两列探针名:
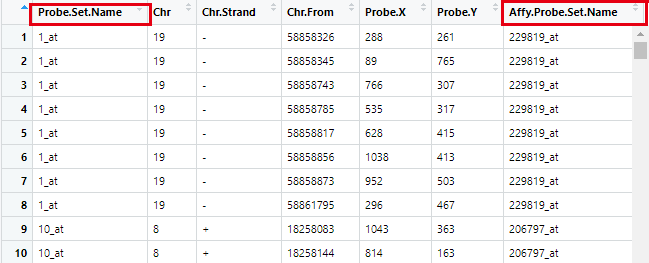
为什么这样的探针也是可以匹配呢?
用Affy Probe Set Name去匹配GPL570里的探针table(gpl2$Affy.Probe.Set.Name %in% GPL570$probe_id) FALSE TRUE 11356 285688这个匹配率好多了,先看看是否为正确匹配!
a <- gpl2[gpl2$Affy.Probe.Set.Name %in% GPL570$probe_id,] head(a) Probe.Set.Name Chr Chr.Strand Chr.From Probe.X Probe.Y Affy.Probe.Set.Name 1 1_at 19 - 58858326 288 261 229819_at 2 1_at 19 - 58858345 89 765 229819_at 3 1_at 19 - 58858743 766 307 229819_at 4 1_at 19 - 58858785 535 317 229819_at 5 1_at 19 - 58858817 628 415 229819_at 6 1_at 19 - 58858856 1038 413 229819_atGPL570 中 229819_at 对应A1BG 基因
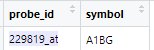

GPL14887也是这个基因!
终于对了!!
稀里糊涂一个星期又过去了!!!
气死我了!!!