我有一个 免费视频课程《ChIP-seq数据分析》 ,其视频观看方式:
- 首先视频免费共享在B站:https://www.bilibili.com/video/BV16s411T7Fh
- 同步查看视频配套代码 :https://www.jianshu.com/p/1384173c353b
- ChIP-SEQ实战演练的素材:链接:https://share.weiyun.com/53CwQ8B 密码:ju3rrh, 包括一些公司PPT,综述以及文献
- ChIP-SEQ 实战演练的思维导图:文档链接:https://mubu.com/doc/11taEb9ZYg 密码:wk29
但是呢,里面关于文献测序数据那一块其实是过气了,因为sra-tools的prefetch下载方式并不适合中国大陆的用户,简单的参考:使用ebi数据库直接下载fastq测序数据 , 需要自行配置,其实就可以解决数据下载的问题啦。
数据下载仅仅是一个开始,后续的流程化分析才是大戏,也有很多粉丝follow了我们的教程完成了系列图表最后仍然是云里雾里。其实是因为文献看得不够多,并不理解这个测序技术的来龙去脉以及它的花式应用。而且我们的视频课程因为时间关系,仅仅是给大家一个最简单的例子啦,我最近梳理文献发现了一个更好的,甚至可以说是最适合ChIP-seq实战的文献推荐。
那就是发表在 Genome Res . 2018 Feb; 的文章:Enhancer transcription reveals subtype-specific gene expression programs controlling breast cancer pathogenesis
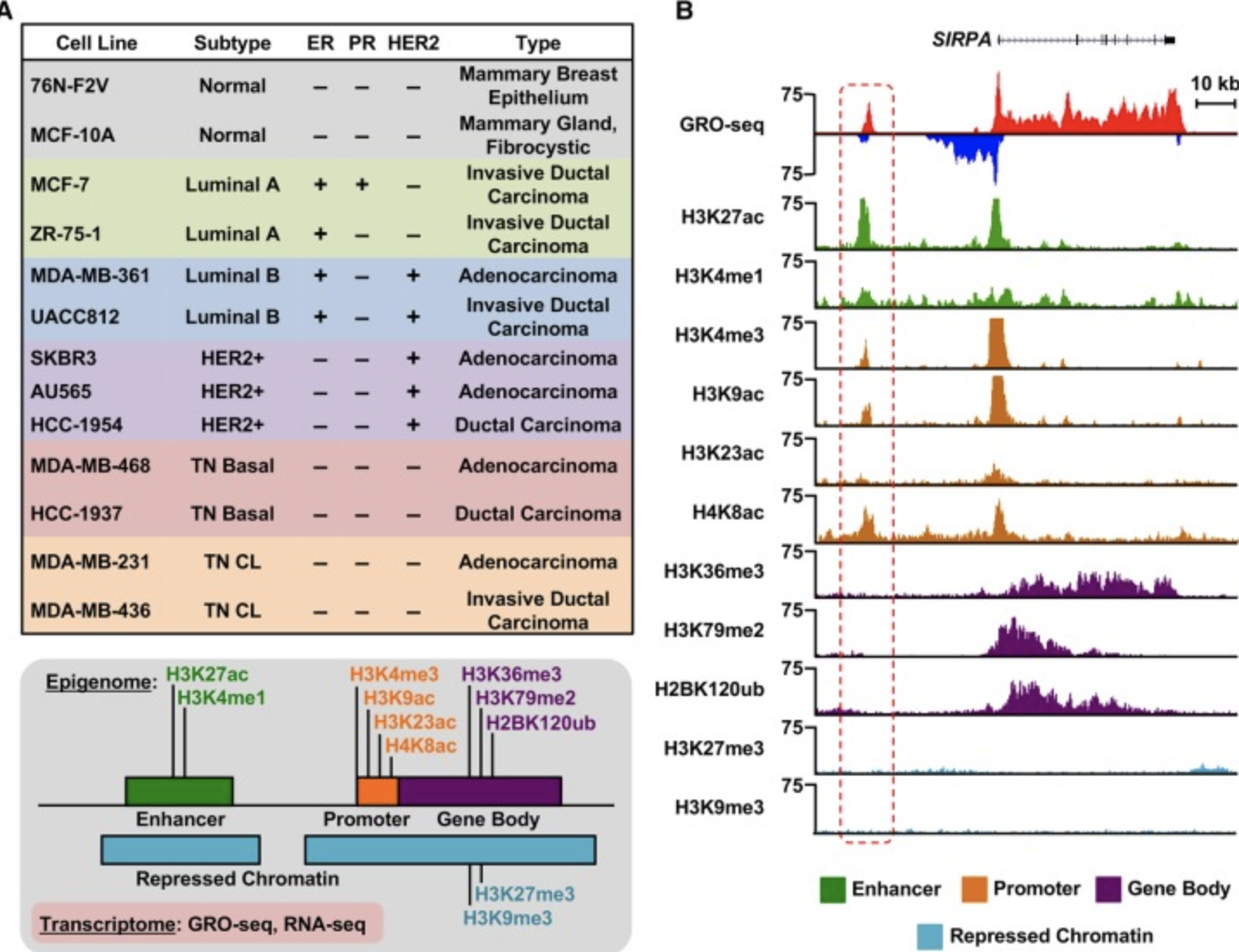
数据都是公开的,在:https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE85158 - 涉及到13个细胞系:76NF2V, MCF10A, MCF7, ZR751, MB361, UACC812, SKBR3, AU565, HCC1954, MB231, MB436, MB468, and HCC1937 cells.
- 有11种组蛋白修饰: H3K4me1, H3K4me3, H3K27me3, H3K36me3, H3K9me3, H3K9ac, H3K79me2, H3K27ac, H2BK120ub1, H3K23ac, and H4K8ac
文章的落脚点是:enhancer identification pipeline
不过,你跟着我流程可以拿到全部的ChIP-seq样品的bw文件,然后载入IGV后作出上面的(B) - Genome browser views of GRO-seq and histone modification ChIP-seq data from a normal breast epithelial cell line (76N-F2V) showing the features of a typical bidirectionally transcribed enhancer (red box with dashed line) and its nearest neighboring gene (SIRPA).
- Key features include transcription (red/blue), as well as histone modifications typically enriched at enhancers (green), promoters (brown), gene bodies (purple), and repressed chromatin (turquoise).
起码可以马上理解信号值的概念了!
还跟转录水平的数据相互验证:GRO-seq and RNA-seq作为学徒作业
这个作业起点有一点高,首先你得有自己的服务器了,然后才有可能下载这两百多个ChIP-seq样品的测序数据走完我给出来的流程,得到全部的bw文件,然后载入IGV后作出上面的图!