上个月我们组建了:《单细胞CNS图表复现交流群》,见:你要的rmarkdown文献图表复现全套代码来了(单细胞),也分享了单细胞转录组数据分析的流程:
交流群里大家讨论的热火朝天,而且也都开始了图表复现之旅,在这里我还是带大家一步步学习CNS图表吧。如果你也想加入交流群,自己去:你要的rmarkdown文献图表复现全套代码来了(单细胞)找到我们的拉群小助手哈。
今天讲解第三步:根据一些基因的表达来区分细胞是否属于免疫细胞。
我在单细胞天地的教程:是否是免疫细胞很容易区分那是否是肿瘤细胞呢?提到过Cells were defined as non-immune if belonging to a cluster low for PTPRC (gene for CD45)
rm(list=ls())
options(stringsAsFactors = F)
library(Seurat)
library(ggplot2)
load(file = 'first_sce.Rdata')
# Specify genes
genes_to_check = c("PTPRC","EPCAM","CD3G","CD3E", "CD79A", "BLNK","MS4A1", "CD68", "CSF1R",
"MARCO", "CD207", "PMEL", "ALB", "C1QB", "CLDN5", "FCGR3B", "COL1A1")
# All on Dotplot
p <- DotPlot(sce, features = genes_to_check) + coord_flip()
p
可以看到;
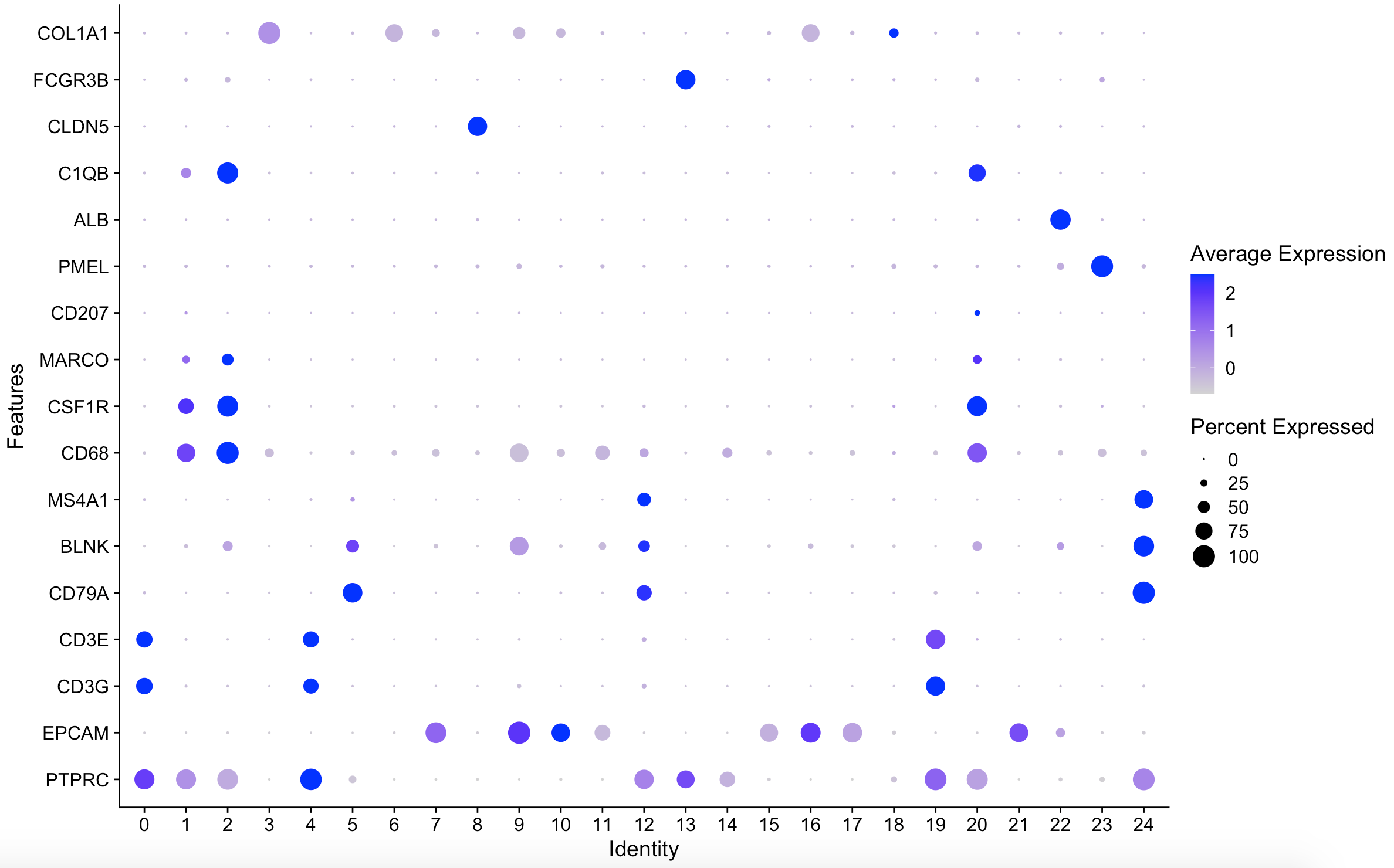
其中PTPRC基因代表的是CD45分子,是免疫细胞的标记,所以可以使用它来区分:
# Annotate Immune vs Nonimmune clusters
# At this point we dont care for a more detailed annotation as we will annotate immune and non-immune separately later
dat=p$data
cd45=dat[dat$features.plot=='PTPRC',]
fivenum(cd45$avg.exp.scaled)
imm=cd45[cd45$avg.exp.scaled > -0.5,]$id
imm
sce@meta.data$immune_annotation <-ifelse(sce@meta.data$RNA_snn_res.0.5 %in% imm ,'immune','non-immune')
# MAke a table
table(sce@meta.data$immune_annotation)
# Make and save relevant plots
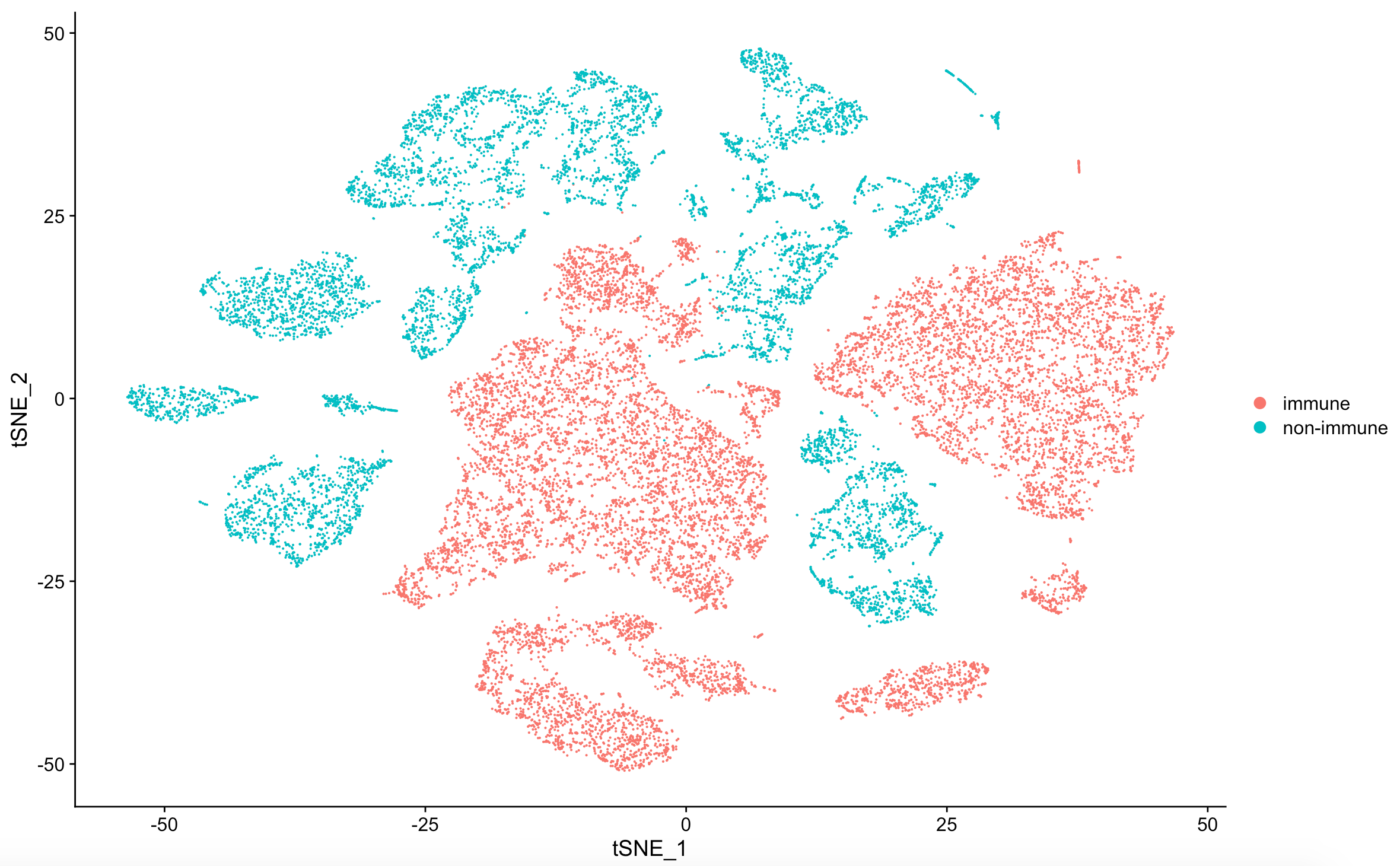
接下来可以进行 TSNE plot 可视化,看到免疫细胞和非免疫细胞是泾渭分明:
p <- TSNEPlot(object = sce, group.by = 'immune_annotation')
p
文末友情推荐
要想真正入门生物信息学建议务必购买全套书籍,一点一滴攻克计算机基础知识,书单在:什么,生信入门全套书籍仅需160 。
如果大家没有时间自行慢慢摸索着学习,可以考虑我们生信技能树官方举办的学习班:
- 数据挖掘学习班第7期(线上直播3周,马拉松式陪伴,带你入门),原价4800的数据挖掘全套课程, 疫情期间半价即可抢购。
- 生信爆款入门-第9期(线上直播4周,马拉松式陪伴,带你入门),原价9600的生信入门全套课程,疫情期间3.3折即可抢购。
如果你课题涉及到转录组,欢迎添加一对一客服:详见:你还在花三五万做一个单细胞转录组吗?
号外:生信技能树知识整理实习生招募,长期招募,也可以简单参与软件测评笔记撰写,开启你的分享人生!另外,:绝大部分生信技能树粉丝都没有机会加我微信,已经多次满了5000好友,所以我开通了一个微信好友,前100名添加我,仅需150元即可,3折优惠期机会不容错过哈。我的微信小号二维码在:0元,10小时教学视频直播《跟着百度李彦宏学习肿瘤基因组测序数据分析》