众所周知,在R里面使用cor函数可以计算两个向量的相似情况,有两个参数尤为需要注意:
其中method参数是:One of "pearson" (default), "kendall", or "spearman": can be abbreviated.
然后use参数是:This must be (an abbreviation of) one of the strings "everything", "all.obs", "complete.obs", "na.or.complete", or "pairwise.complete.obs".
本来呢,pearson,kendall以及spearman这3个相关性公式就让人头疼了,但是最近我在教程:比较不同的肿瘤somatic突变的signature 发现两个不同算法的signature的相似性并不是和文章完全一致,原因是作者使用了一个cosine similarity(余弦相似度)的概念。
cosine similarity(余弦相似度)如何计算
简单搜索了一下它的介绍:
- 余弦值的范围在[-1,1]之间,值越趋近于1,代表两个向量的方向越接近;越趋近于-1,他们的方向越相反;接近于0,表示两个向量近乎于正交。
- 最常见的应用就是计算文本相似度。将两个文本根据他们词,建立两个向量,计算这两个向量的余弦值,就可以知道两个文本在统计学方法中他们的相似度情况。实践证明,这是一个非常有效的方法。
第一次搜索它在R里面的用法,发现了tcR包里面的cosine.similarity函数,就简单试用了一下。但是计算得到的结果很诡异,并不是范围在[-1,1]之间。
再次尝试搜索cosine similarity(余弦相似度),发现在 The repertoire of mutational signatures in human cancer 文章里面也提到了:

为何使用cosine similarity(余弦相似度)而不是简单的相关性系数呢?
前面我们搜索了解到,cosine similarity(余弦相似度)最常见的应用就是计算文本相似度,那么,为什么生物信息学领域里面的cosmic的signature的相似性要采用cosine similarity(余弦相似度)而不是常见的简单的相关性系数呢?
比如,同样的是对cosmic内置的30个signature互相计算相关性,如下:# https://cancer.sanger.ac.uk/cancergenome/assets/signatures_probabilities.txt cosmic=read.table('https://cancer.sanger.ac.uk/cancergenome/assets/signatures_probabilities.txt', header = T,sep = '\t')[,1:33] head(cosmic[,1:4]) M=cor(cosmic[,4:33]) pheatmap::pheatmap(M)出图如下:
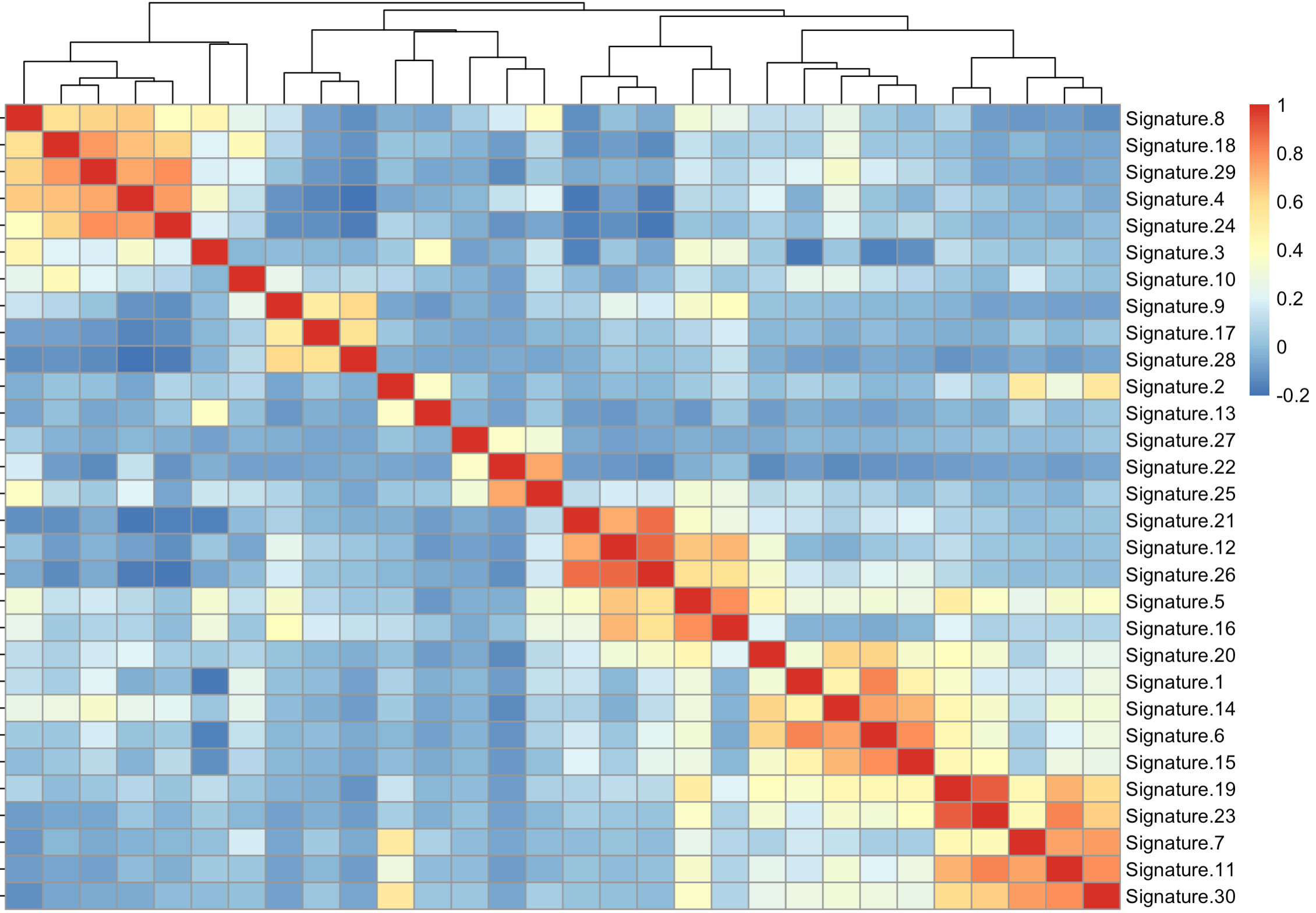
而计算cosine similarity(余弦相似度)代码如下;# 具体数学公式参考:https://www.jianshu.com/p/a894ebba4a1a cos=function(x,y){ sum(x * y) / (sqrt(sum(x ^ 2)) * sqrt(sum(y ^ 2))); } M2=apply(cosmic[,4:33], 2, function(x){ apply(cosmic[,4:33], 2, function(y){ cos(x,y) }) }) rownames(M2)=rownames(M) pheatmap::pheatmap(M2)出图如下:
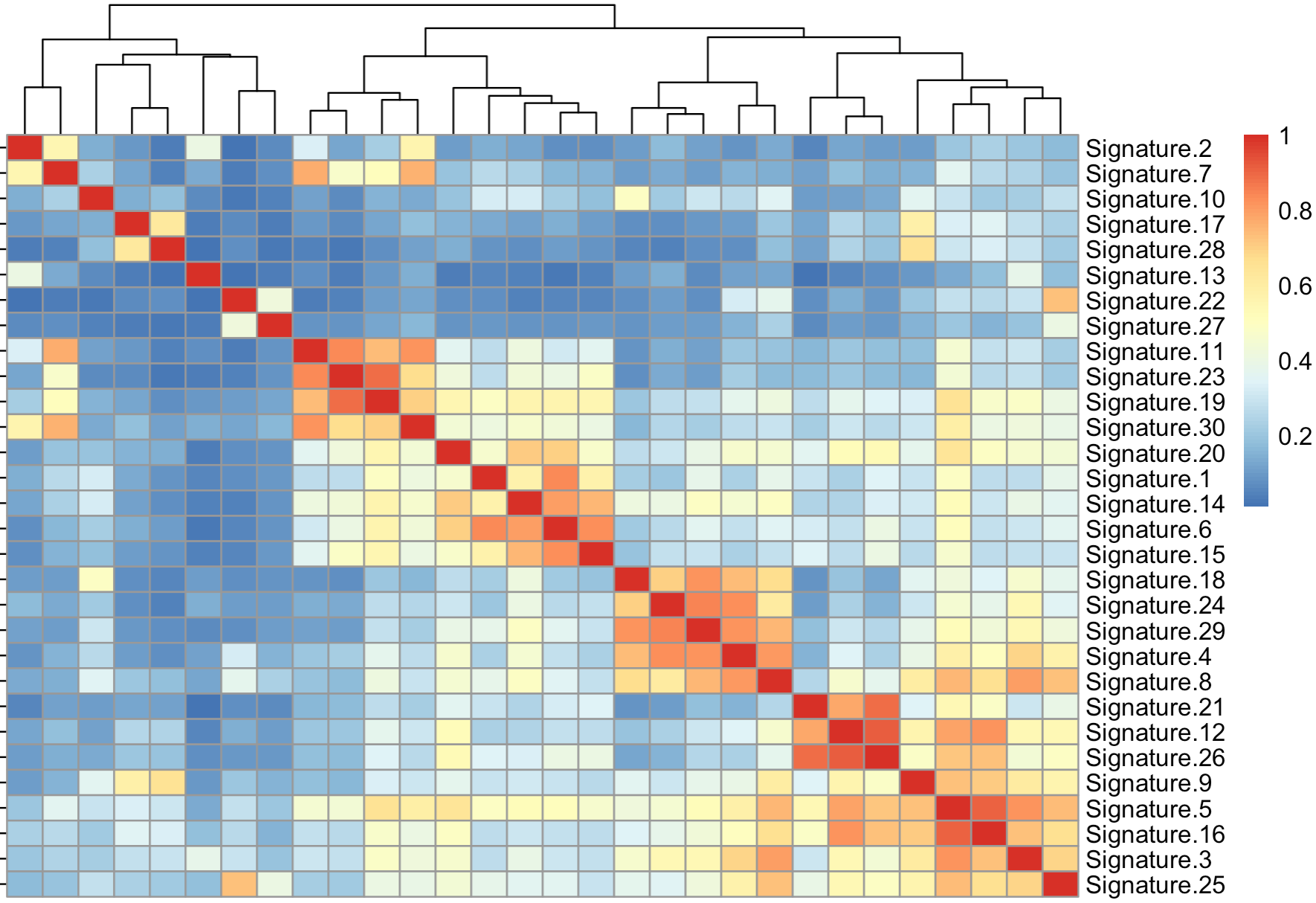
虽然我做了探索,但是我其实并不明白为什么cosmic的signature的相似性要采用cosine similarity(余弦相似度)而不是常见的简单的相关性系数。
不过上面的代码亲测可以用,打开你的R即可,如果你觉得这些代码很好玩,欢迎参加我们的生信技能树官方举办的学习班: - 数据挖掘学习班第5期(线上直播3周,马拉松式陪伴,带你入门),原价4800的数据挖掘全套课程, 疫情期间半价即可抢购。
- 生信爆款入门-第7期(线上直播4周,马拉松式陪伴,带你入门),原价9600的生信入门全套课程,疫情期间3.3折即可抢购。
文末友情推荐
要想真正入门生物信息学建议务必购买全套书籍,一点一滴攻克计算机基础知识,书单在:什么,生信入门全套书籍仅需160 。
如果大家没有时间自行慢慢摸索着学习,可以考虑我们生信技能树官方举办的学习班: - 数据挖掘学习班第5期(线上直播3周,马拉松式陪伴,带你入门),原价4800的数据挖掘全套课程, 疫情期间半价即可抢购。
- 生信爆款入门-第7期(线上直播4周,马拉松式陪伴,带你入门),原价9600的生信入门全套课程,疫情期间3.3折即可抢购。
如果你课题涉及到转录组,欢迎添加一对一客服:详见:你还在花三五万做一个单细胞转录组吗?
号外:生信技能树知识整理实习生招募,长期招募,也可以简单参与软件测评笔记撰写,开启你的分享人生!另外:绝大部分生信技能树粉丝都没有机会加我微信,已经多次满了5000好友,所以我开通了一个微信好友,前100名添加我,仅需150元即可,3折优惠期机会不容错过哈。我的微信小号二维码在:0元,10小时教学视频直播《跟着百度李彦宏学习肿瘤基因组测序数据分析》