十多年前的普通转录组一个样品测序跟现在的单细胞转录组类似,成本也是一两万块钱,然后随便五六个样本分两组做一下差异分析,富集到通路就是一个文章。但是这些年过去了,转录组已经深入到生命科学领域的方方面面,区别是它不再是唯一的主角,现在转录组不到一千块钱一个样品,只要你有好的课题思路,好的实验设计,让转录组为你的课题增光添彩吧!
我这边分享的转录组流程通常是hisat2+featureCounts软件组合,见免费视频课程《RNA-seq数据分析》,最近读单细胞转录组文献,发现大家开始倾向于把自己做的单细胞转录组数据分析结果跟以前的传统转录组公共数据进行比较,比如下面的:
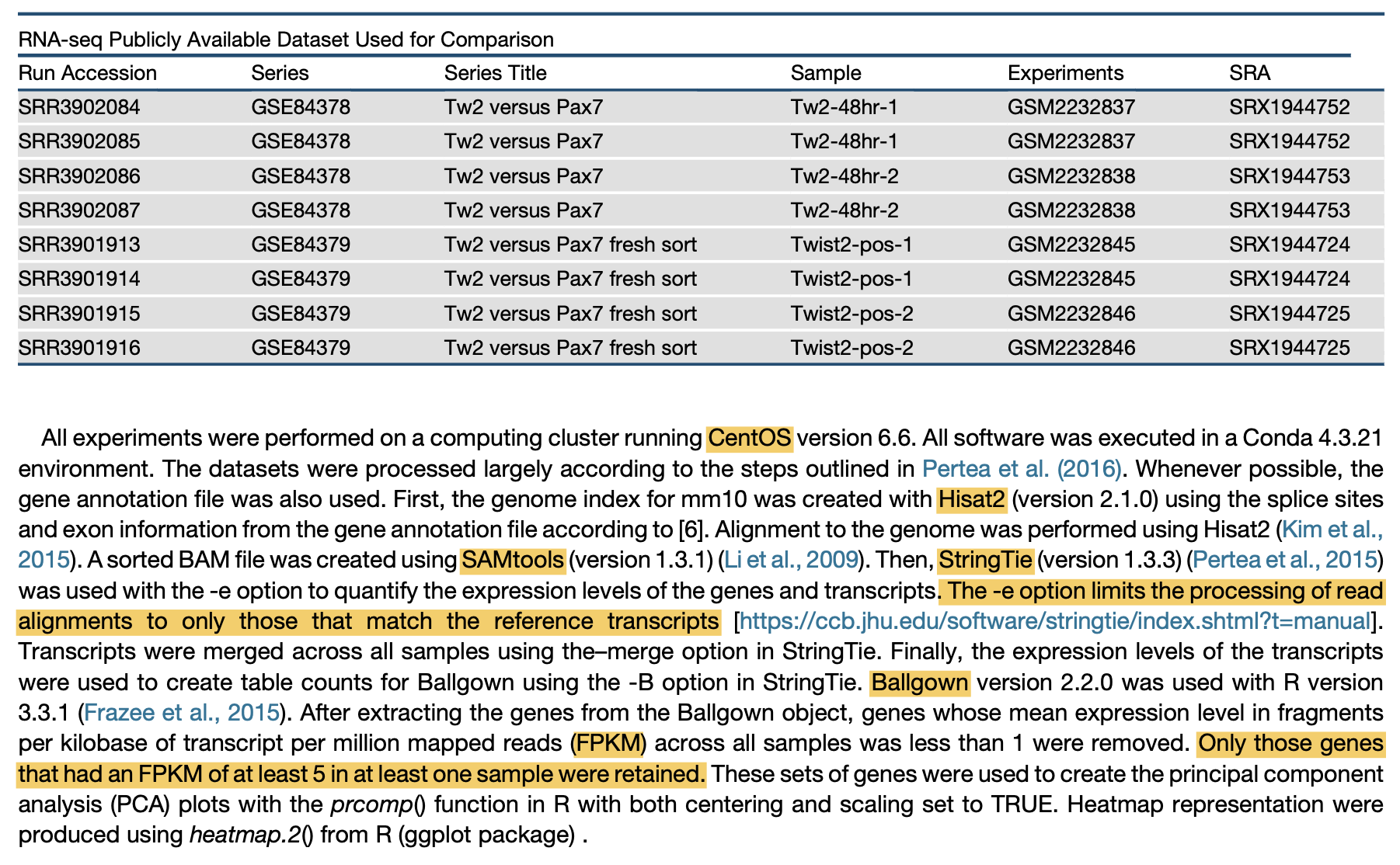
来源于文章:High-Dimensional Single-Cell Cartography Reveals Novel Skeletal Muscle-Resident Cell Populations,这个流程如果你会简单Linux其实很容易自己摸索一下。
其实这个hisat2+stringtie+ballgown流程还挺出名的, 引用早就破千了,如下:

我看到一个GitHub分享了简单shell代码,见:https://gist.github.com/mbk0asis/ac3309450f9648e98bec034377beba46
# For RNA-seq reads, use "--dta/--downstream-transcriptome-assembly"
# the command is similar to 'bowtie2'
$ hisat2 -x genome -1 read1.fq.gz -2 read2.fq.gz -S Sample.sam -p 8 --dta 2>&1 | tee $l.stat
$ samtools view -bS Sample.sam | samtools sort -@ 8 - Sample.sorted
# The bam files can be fed into other pipelines e.g DESeq, edgeR, and etc
# transcriptome assembly using 'stringtie'
$ stringtie -p 8 -G genes.gtf -o Sample.gtf –l Sample.sorted.bam
$ stringtie --merge -p 8 -G genes.gtf -o merged.gtf mergelist.txt
# expression level estimation
$ stringtie –e –B -p 8 -G merged.gtf -o Sample.gtf Sample.sorted.bam
# count data can be extracted from 'stringtie' output using 'prepDE.py',
# and you can use them for DESeq/edgeR analysis
# Run the differential expression analysis protocol using 'Ballgown'
这个就是学徒作业啦,使用aspera高速下载ebi数据库里面的这个转录组原始测序数据,然后走hisat2+stringtie+ballgown流程,摸索这些软件的用法,如果你能比较轻松的完成,欢迎参加:生信技能树知识整理实习生招募,长期招募,也可以简单参与软件测评笔记撰写,开启你的分享人生!
我五年前做过生物信息学百款软件教程:
- Web-app: webGO/David/SMS2/ExPaSy/ iPathwayGuide/
- Basic: DNAman/bioEDIT/mega/clustalw/blast/blast++/fastqc/SRA-toolkit/SolexaQA/NGS-QC-toolkit/bedtools/circos/cytoscape/Emboss/ cutadapt/fastx/
- Snp-calling: BWA/Bowtie2/samtools/picard/GATK/varscan/freebayes/bcftools/annovar/samStat/snpEFF/VEP/
- RNA-seq: RseQC/tophat2/STAR/Hisat/htseq/cufflinks/cummedund/edgeR/DeSeq/Trinity/TransDecoder/GMAP
- Evolution: muscle/figtree/PhyML/seq2HLA/Plink/
- Other: LiftOver/ PICNIC/mutsig/Matlab MCR/tRNAscan-SE/FLASH/igBlast/IGV
- R语言软件包:GEOquery/affy/limma/ GOstats/ConsensusClusterPlus/SPIA/ seqinr/ AnnotationHub/biostring/ GEOmetadb/biomaRt/ DawnRank
当时还录制了视频 - 大家可以去http://i.youku.com/trainee 上面找到我的全部视频,免费观看!
- 视频列表是http://www.bio-info-trainee.com/tmp/tutorial/video_list.html ,能顺利点击的链接代表视频录制完毕。
- 这次视频课程的大纲是http://www.bio-info-trainee.com/tmp/tutorial/syllabus.htm,但是很有可能会修改!
希望你完成我的学徒作业,然后加入我们!
期待···