还省掉了几百块钱的会员费!!!
最近有粉丝求助,说自己已经耗费十多天了还没有把一个NT数据库下载下来,不得已充值会员费希望可以加速,但是效果也很惨淡,如下图:
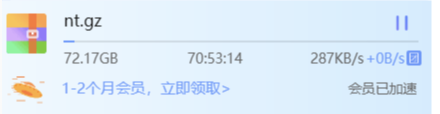
需要从 https://ftp.ncbi.nih.gov/blast/db/FASTA/ 下载核酸数据库nt.gz和nr.gz文件,这两个文件大的如此吓人(nr.gz:75G;nt.gz:72G)。用wget命令龟速(56.00K 7.03KB/s 剩余 124d),使用迅雷也就白天300KB/s晚上2MB。
我直接甩给他一个关键词:aspera
学员表示非常诧异,的确以前看到过我的教程,见:使用ebi数据库直接下载fastq测序数据 , 首先使用conda安装aspera
conda create -n download
conda activate download
conda install -y -c hcc aspera-cli
conda install -y -c bioconda sra-tools
which ascp
## 一定要搞清楚你的软件被conda安装在哪
ls -lh ~/miniconda3/etc/asperaweb_id_dsa.openssh
我们已经多次介绍过conda细节了,这里就不再赘述。
- conda管理生信软件一文就够
- 生信技能树B站软件安装视频
- https://www.bilibili.com/video/av28836717
可使用conda配置好的aspera软件进行高速下载,但是这个教程不是仅限于ebi的测序数据吗?
之后学员半信半疑的尝试拼接出来了下面的命令:
ascp -v -k 1 -T -l 200m -i ~/miniconda2/envs/rna/etc/asperaweb_id_dsa.openssh https://ftp.ncbi.nih.gov/blast/db/FASTA/nr.gz .
然后果然报错,如下:
ascp: no remote host specified
Startup failed, exit
我非常尴尬,首先学员没有认真看教程,没有扩展思维,其次,没有理解aspera下载是需要特殊的链接,我就又苦口婆心的语音指导了,成功写成下载链接如下:
# 安装完成后可以使用ascp --help查看帮助,Aspera需要私钥asperaweb_id_dsa.openssh
# 由于我使用conda安装的所以在~/miniconda2/envs/rna/etc中。
# 在/media/yang/data/nt目录下下载nt.gz
ascp -v -k 1 -T -l 200m -i ~/miniconda2/envs/rna/etc/asperaweb_id_dsa.openssh anonftp@ftp.ncbi.nlm.nih.gov:/blast/db/FASTA/nt.gz ./
# 然后在/media/yang/data/nt目录下下载nr.gz
ascp -v -k 1 -T -l 200m -i ~/miniconda2/envs/rna/etc/asperaweb_id_dsa.openssh anonftp@ftp.ncbi.nlm.nih.gov:/blast/db/FASTA/nr.gz ./
下载速度令人激动:

这才是:生命如此美好!
如果你学习生信也需要帮助,可以考虑我们生信技能树官方举办的学习班哈!
文末友情推荐
要想真正入门生物信息学建议务必购买全套书籍,一点一滴攻克计算机基础知识,书单在:什么,生信入门全套书籍仅需160 。
如果大家没有时间自行慢慢摸索着学习,可以考虑我们生信技能树官方举办的学习班:
如果你没有服务器的话,做NGS数据分析实战可能会有点勉强,建议考虑:每天不足一块钱,定制生信云送给你