在我们生信技能树好久以前的一个教程,看到了邢老师的留言,如下:

这个留言恰好回答了最近学员的一个疑问:在转录组大行其道的今天,简单的药物处理细胞系前后转录组数据分析结果如何玩出新意呢,毕竟一般来说就2个分组,6个样本,看起来的确是只能走差异分析策略。
其实奥秘就蕴藏在可变剪切,我们以刚刚在2020发表的一个药物处理细胞系前后转录组文章为例,像大家说明这个可变剪切的神奇。下一期我们就更新邢老师实验室出品的rmarts软件的4.1版本使用说明书。
文章标题是:Differential Expression and Alternative Splicing of Transcripts Associated With Cisplatin-Induced Chemoresistance in Nasopharyngeal Carcinoma,发的杂志很一般,因为确实实验设计超级简单,就是细胞系被药物处理前后转录组数据分析。
铂类化疗药物
Cisplatin (DDP), a DNA damaging agent, exhibits its cytotoxicity and apoptosis-inducing activities by forming DNA adducts or by targeting cancer signaling pathways
铂是一种贵重金属,铂类药物在临床上主要应用于肿瘤疾病的治疗,治疗机理主要是依靠铂类产品对细胞的细胞毒作用。
从1967年顺铂被人们发现具有抗癌活性以来,含铂金属的抗癌药物的应用和研究得到了前所未有的发展。最出名的介绍顺铂(DDP)
化疗药物有:DDP、奈达铂、5-FU及其衍生物、紫杉类、吉西他滨、长春瑞滨和伊立替康等!
鼻咽癌细胞系
Nasopharyngeal carcinoma (NPC) is one of the most highly invasive and metastatic head and neck cancers in southern China and Southeast Asia
我看到Int J Cancer. 1999 Sep 的文献:Nasopharyngeal Carcinoma Cell Line (C666-1) Consistently Harbouring Epstein-Barr Virus,原来那个年代,在实验室成功培养一个鼻咽癌细胞系。
我在:https://www.covance.com/industry-solutions/oncology/preclinical/complete-cell-line-list.html 查到的鼻咽癌细胞系很少。
转录组分析的一般流程
转录组的基本分析教程合辑:
- 上游分析视频以及代码资料在:https://share.weiyun.com/5QwKGxi
- 下游主要是基于counts矩阵的标准分析的代码 https://share.weiyun.com/50hfuLi
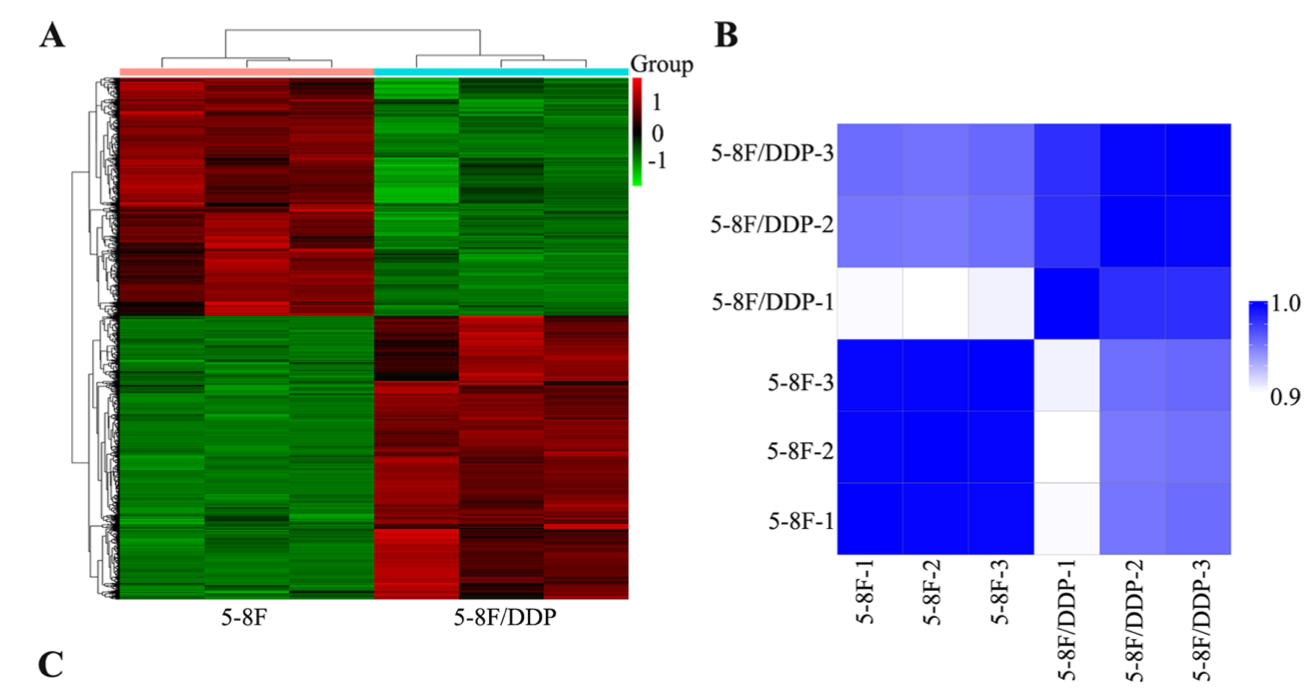
其实就是表达矩阵样本相关性,以及差异基因表达量热图可视化,可以看到这篇文章的细胞系药物处理前后的全局转录水平变化很大,组间差异也远大于组内差异,差异基因也非常明显。
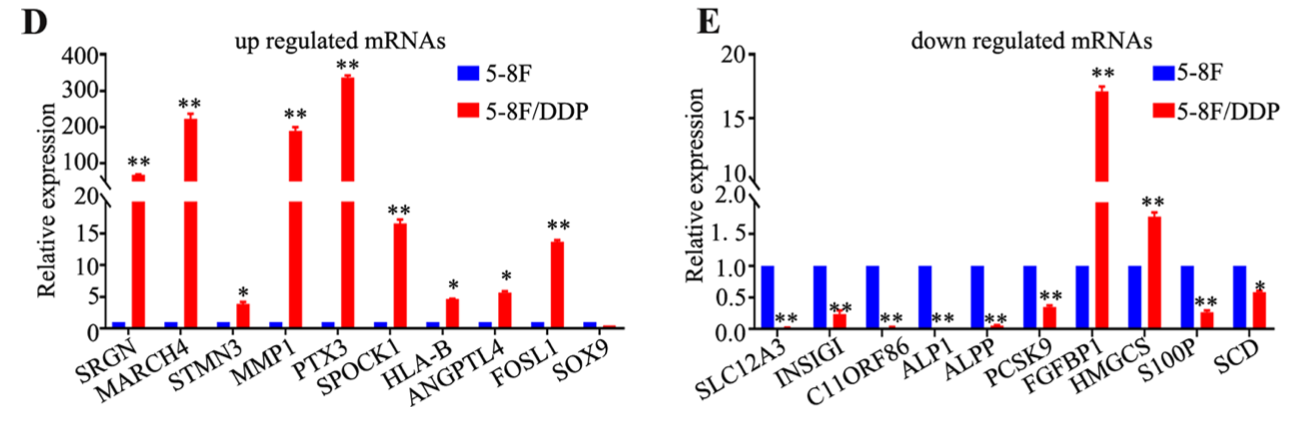
接着作者单独展示top的上下调基因,以截断Y轴条形图显示,其实就是左上角热图的具体基因而已:
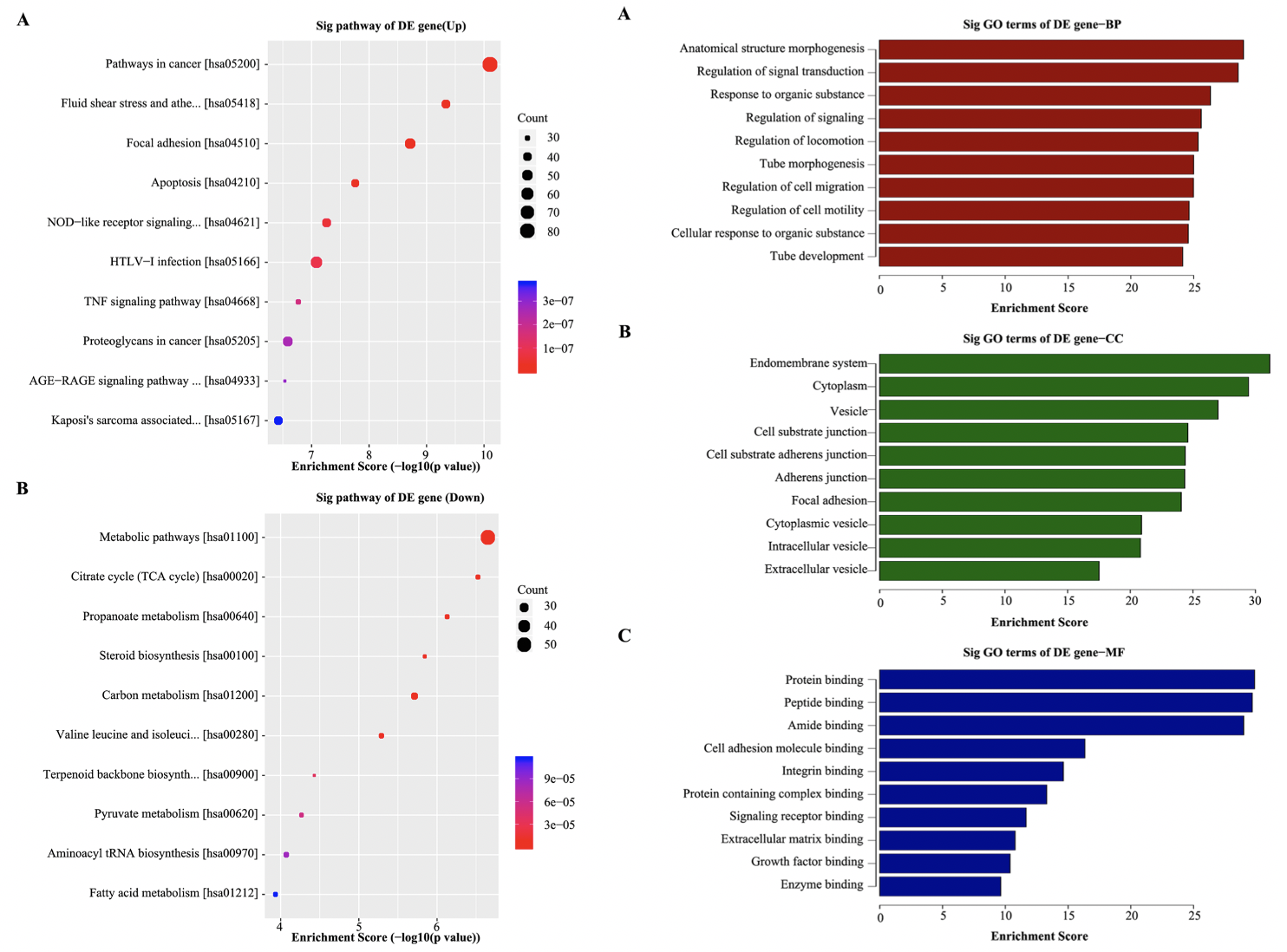
然后是go和kegg数据库对统计学显著的上下调基因的注释:
如果仅仅是做到这个步骤,其实很难发出去了,同质化超级严重,6个样本的转录组也就五六千块钱的消耗,一个博士生每个月的补贴都可以做两次这样的课题了。
既然已经是十年前的策略了,并没有什么新意,值得一提的反而是鼻咽癌领域的研究,因为这个癌症好发于中国大陆的两广地区,所以国内外研究鼻咽癌的实验室并不多!
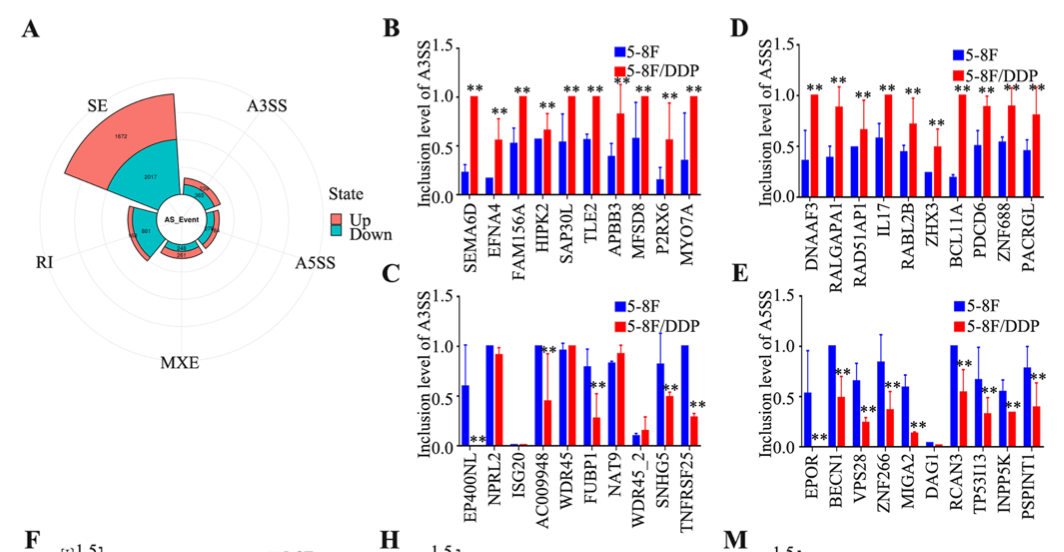
但是,这篇文章的研究者并没有把自己的思维仅限于我们生信技能树课题所讲解的知识点,进行了一个最小程度的扩展,就是可变剪切的查找,使用的就是邢老师实验室出品的rmarts软件,结果如下:
可变剪切丰富多样的软件工具
文章是Katz et. al., Analysis and design of RNA sequencing experiments for identifying isoform regulation. Nature Methods (2010). 可以说是比较早的方法,提出了很多基础概念。 软件说明书也写成了一本书那么复杂;https://miso.readthedocs.io/en/fastmiso
Multivariate Analysis of Transcript Splicing (MATS)软件列出5种可变剪切,分别是skipped exon (SE)外显子跳跃,alternative 5′ splice site (A5SS)第一个外显子可变剪切,alternative 3′ splice site (A3SS)最后一个外显子可变剪切,mutually exclusive exons (MXE)外显子选择性跳跃和 retained intron (RI)内含子滞留。
两种分析方法:
- Estimate expression level of exons (“exon-centric” analysis) or,
- Estimate expression level of whole transcripts (“isoform-centric” analysis)
也提出了可变剪切的分类: - Skipped exons (SE)
- Alternative 3’/5’ splice sites (A3SS, A5SS)
- Mutually exclusive exons (MXE)
- Tandem 3’ UTRs (TandemUTR)
- Retained introns (RI)
- Alternative first exons (AFE)
- Alternative last exons (ALE)
SplicSeq软件可以拿到7种可变剪切形式信息:
- 可变受体位点(AA)
- 可变供体位点(AD)
- 可变启动子(AP)
- 可变终止子(AT)
- 内含子保留(RI)
- 外显子跳跃(ES)
- 外显子互斥(ME)
下一期我们再更新邢老师实验室出品的rmarts软件的4.1版本使用说明书,敬请期待哦!