最近看到某公司宣传他们的科研服务产品,是miRNA的芯片,而且文章居然是2011发表的,那个时候我还不知道生信是啥子。(我一直以为自己足够老了)
该研究使用的是 CapitalBio 平台 (CapitalBio 公司) 芯片,非常清晰的研究思路;
- 60+88个肺鳞癌病人肿瘤组织和癌旁的miRNA芯片表达矩阵,数据集在:GSE15008
- 芯片是 CapitalBio 平台 (CapitalBio Corp.) ,包含 924 mature mam- malian microRNA probes (including 677 human micro- RNA sequences),现在miRBase数据库收录了1917条pre-miRNA(前体),以及2656条成熟的miRNAs。(见:http://www.mirbase.org/ )
- 使用主成分分析和支持向量机建模,拿到 minimal 5- element classifier (hsa-miR-210, hsa-miR-182, hsa-miR- 486-5p, hsa-miR-30a, and hsa-miR-140-3p) 可以很好的区分normal和tumor。
- 生存分析发现:high expression of hsa-miR-31 was associated with poor survival
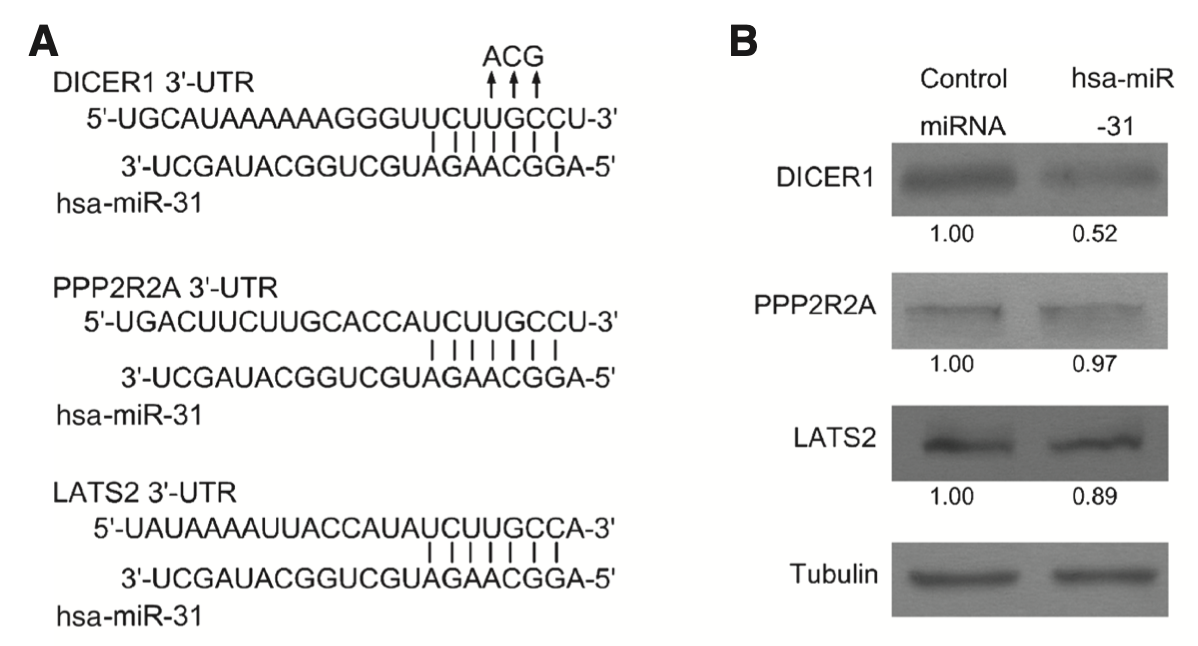
- 分析hsa-miR-31 的靶基因,并且实验验证其中3个:DICER1, PPP2R2A, and LATS2,最后定位到DICER1 30-UTR
我以前在生信技能树分享了几个miRNA的靶向基因的查询工具,分别是:
大家可以尝试看看hsa-miR-31 的靶基因,是否有这3个基因,示意图如下:
学徒作业
大家可以去tcga数据库下载肺鳞癌的miRNA芯片或者测序数据,走同样的诊断建模流程,看看得到的miRNA是否作者的5个miRNA有交叉,这里面变量很多:
- 首先,两个队列的人群地域差异
- 其次,miRNA芯片和miRNA测序技术差异
- 还有,肿瘤组织和癌旁配对问题,两个组数据量问题
对大家来说,比较难的地方就是使用主成分分析和支持向量机建模。可以参考我的4个小时TCGA肿瘤数据库知识图谱视频教程,其中总共使用了四种算法构建模型:
- cox(可做单因素和多因素)
TCGA的cox模型构建和风险森林图 - lasso回归
用lasso回归构建生存模型+ROC曲线绘制 - 随机森林
听起来很霸气用起来并不难的随机森林 - 支持向量机
听起来很霸气用起来并不难的 支持向量机
不管用了那种算法,核心都只是几句代码而已。
点击下面的阅读原文,马上就可以学习起来啦~
https://www.bilibili.com/video/BV1db411L7GX
文末友情宣传
强烈建议你推荐我们生信技能树给身边的博士后以及年轻生物学PI,帮助他们多一点数据认知,让科研更上一个台阶:
- 生信爆款入门-全球听(买一得五)(第4期),你的生物信息学入门课
- 数据挖掘第2期(两天变三周,实力加量),医学生/临床医师首选技能提高课
- 生信技能树的2019年终总结 ,你的生物信息学成长宝藏
- 2020学习主旋律,B站74小时免费教学视频为你领路,还等什么,看啊!!!