最近有粉丝表示,跟着我的最新lncRNA视频学习后,走她自己感兴趣的项目,惊奇的发现,GAPDH这个基因并没有表达量,怀疑是我的芯片探针ID注释有问题。
我勒个去,火都烧到家门口了,必须迎战。
首先,我花了几分钟查看了该文章和数据集
- https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE77013
- A long noncoding RNA signature for ulcerative colitis identifies IFNG-AS1 as an enhancer of inflammation. Am J Physiol Gastrointest Liver Physiol 2016 Sep 1;311(3):G446-57. PMID: 27492330
该研究总共是纳入50个人,但实际上19个做了芯片数据,分别是: - 7 control patients,
- 8 UC-active (UCA),
- 4 UC-inactive (UCI)
因为是 Human LncRNA Array v3.0 (8 × 60 K, Arraystar). 芯片,所以直接使用 GeneSpring GX v12.1软件 (Agilent Technologies).
剩下的31人是:16 control patients and 15 UC patients
3次差异分析,都使用同样的的阈值: (>2-fold and P < 0.05) - UCA and control, 1,931个差异的lncRNA
- UCA and UCI colonic tissues, 1,361个差异的lncRNA
- UCI relative to control tissues,287 个差异的lncRNA
挑选 RP11-81H14.2 and IFNG-AS1 去实验验证, 有意思的是,仅仅是 IFNG-AS1验证成功。
有意思的是最后还根据 IFNG-AS1的表达量把15个 UC and control samples分成高低表达组进行差异分析。
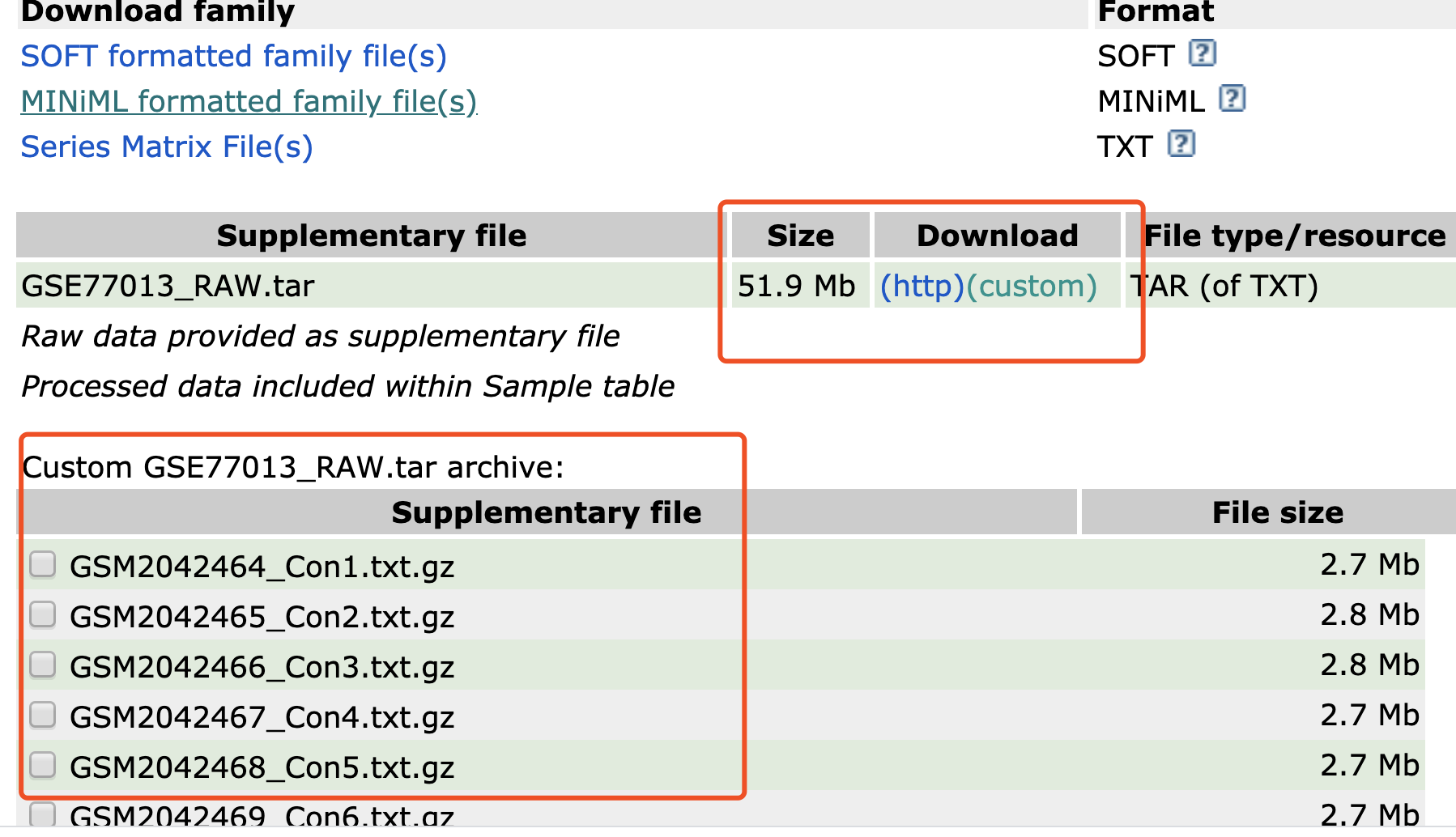
不过,这些并不重要,现在的问题是,为什么作者上传的表达矩阵仅1万多个探针,而该芯片平台明明是有近6万探针,文章自己写说了:The lncRNA expression profiling was performed using the Arraystar lncRNA microarray V3.0, which consists of 30,600 lncRNAs. - 芯片平台信息:https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GPL16956
最合理的猜测是,该芯片的大部分探针不满足要求,被作者在数据处理的过程中删除了,但是我浏览全文,仅仅是提到:After quantile normalization of the raw data, lncRNAs and mRNAs that at least 19 samples have flags in Present or Marginal (“All Targets Value”) were chosen for further data analysis.

我这里使用R语言下载的表达矩阵:library(GEOquery) gset <- getGEO('GSE77013', destdir=".",getGPL = F)#下载很慢 #https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE77013 ## 获取ExpressionSet对象,包括的表达矩阵和分组信息 exprSet=exprs(gset[[1]]) #a现在是一个对象,取a这个对象通过看说明书知道要用exprs这个函数 dim(exprSet)#看一下dat这个矩阵的维度 exprSet[1:4,1:4]检查了一下,的确作者上传的表达矩阵里面的探针就一万多个,那么缺一些基因就很正常了。
我觉得agilent的芯片,就是一个大坑,不仅仅是ID转换的问题。大部分做了芯片研究的都是实验出身科研人员,也不可能去搞清楚芯片原理,仅仅是委托公司处理。
如果我们想搞清楚中间问题所在,可能是得去看看这些原始数据。
文末友情宣传
强烈建议你推荐我们生信技能树给身边的博士后以及年轻生物学PI,帮助他们多一点数据认知,让科研更上一个台阶:
- 生信爆款入门-全球听(买一得五)(第4期),你的生物信息学入门课
- 数据挖掘第2期(两天变三周,实力加量),医学生/临床医师首选技能提高课
- 生信技能树的2019年终总结 ,你的生物信息学成长宝藏
- 2020学习主旋律,B站74小时免费教学视频为你领路,还等什么,看啊!!!