我写了部分常见的TCGA数据库用法:
- TCGA的28篇教程-免疫全景图
- TCGA的28篇教程-指定癌症查看感兴趣基因的表达量
- TCGA的28篇教程-对TCGA数据库的任意癌症中任意基因做生存分析
- TCGA的28篇教程-风险因子关联图-一个价值1000但是迟到的答案
- TCGA的28篇教程-数据挖掘三板斧之ceRNA
- TCGA的28篇教程-所有癌症的突变全景图
- TCGA的28篇教程-早期泛癌研究
- TCGA的28篇教程-CNV全攻略
- TCGA的28篇教程-GTEx数据库-TCGA数据挖掘的好帮手
因为时间关系,这28篇教程可能还得再推迟半年!不过最近看了很多文献,有一些关于TCGA数据库的应用值得推荐,比如文章是:TRIB3 supports breast cancer stemness by suppressing FOXO1 degradation and enhancing SOX2 transcription. 发表在:Nat Commun 2019 Dec 16;10(1):5720. PMID: 31844113 就提到了根据感兴趣的基因的mRNA表达量来把TCGA数据库的某个癌症的全部病人分组,比如取高表达的前10%和低表达的10%病人,然后两个分组进行差异分析,这样的话每个基因都会有一个变化倍数,这个数值就可以把基因排序啦。排序后的基因列表,毫无疑问就可以进行GSEA分析咯。
mRNA表达量高低分组病人
原文描述的方法是:

正文描述比较简单,而且是一回事:

根据生物学背景挑选需要检验的基因集
GSEA分析,除了需要排序好的基因列表,还需要一个待检验的基因集,如果大家感兴趣GSEA分析原理和用法,看合辑:
文章使用的是java软件做gsea分析,出图是:
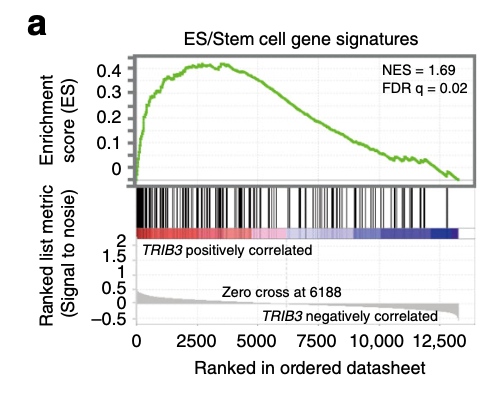
很好描述,从TCGA数据库,下载了BRCA病人1215个的mRNA表达矩阵,然后根据TRIB3这个基因表达量分组,差异分析后GSEA,看到他们感兴趣的ES stem cell gene signatures基因集是显著富集的。
GSEA demonstrating the enrichment of gene sets related to ES stem cell gene signatures in the ranked gene list of the top 10th percentile (n = 121) vs. the bottom 10th percentile (n = 121) of breast cancer patients expressing TRIB3 from the TCGA database (n = 1215). NES, normalized enrichment score. FDR, false discovery rate.
首先需要学会下载TCGA指定数据
我挑选了部分,写了6个数据下载系列教程:
- TCGA的28篇教程- 使用R语言的cgdsr包获取TCGA数据(cBioPortal)
- TCGA的28篇教程- 使用R语言的RTCGA包获取TCGA数据 (离线打包版本)
- TCGA的28篇教程-使用R语言的RTCGAToolbox包获取TCGA数据(FireBrowse portal)
- TCGA的28篇教程- 批量下载TCGA所有数据 ( UCSC的 XENA)
- TCGA的28篇教程-数据下载就到此为止吧
- TCGA的28篇教程-整理GDC下载的xml格式的临床资料
然后需要有自己感兴趣的基因和基因集,然后你也想做同样的探索,但是代码能力不行,可以委托我们哈!
- TCGA的28篇教程-免疫全景图
- TCGA的28篇教程-指定癌症查看感兴趣基因的表达量
- TCGA的28篇教程-对TCGA数据库的任意癌症中任意基因做生存分析
- TCGA的28篇教程-风险因子关联图-一个价值1000但是迟到的答案
- TCGA的28篇教程-数据挖掘三板斧之ceRNA
- TCGA的28篇教程-所有癌症的突变全景图
- TCGA的28篇教程-早期泛癌研究
- TCGA的28篇教程-CNV全攻略
- TCGA的28篇教程-GTEx数据库-TCGA数据挖掘的好帮手
数据分析免费做
2019-2020的春节假期我们生信技能树推出一系列生物信息学数据分析免费做活动,先到先得哈,我们推文里面提到的数据分析环节都是我非常有经验的, 对我来说是举手之劳,希望可以帮助到你!
- 转录组数据分析的4个维度认识(数据分析继续免费哦) RNA-seq数据的2个分组差异分析,热图,PCA图,火山图等等
- 根据感兴趣基因看肝癌免疫微环境的T细胞亚群差异 条形图或者箱线图
- 查看感兴趣基因的甲基化水平和RNA表达水平(数据分析免费做)相关性 散点图或者箱线图
- 我不相信kmplot这个网页工具的结果(生存分析免费做)
发送数据分析要求,以及简短的项目描述到我的邮箱 jmzeng1314@163.com
邮件正文最好是加上你是啥时候认识生信技能树的哦,或者其它一些寒暄的话,自我介绍也行。主要是考虑到可能想免费分析数据的朋友很多,所以会根据你的来信,我主观判定一个优先级哦。目前我有20多个愿意长期在我的指导下进行数据探索的学徒,等我的团队扩大到200人,我们应该是可以做到数据分析全部免费,敬请期待哈!