数据是一切的开始,万事开头难哦!
前面我们介绍了一些背景知识,主要是理解什么是DNA甲基化,为什么要检测它,以及芯片和测序两个方向的DNA甲基化检测技术。具体介绍在:甲基化的一些基础知识,也了解了甲基化芯片的一般分析流程 。
既然要开始甲基化芯片数据挖掘实战,那么首先要有数据咯!需要区别的是甲基化芯片样本的idat原始文件,以及甲基化信号值矩阵。
如果你有mRNA表达芯片数据分析经验,可以类比于 affymetrix的 cel 文件和基因表达矩阵。
GEO官网直接下载
比如 https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE68777
需要自行阅读 解读GEO数据存放规律及下载,一文就够 教程,那个raw.tar是把该项目的每个样本的idat原始文件打包好了。
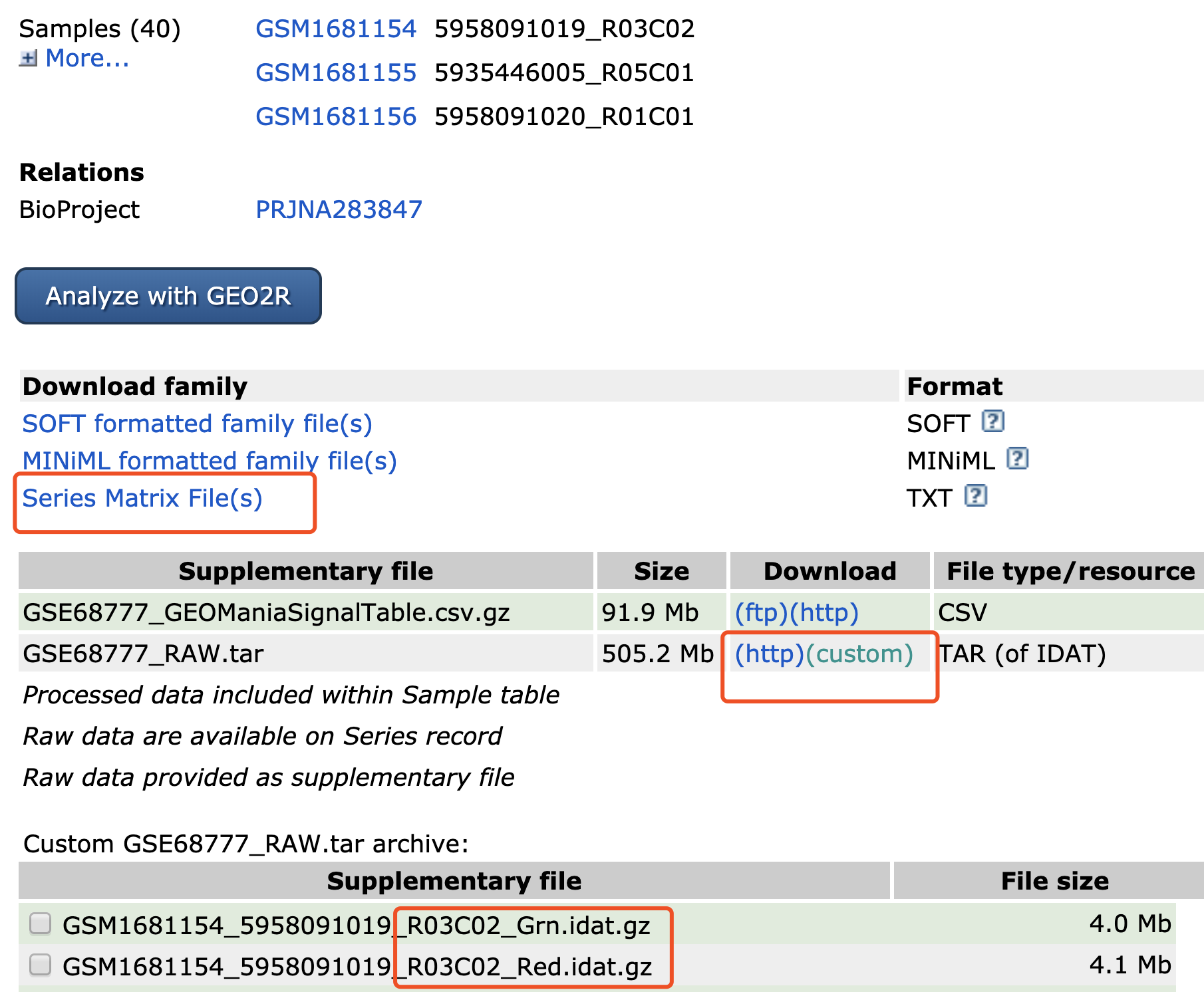
下载得到的 GSE68777_RAW.tar 解压后也可以使用minfi读取,然后走甲基化芯片流程哈。
使用 GEOquery包下载
可以使用 GEOquery包getGEOSuppFiles函数的下载直接下载甲基化芯片的idat原始文件,然后自己minfi包的read.metharray.exp函数处理它们。
但是,强烈建议不要使用GEOquery包getGEOSuppFiles函数,下面的代码没太大意思。
library(GEOquery)
getGEOSuppFiles("GSE68777")
# 相当于直接下载ftp://ftp.ncbi.nlm.nih.gov/geo/series/GSE68nnn/GSE68777/suppl/GSE68777_RAW.tar
untar("GSE68777/GSE68777_RAW.tar", exdir = "GSE68777/idat")
head(list.files("GSE68777/idat", pattern = "idat"))
# minfi 无法读取压缩的idat文件,所以需要解压
idatFiles <- list.files("GSE68777/idat", pattern = "idat.gz$", full = TRUE)
sapply(idatFiles, gunzip, overwrite = TRUE)
library("minfi")
rgSet <- read.metharray.exp("GSE68777/idat")
rgSet
pData(rgSet)
save(rgSet,file = 'GSE68777_minfi_rgSet.Rdata')
# 这个数据集也是有 https://ftp.ncbi.nlm.nih.gov/geo/series/GSE68nnn/GSE68777/matrix/GSE68777_series_matrix.txt.gz
# 大家可以比较两个读取idata文件走minfi流程拿到的甲基化信号值矩阵,与作者上传的有什么区别
也可以直接下载甲基化信号值矩阵,取决于你是否相信作者对芯片原始数据的处理。代码很简单
如下:
require(GEOquery)
require(Biobase)
GSE80559 <- getGEO("GSE80559")
beta.m <- exprs(GSE80559[[1]])
再次强调,这个方法适用于数据集的研究者处理好了idat芯片原始数据,而且处理的格式符合要求哈。大概率上,你还是得自己去下载idat芯片原始数据走minfi流程的。
ftp网页下载
如下图,点击 Series Matrix File(s) 可以直达ftp网页,在里面为所欲为哈,
- ftp://ftp.ncbi.nlm.nih.gov/geo/series/GSE68nnn/GSE68777
可以看到,geo页面显示的文件,其实都在这个ftp里面

拿到那个raw.tar下载链接 - ftp://ftp.ncbi.nlm.nih.gov/geo/series/GSE68nnn/GSE68777/suppl/GSE68777_RAW.tar
就可以发给海外的朋友帮忙下载后,通过云盘等手段传递给你哈!
这个 GSE68777_RAW.tar 解压后也可以使用minfi读取,然后走甲基化芯片流程哈。等价于library(GEOquery) getGEOSuppFiles("GSE68777")这样的代码,再次强调,这个代码在中国大陆基本上下载失败的哦。
并不是说raw里面都是芯片的idat文件
并不是所有使用了甲基化芯片数据的研究者都非常理解它,所以不一定会上传idat文件,那就有点麻烦了,如下所示的 GSE58651 的RAW.tar 里面就不是该数据集的各个样本的idat文件:
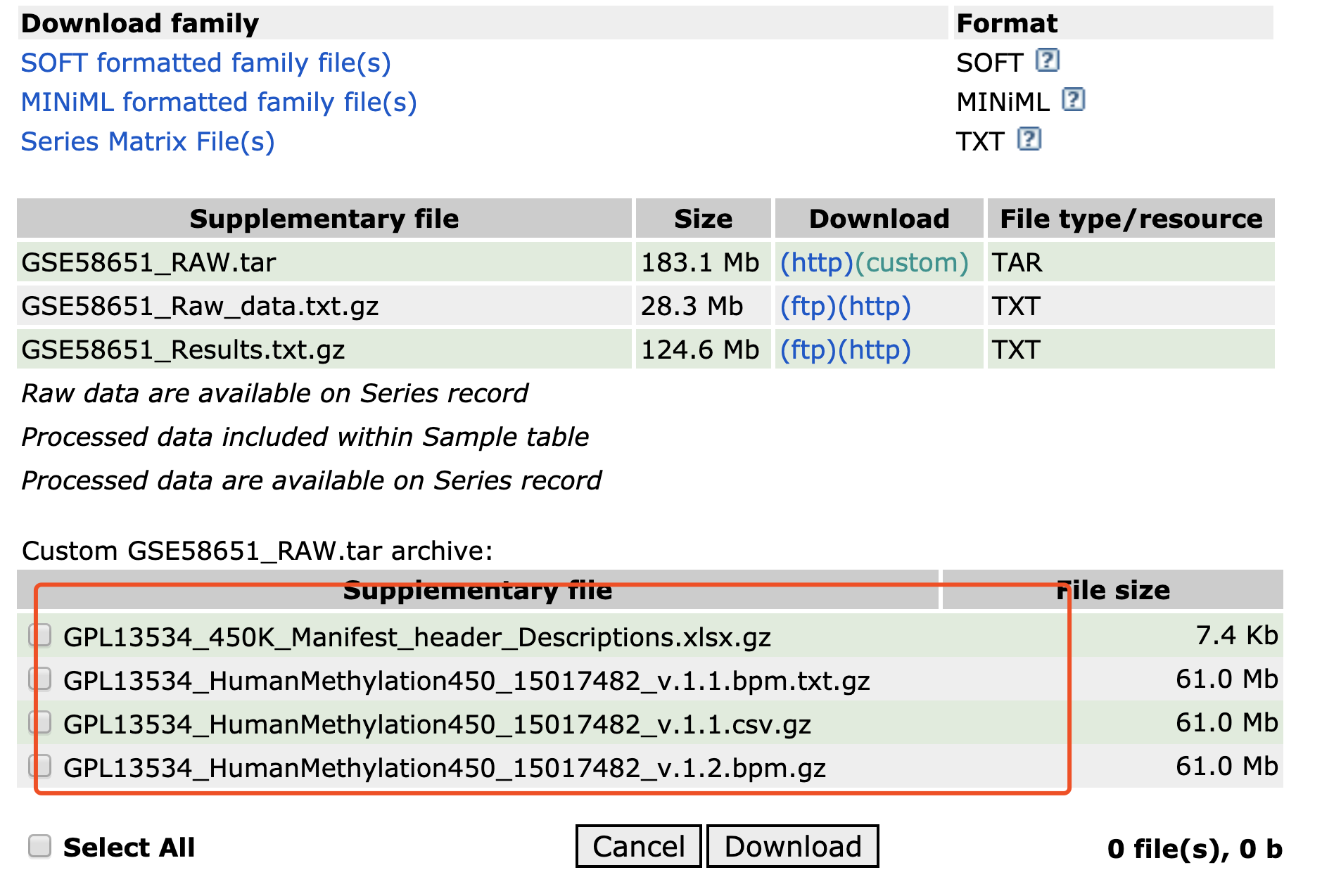
再次强调,这个时候的的RAW.tar 里面就不是该数据集的各个样本的idat文件,仅仅是这个甲基化芯片平台的注释信息。
反正没有了idat芯片原始文件,你需要自己想办法去理解另外两个文件,争取可以拿到能被挖掘的甲基化信号值矩阵。
这样的数据集还不在少数,再比如:https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE75679
这样的情况下,下载RAW.tar 文件是没有用的,需要下载signal_intensities文件,然后去理解,作者到底当年是如何处理这些数据的。
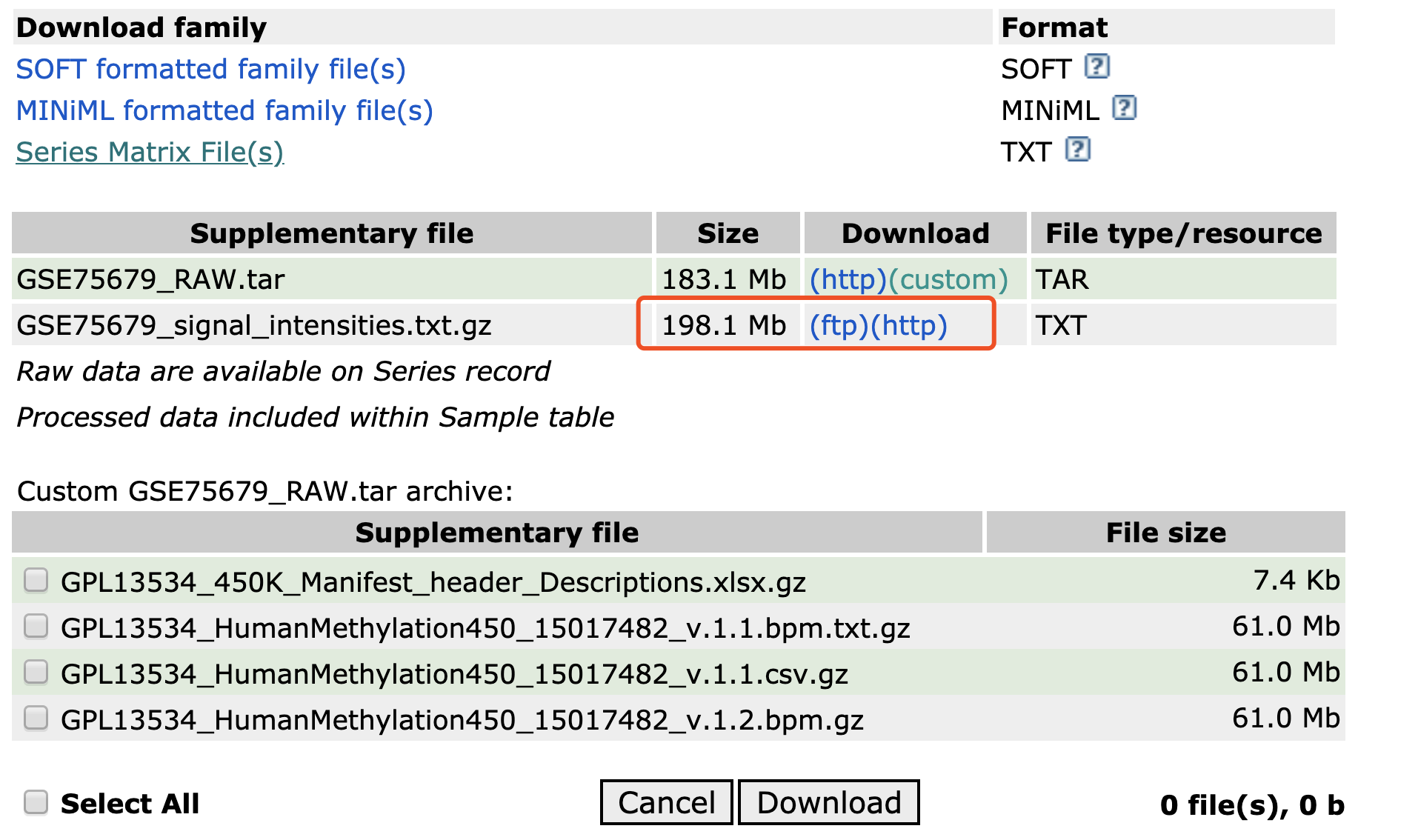
下面两个文件都需要探索: - ftp://ftp.ncbi.nlm.nih.gov/geo/series/GSE75nnn/GSE75679/matrix/GSE75679_series_matrix.txt.gz
- ftp://ftp.ncbi.nlm.nih.gov/geo/series/GSE75nnn/GSE75679/suppl/GSE75679_signal_intensities.txt.gz
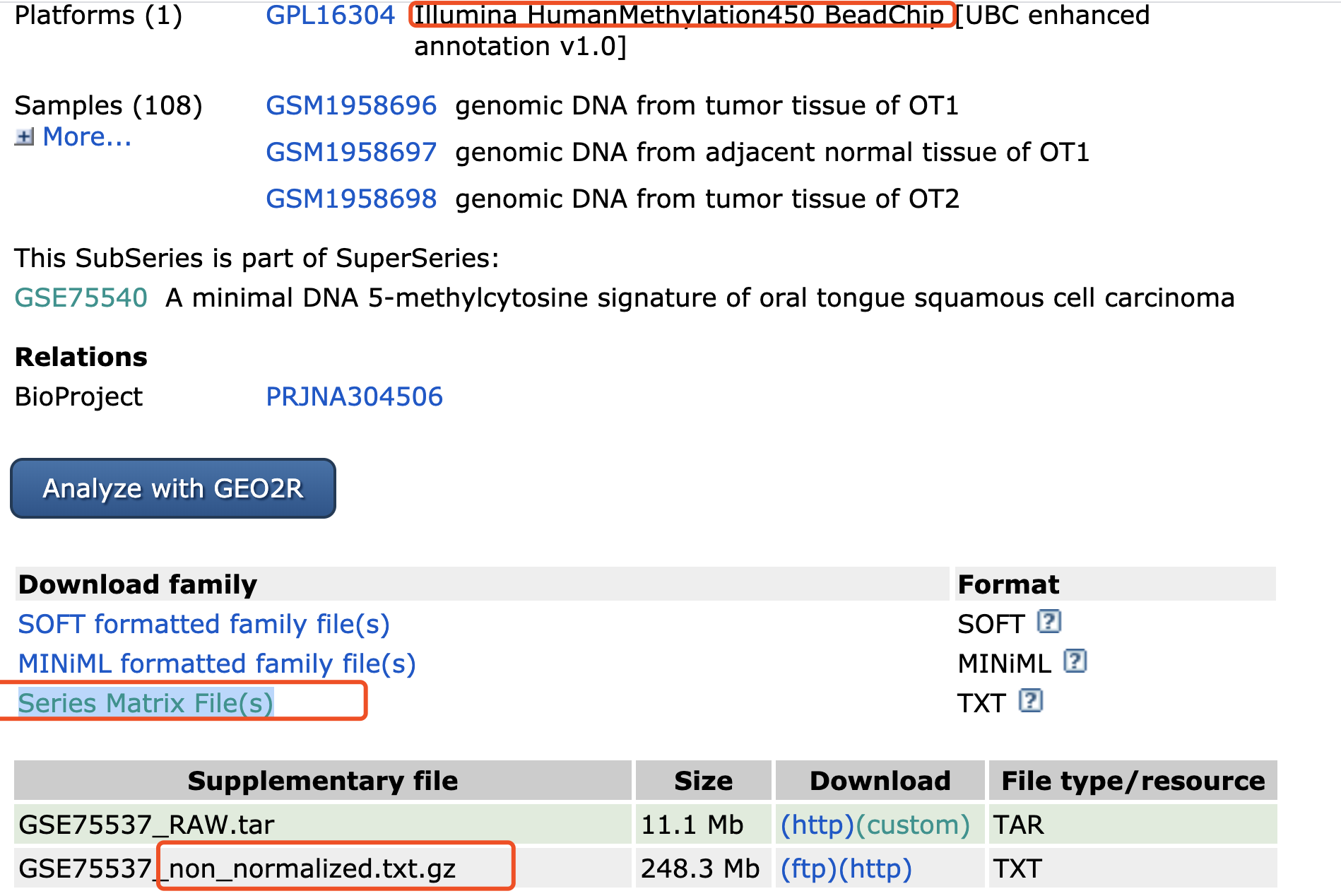
还有一些是 non_normalized.txt.gz ,比如: https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE75537
也需要去仔细学习这个数据集作者写出来的处理方法哈
给大家一个作业
理解 https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE61441 数据集页面给出的多种数据形式。该数据集的文章发表在 Nat Commun 2015 Oct 30;6:8699. PMID: 26515236
The Illumina Infinium 450k Human DNA methylation Beadchip was used to obtain DNA methylation profiles across approximately 450,000 CpGs in tumor and adjacent normal kidney tissue samples from ccRCC patients. Samples included 46 paired fresh frozen ccRCC tumor and adjacent normal kidney tissues.
有下面这些文件:Supplementary file Size Download File type/resource GSE61441_Normalized_data.csv.gz 102.1 Mb (ftp)(http) CSV GSE61441_RAW.tar 943.2 Mb (http)(custom) TAR (of IDAT) GSE61441_Unmethylated_and_methylated_signal_intensities.csv.gz 219.2 Mb (ftp)(http) CSV还有 Series Matrix File(s) 文件,都可以下载并且探索一下。
总结一下
如果你相信作者对他自己的甲基化芯片数据处理,就可以直接使用其 _series_matrix.txt.gz 存储的甲基化信号矩阵。
如果你不相信作者,就下载他上传的idat芯片原始数据,然后自己走minfi或者champ流程,自己拿甲基化信号矩阵走下游分析。
但是呢,在中国大陆,很大概率上,idat芯片原始数据或者甲基化信号矩阵,你都会下载失败!