一直听说Functional Annotation Tool DAVID Bioinformatics Resources 6.8, NIAID/NIH 是可以做GO/KEGG数据库注释的,只需要用户上传自己拿的的基因集就可以,大大的方便了生物学家对数据库的使用。
因为自己都是在R里面批量做,所以没有机会使用DAVID。最近反馈结果给合作的博士后才发现,原来二者是有差异的。(因为合作的博士也不相信我的R分析结果,要用网页工具验证一波)
现在让我们一起来探索要的差异来源可能是什么吧。就拿最近的一个项目的Mus musculus的一个基因集来举例说明:
可以看到,我们输入了小鼠的454个基因,然后DAVID网页数据库(操作方法就不赘述,基本上就是鼠标点点点,下一步即可)结果如下:
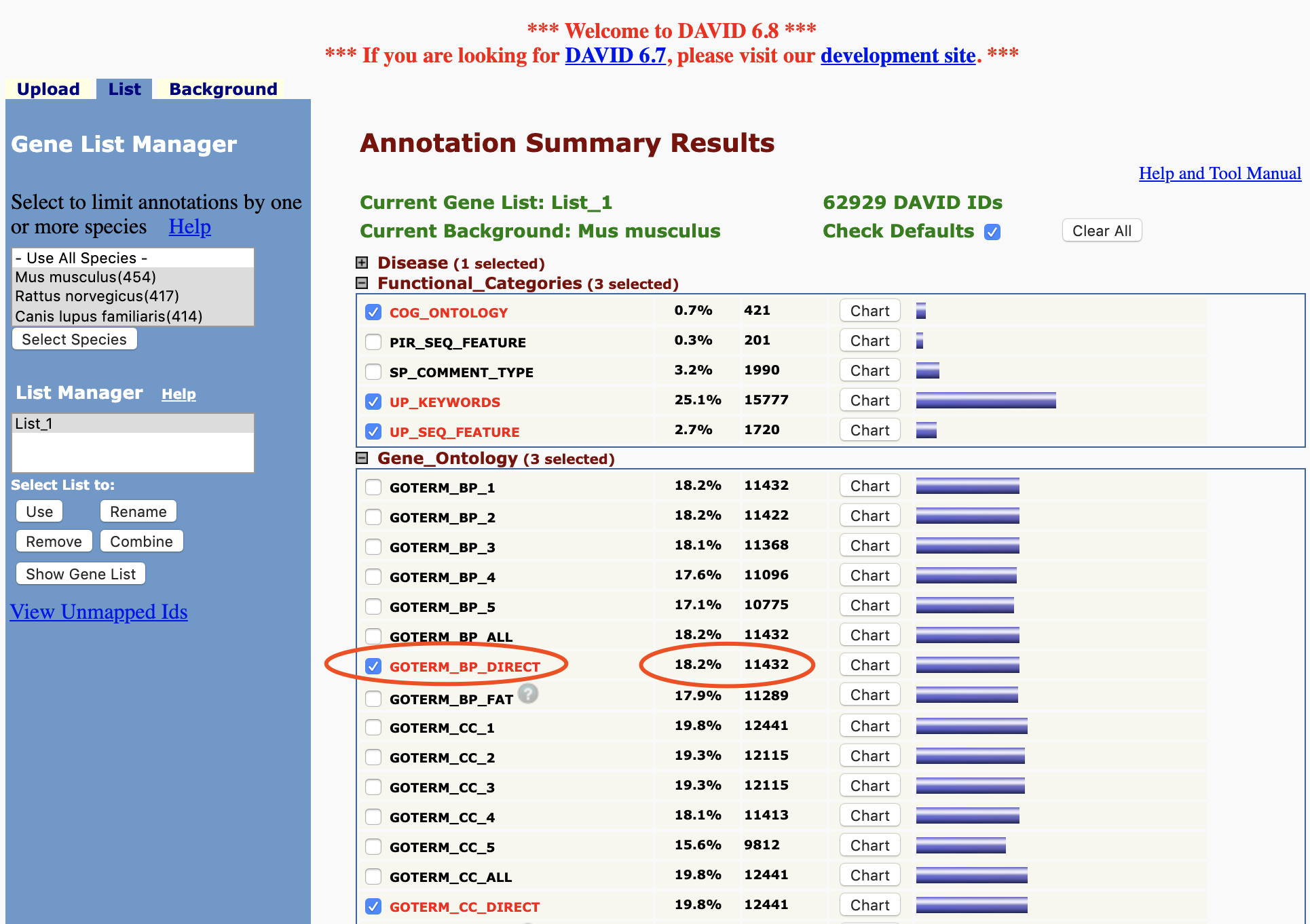
同样的,因为GO数据库比较复杂,即使是仅仅关注BP,基因集也很多,我们就看前几名即可:
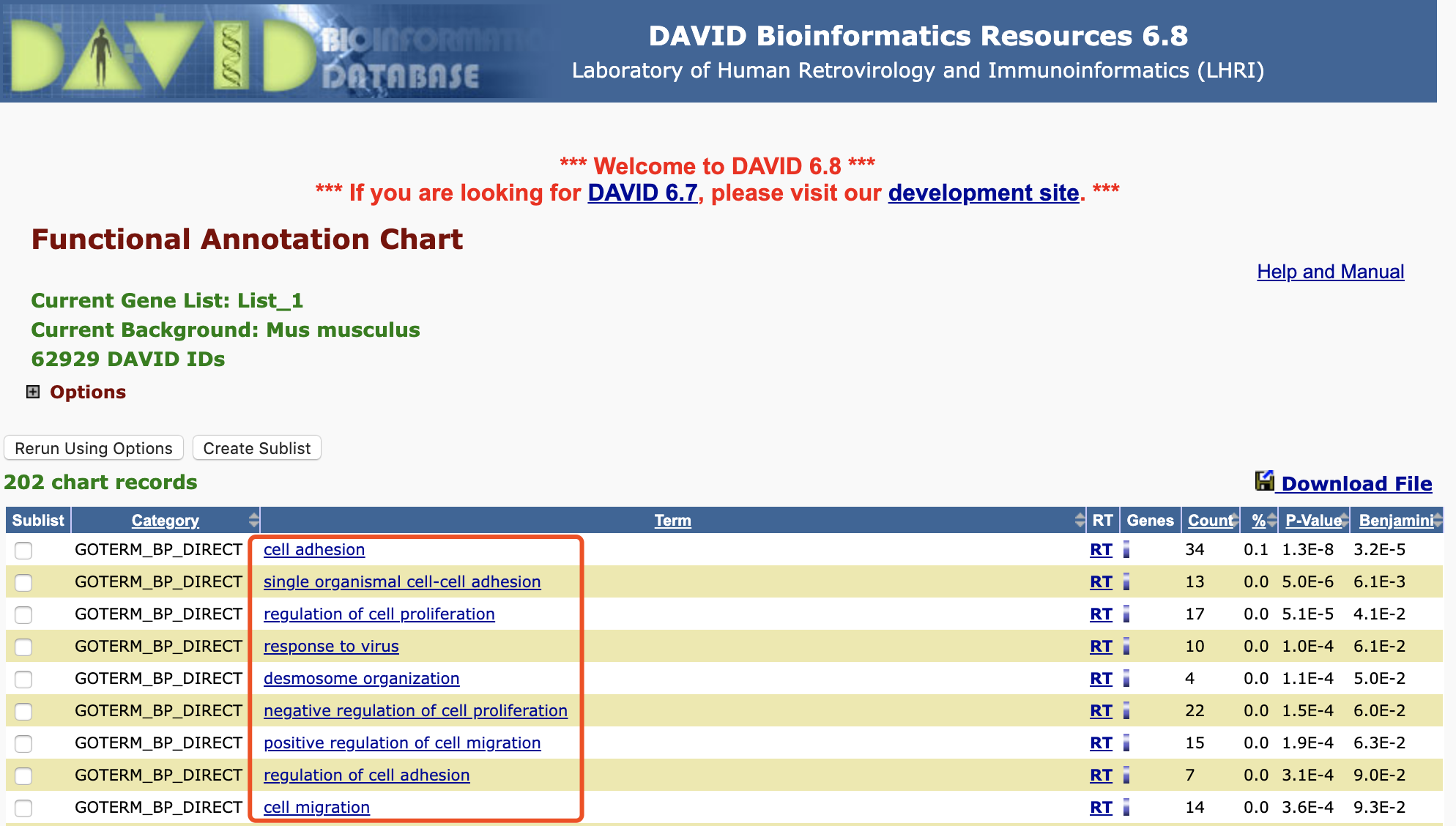
先比较第一个,可以看到DAVID数据库里面显示着有34个基因属于 cell adhesion 基因集,如下所示:
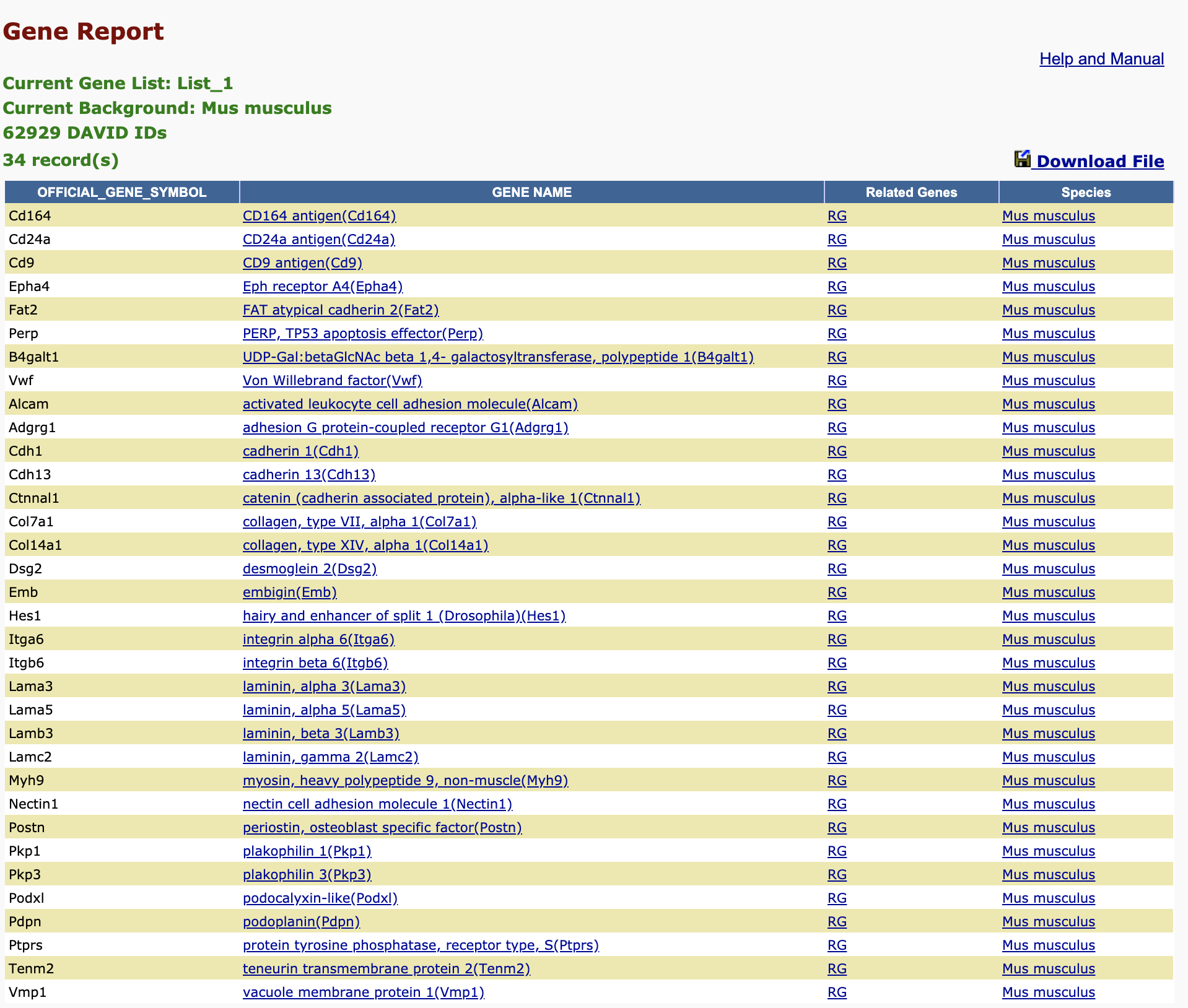
但是走R的注释代码,得到的结果差异非常大。首先,这个cell adhesion 基因集并不显著!这个是非常致命的冲突了,一般来说,如果仅仅是基因集的数量差异,我们可以认为是数据库版本问题。
那我们仔细看看 cell adhesion 相关的基因集,是不是有ID和名字的冲突,在我的R结果里面可以搜索到的,如下:

而在DAVID网页数据库结果里面可以看到的是:
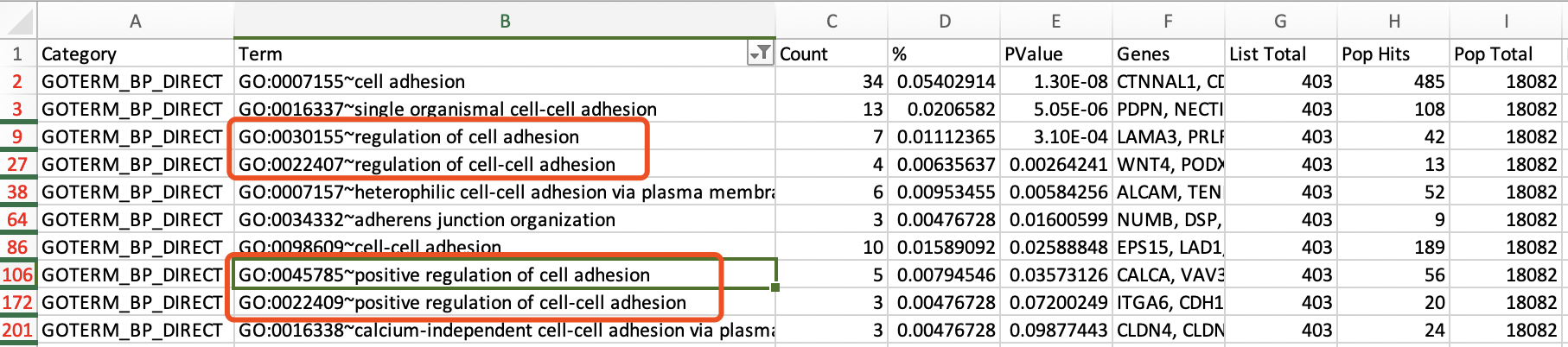
看起来,能够拿的出来进行比较的只有:GO:0045785 名字是:positive regulation of cell adhesion,它在DAVID网页数据库被记录56个基因,然后被富集到了5个,所以是显著的。而在R的分析里面,它被记录有410个基因,被富集到的有21个。
那么这个条目,GO:0045785 名字是:positive regulation of cell adhesion 到底应该是多少个基因呢?很简单,谷歌搜索就找到了官网:
http://www.informatics.jax.org/vocab/gene_ontology/GO:0045785
的确是415个基因,如果R语言包注释结果是正确的,那么问题来了,为什么DAVID网页工具的GO数据库注释结果会少那么多?
DAVID网页工具的KEGG数据库其实也是在更新
这个时候,可能很多人都会说,是DAVID网页工具不更新了,其实就算是说它不更新,也只可能是KEGG的延迟而已,并不是说它一无是处,而且它的确是更新了的。
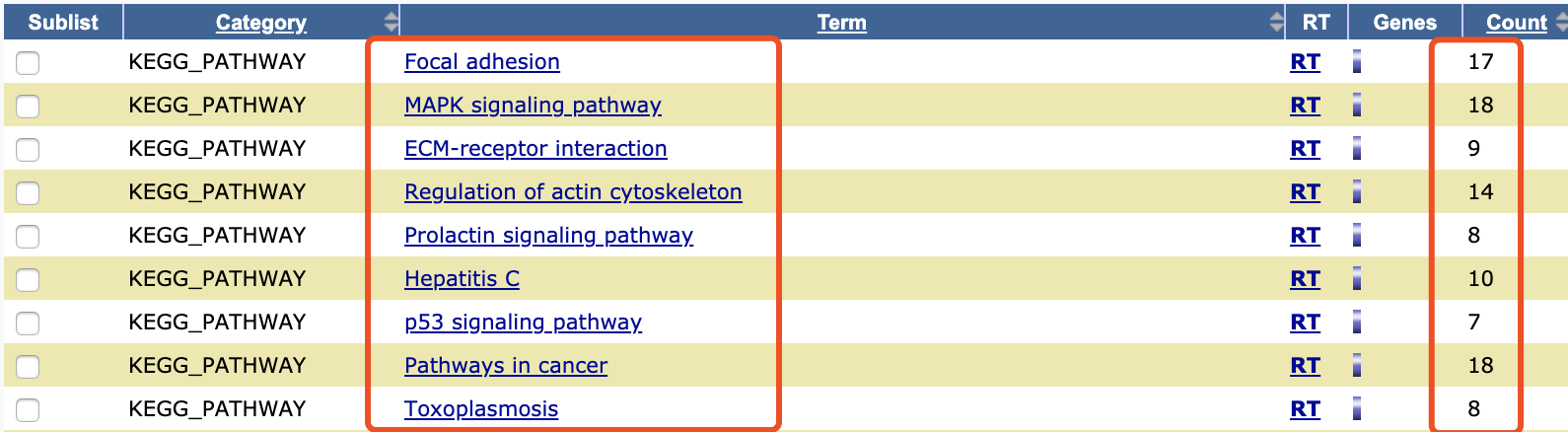
在R里面是:

可以看到,具体的每个基因集富集到的基因数量是没有出入的,很吻合。
那么问题到底出在哪里了呢?
其实是DIRECT的问题
后来我们仔细瞅了瞅,发现问题出在DIRECT上面,也就是说GO:0045785 名字是:positive regulation of cell adhesion,在官网的确是415个基因,在R里面也是这么多,但是呢,在DAVID里面,数量只剩下56了。其它通路也是如此,都是少很多。
我没有搜索到DIRECT的相关权威的英文资料,所以自己摸索了一下。大概的意思应该是,如果一个GO通路有100个基因,但是呢,这个GO通路有几个子节点,那么它的100个基因里面就会必然有很多基因其实也属于它的那几个子节点,也会有一些基因不属于它的任何的子节点,这样的基因很少,就是DIRECT的基因啦。
这就是为什么GO:0045785 名字是:positive regulation of cell adhesion,在官网的确是415个基因,在R里面也是这么多,但是呢,在DAVID里面,数量只剩下56了。因为这个DAVID里面仅仅是看DIRECT的基因。