看到朋友圈很多人转发一篇研究,至少4个公众号跟踪报导分享了,我也简单阅读了一下,说说自己的感受哈!
- Cell重磅:肿瘤微生物断生死,粪菌移植逆天改命,缩小肿瘤、活化免疫、抗癌续命
- 肠道菌群又一发现!《Cell》重磅揭示,胰腺癌患者生存期的长短由肠道内细菌决定,冥冥中并非天注定
- Cell | 张煜博士等破解癌症之王——胰腺癌“长寿”人群的秘密
- Cell重磅证据:胰腺癌患者活2年还是10年,肿瘤中的细菌或是决定因素!
我觉得他们的解读都过分强调了菌群多样性是肿瘤病人生存时间的决定因素。胰腺癌是致死率最高的恶性肿瘤之一,根治性手术切除仍是目前胰腺癌患者唯一有可能获得长期生存的治疗手段。目前由于缺乏筛查早期胰腺癌的有效手段,因此80%胰腺癌患者确诊时已属中、晚期,肿瘤已经远处转移,失去了根治性手术切除的机会,预后很差,即使患者可以接受手术切除,术后复发率也很高,中位总生存期在24-30个月之间。尽管如此,手术后仍有一小部分患者存活期超过5年。那么是什么决定了胰腺癌患者生存期的长短呢?目前科学家一般认为,肿瘤基因组格局可以预测患者总体生存率和对治疗的反应。
但是发表于August 2019,在CELL杂志的研究https://doi.org/10.1016/j.cell.2019.07.008,肿瘤微生物多样性和组成能影响胰腺癌预后!
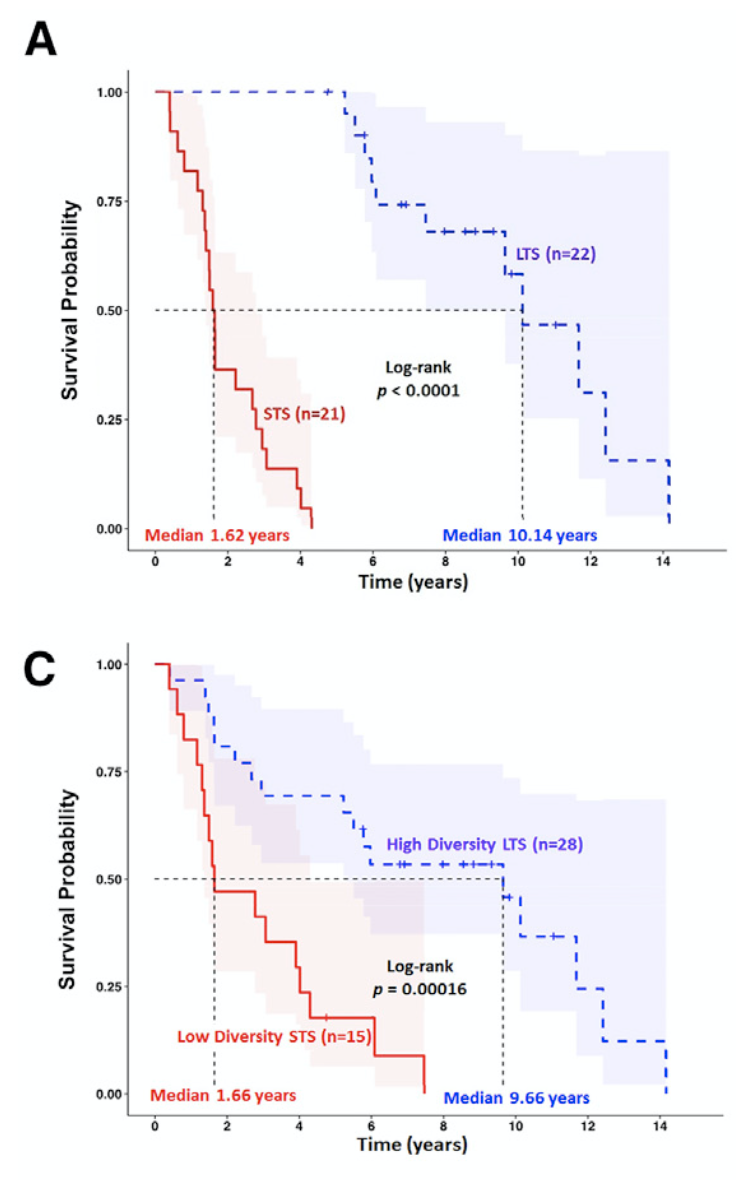
当然,虽然我承认这个事实,就是生存长短的两个病人组,菌群差异很大,但是我并不认为这个是他们生存差异的原因。胰腺癌短期生存者(short-term survival,STS)和长期幸存者(long-term survival,LTS),两个完全不同的组别,可以分析的差异太多了。围绕着中心法则的各种NGS技术都可以来一波,都是有差异。凭什么他们做了菌群,就说菌群是因???(36 LTS and 32 STS)的病人队列
Bacterial DNA was extracted from 68 surgically resected PDAC tumor (36 LTS and 32 STS), and taxonomic profiling via 16S rRNA gene sequencing was performed.
生存时间区分 - more than 5 years after surgery, or long-term survivors (LTS) (median survival 10.1 years),
- less than 5 years after surgery (short-term survival [STS]; median survival 1.6 years)
来源于两个单位: - UT MD Anderson Cancer Center (MDACC)
- Johns Hopkins Hospital (JHH) in Baltimore, Maryland
很容易看到生存差异
16S rRNA 测序(扩增子测序)及数据分析
细菌rRNA(核糖体RNA)按沉降系数分为3种,分别为5S、16S、23S rRNA。16S rDNA只存在于所有细菌染色体基因中,真菌没有,病毒没有,高等动物更没有。
16S rDNA全长约1500bp,由V1-V9九个可变区和若干保守区组成。我们通常扩增的是其中的V3-V4区。尽管这段区域在不同物种间差异度更高,但不能保证将所有的细菌种类完全鉴定区分,这也是扩增子测序对鉴定出的微生物有分类水平限制的重要原因之一。
16S分析流程主要包括: - Hiseq/Miseq测序获得的Paired-end (PE) reads拼接成一条序列
- 对目标序列进行质控过滤
- 过滤后的序列与参考数据库作比对
- 去除嵌合体序列得到最终得优化序列
- 基于优化序列进行OTU聚类分析和物种分类注释
- 基于OTU聚类结果进行多样性指数分析
- 基于分类学信息进行物种结构分析和物种差异分析
文章描述如下:

LTS 和 STS 的菌群多样性差异
Alpha多样性是指一个特定区域或生态系统内的多样性。多样性指数是反映样本中微生物丰富度和均匀度的综合指标。Alpha多样性分析主要包括Shannon多样性指数、Simpson指数、Chao1丰富度指数等多样性指数的计算.
来源于两个不同单位的病人队列都显示同样的统计学分析结果,活得久的病人的菌群多样性好。
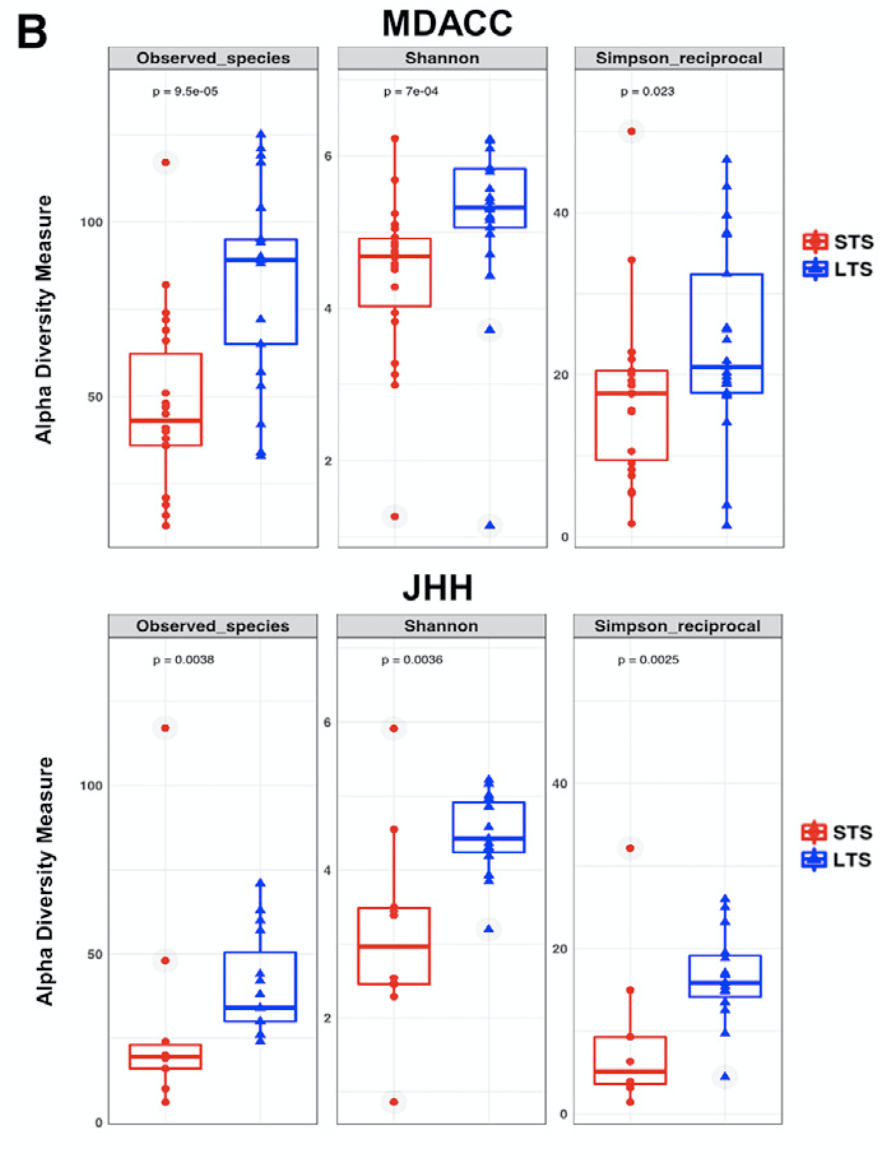
而且与其它临床特征并没有统计学显著:Importantly, we assessed for potential contributors to microbial diversity, including clinico-pathological features, body mass index, sex, smoking, neoadjuvant and adjuvant therapies, as well as antibiotics use, and were not able to find any significant association
3大主要差异菌群
生存好的病人组别里面Proteobacteria (Pseudoxanthomonas) and Actinobacteria (Saccharopolyspora and Streptomyces), 这3大菌群非常丰富,简单的差异分析即可,差异分析后的热图显示如下:
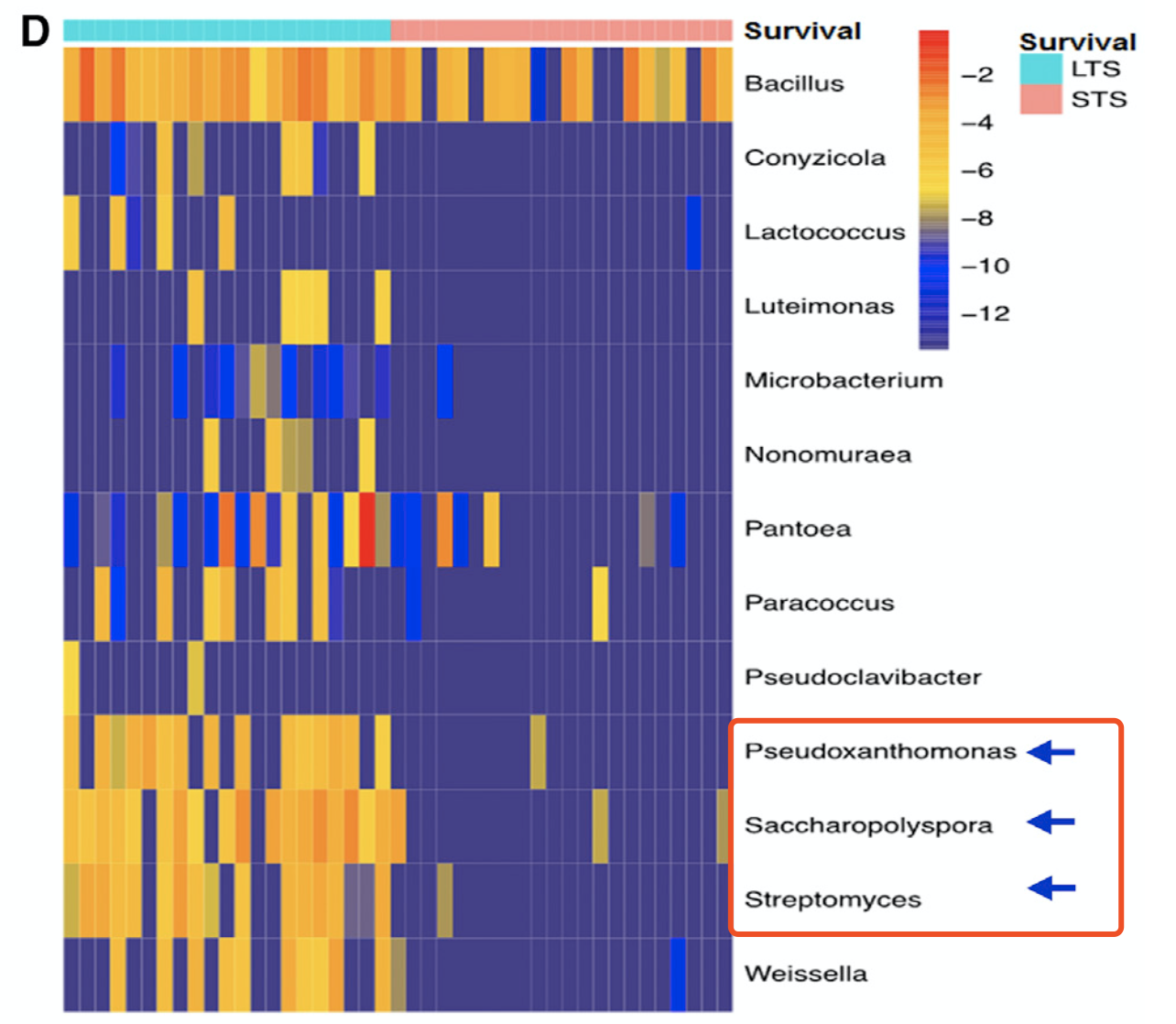
LTS组肿瘤在类水平上表现出AlphaProobacteria(α变形杆菌属),Sphingoobacteria(鞘脂杆菌)和Flavobacteria(黄杆菌)的优势。
LTS患者表现出对变形杆菌Proteobacteria(假黄单胞菌Pseudoxanthomonas)和Actinobacteria放线菌(Saccharopolyspora糖多孢菌和Streptomyces链霉菌)的富集,而在STS患者未检测到优势菌属。
然后对3大菌群独立分析
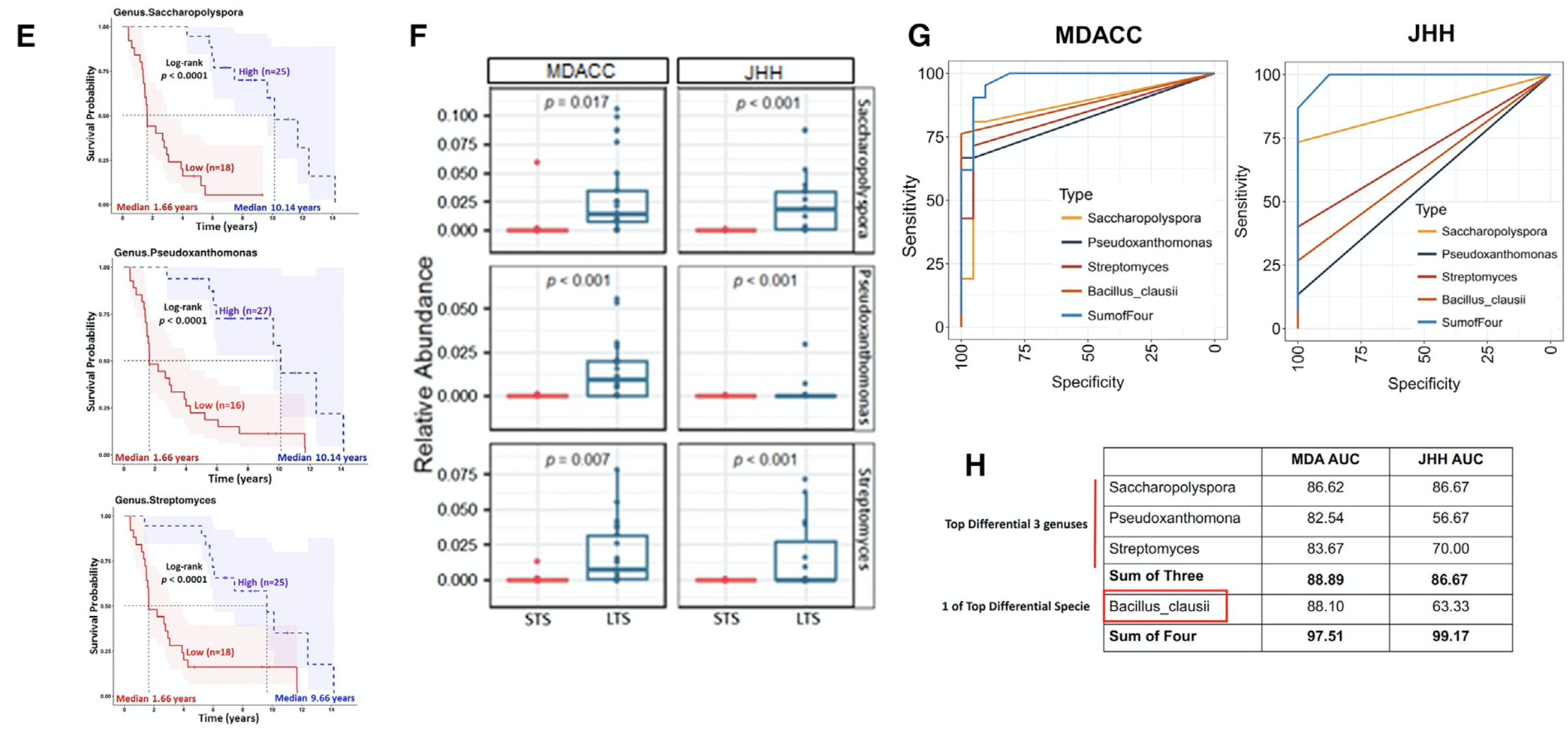
值得注意的是Bacillus是species的差异,其它3个都是genuses的差异哦。
最后定义一个:intra-tumoral microbiome signature (Pseudoxanthomonas-Streptomyces-Saccharopolyspora-Bacillus clausii) 这个是数据分析环节,值得大家仔细研读。肿瘤微生物群形成免疫应答促进T细胞活化
在进一步对于其中机制的研究中,研究者通过对长/短生存期病人肿瘤组织的immune profiling发现,长生存期患者的肿瘤内细胞毒性T细胞(CD3+CD8+)细胞的密度显著高于一般短生存期患者,提示肿瘤组织内特定的细菌菌群可以通过招募和激活细胞毒性T细胞改善抗肿瘤的免疫反应。
人-鼠粪便微生物菌群移植
在回答如何影响和干预肿瘤细菌菌群的问题上,研究者发现胰腺癌病人肿瘤内的菌群和肠道菌群有相当程度的重叠;不仅如此,通过人对鼠的粪便微生物移植实验,将经历胰腺癌Whipple手术病人的粪便微生物以灌胃的方法 (FMT) 移植到胰腺癌模型小鼠上,发现病人消化道细菌可以特异性地定植到小鼠胰腺肿瘤内。
接下来,研究者收集了长生存期和一般短生存期胰腺癌患者的粪便,通过FMT的方法分别移植到胰腺癌模型小鼠的方法,发现长生存期患者的粪便微生物可以有效的激活CD8+T细胞,显著延长小鼠的生存期。最终证明消化道细菌菌群可以定植到胰腺癌肿瘤内而影响肿瘤细菌菌群组成,从而调节免疫功能,最终改变患者的生存期。你怎么看?
菌群多样性是肿瘤病人生存时间长的因还是果呢?其它形式的组学数据,比如:
- DNA Sequencing
- miRNA Sequencing
- Protein Expression array
- mRNA Sequencing
- Total RNA Sequencing
- Array-based Expression
- DNA Methylation
- Copy Number array
难道就不重要吗?