前面我们在生信技能树公众号系统性总结分享过:CRC稳定的分子分型,没有背景知识的朋友可以去深入读文献:October 2015 The consensus molecular subtypes of colorectal cancer。
是CRC分子分型领域的权威,提出4个consensus molecular subtypes (CMSs),该研究整合了18个公共数据集,成功的划分成为了稳定的4类:
- CMS1 (microsatellite instability immune, 14%), hypermutated, microsatellite unstable and strong immune activation;
- CMS2 (canonical, 37%), epithelial, marked WNT and MYC signaling activation;
- CMS3 (metabolic, 13%), epithelial and evident metabolic dysregulation;
- CMS4 (mesenchymal, 23%), prominent transforming growth factor–β activation, stromal invasion and angiogenesis.
那么如果大家拿到了CRC的表达矩阵,如何根据这个分类,来定义自己的样本呢?首先看CMScaller这个R包
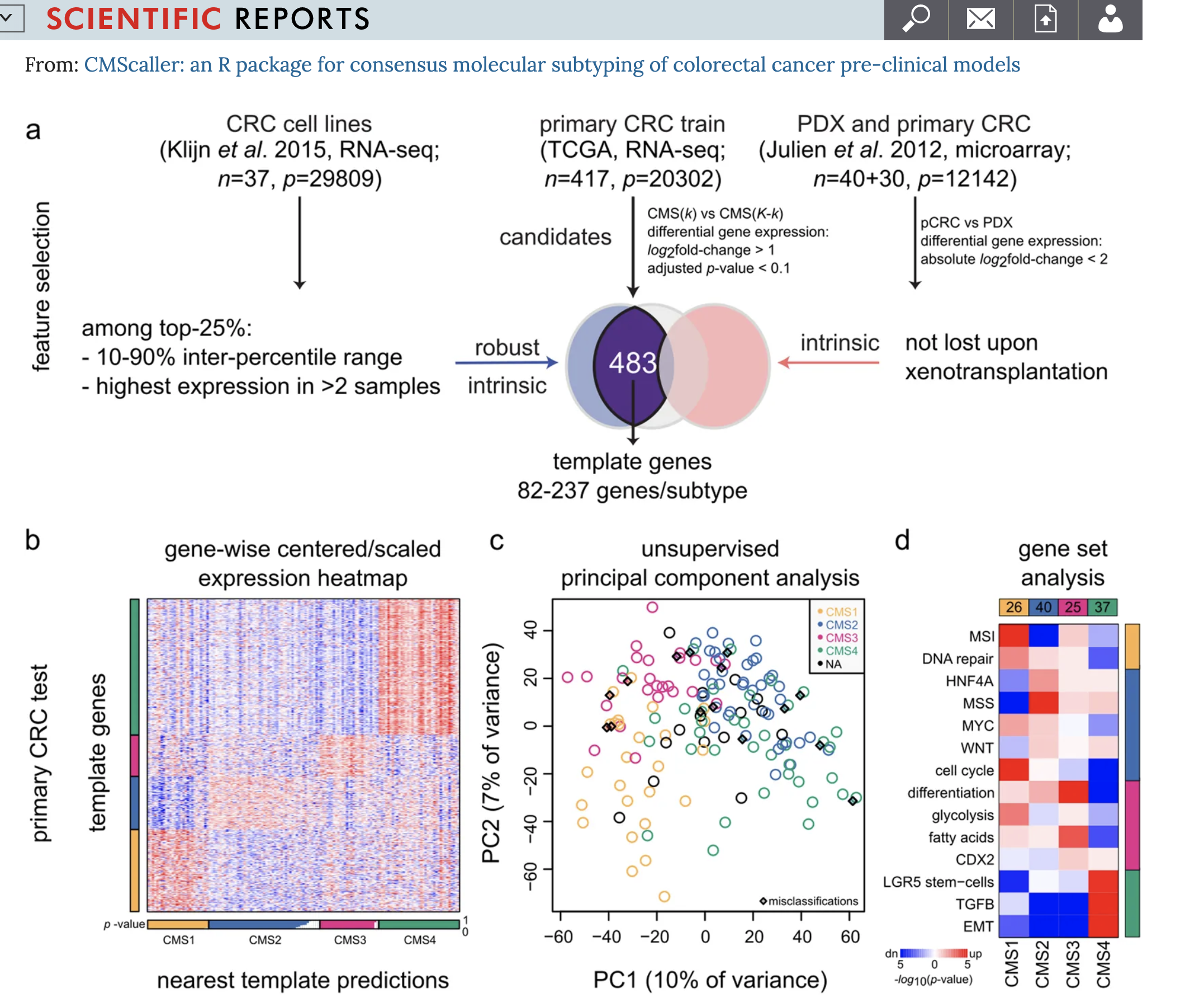
文章:November 2017 提出的R包CMScaller,虽然其发表的杂志不怎么样。
CMScaller: an R package for consensus molecular subtyping of colorectal cancer pre-clinical models
然后看DeepCC
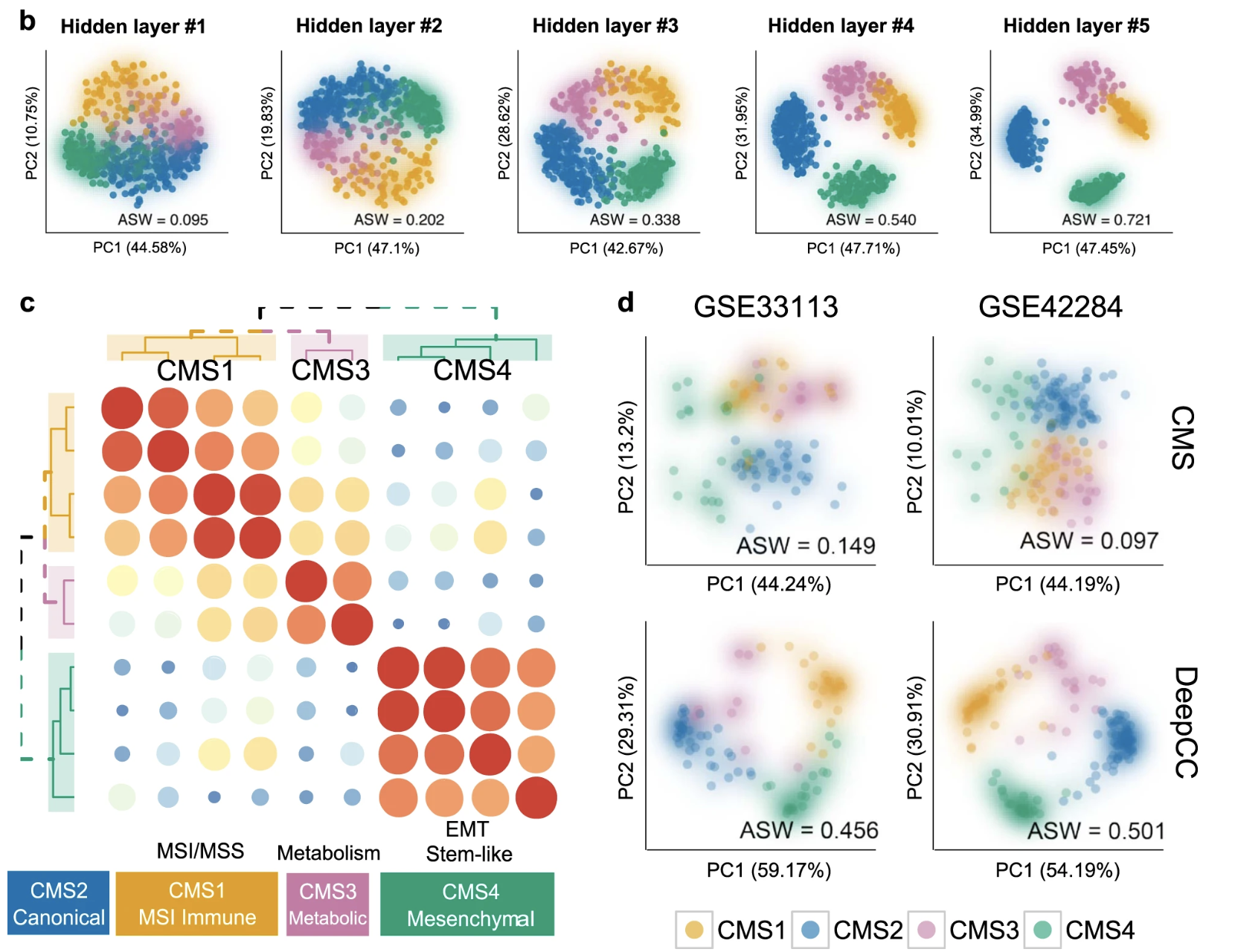
最近(August 2019)从发表的:DeepCC: a novel deep learning-based framework for cancer molecular subtype classification !
区分CRC稳定的分子亚型的效果非常好!
作者是高峰,在香港城市大学和美国康奈尔大学的联合项目中取得肿瘤学博士学位,经中山大学百人计划引进,现任中山大学附属第六医院副研究员,为icgc-argo结直肠癌项目核心成员,团队拥有病人队列全周期的高通量组学、数字病理、医学影像等pb级大数据。主要研究方向为结合人工智能、机器学习分析大规模、高通量、多组学生物医学大数据,开发用于肿瘤精准医学的筛查、早期诊断、精确分型、敏感药物筛选、疗效监控和预后判断等分子诊断技术。近两年在胃肠病学顶级杂志gastroenterology、外科学顶级杂志annals of surgery等发表sci论文17篇(2018-2019,总影响因子 > 130分),其中第一作者及通讯作者(含共同贡献)13篇(总影响因子> 90分),并开发多个生物信息学分析软件包deepcc、htsanalyzer2等,申请12项国家发明专利。
谷歌scholar:http://1t.click/bpvh
research gate:https://www.researchgate.net/profile/feng_gao103
高峰课题组正在招聘博士后名额若干,随时开放,基本待遇为30-34万年薪,可依托深圳校区有额外18万/年的补助,叠加广东省各项人才政策,优秀工科背景均可接收。基本要求年龄35岁以下,毕业不超过两年,5分以上或本专业一区论著一作一篇。
简历请发送至gaof57@mail.sysu.edu.cn