日本研究团队关于CRC肿瘤内部异质性的文章 PLoS Genet. 2016 ,在肿瘤内部异质性方向,这个研究不算早,但是多组学(WES,甲基化芯片和拷贝数芯片)是一个亮点,样本量也不错,但是发表在这个杂志让我比较惊讶,可能是他们不太在乎影响因子吧。
多位点取样
如下图:
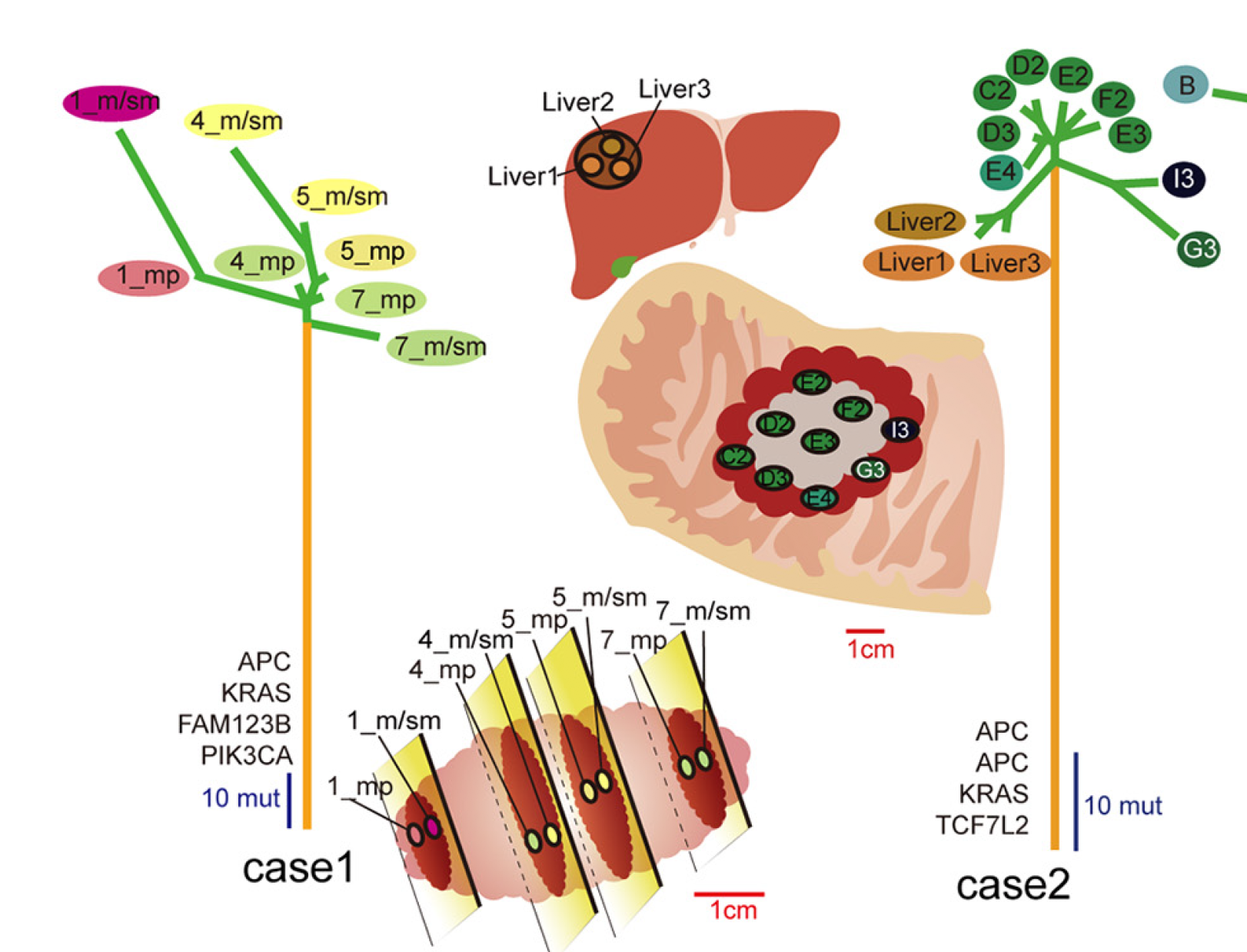
4 种数据
首先必须是WES数据啦,然后还有甲基化芯片和拷贝数芯片数据,还有表达量数据:
| GPL13534 | Illumina HumanMethylation450 BeadChip (HumanMethylation450_15017482) |
| —————————————————————————————— | —————————————————————————————— |
| GPL17077 | Agilent-039494 SurePrint G3 Human GE v2 8x60K Microarray 039381 (Probe Name version) 仅仅是case3有表达量数据 |
| GPL18900 | HumanOmniExpress-12v1 BeadChip |
数据都是可以下载,有条件并且感兴趣的可以跟着我们生信技能树的视频教程处理它们。https://space.bilibili.com/338686099/#/
突变结果
总共是:16857 mutations in total, for an average of 58–1195 mutations per sample,这样计算TMB是,1.57–20.2 mutations per megabase.
然后突变分成 “founder” and “progressor” mutations are present in all regions and a subset of regions, respectively. 因为每个病人测序的部位大于3个,所以 progressor 还需要继续分成 “unique” and “shared” mutations
这3种不同的突变使用超深度靶向测序验证: 100% (5068/5068), 93.9% (1745/1857) and 95.4% (1362/1427) of founder, shared, and unique mutations, respectively.
从NGS的角度来看, 这个数据是很可靠的。
肿瘤内部异质性
针对多种数据都可以看异质性
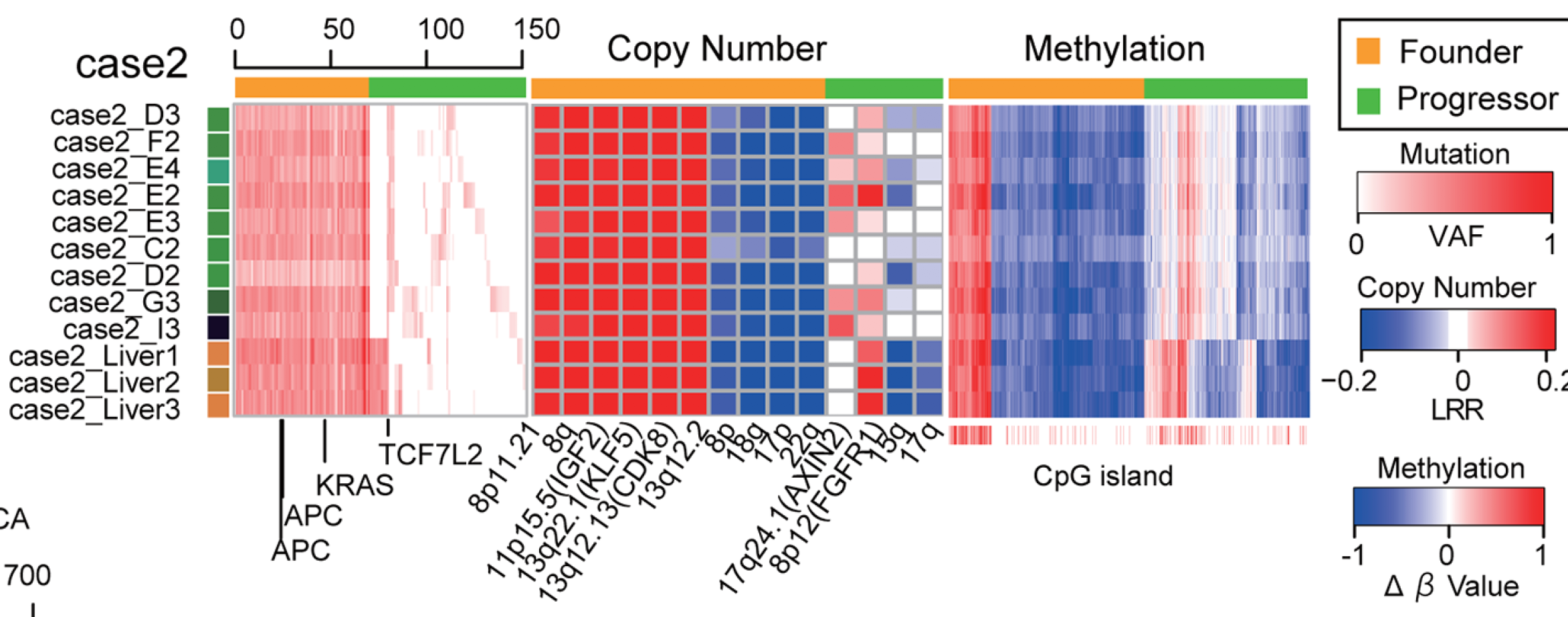
文章亮点
首先是突变跟年龄相关。
然后是作者自己建立了Simulation of cancer evolution , 模拟数据来说明肿瘤是如何进化的。