前些天我在生信技能树介绍过star-fusion:最好用的融合基因查找工具终于正式发表了 ,然后在另外一个教程:一个好像没有做任何改变的参数 提到了目前大量的单细胞转录组数据出来了,却没有一个文章去探索融合基因,也没有人开发工具,是一个空白市场,大家可以试试看哦。虽然商业化很成功的10X仪器做单细胞其实找融合基因还是有点勉强的,毕竟它并不是转录组全长测序,所以基本上很难获得融合位点融合事件,不过,如果是smart-seq2技术实际上是可以的啊!
我不知道哪个研究团队会率先开发出来融合基因流程,不过今天刷Twitter看到了Nathan Castro-Pacheco is presenting STARsolo-Quant, a method for quantifying isoform expression in scRNA-seq data的新闻,是美国生物学圣地冷泉港的Genome Informatics 会议 November 6 - 9, 2019
- https://meetings.cshl.edu/abstracts.aspx?meet=info&year=19
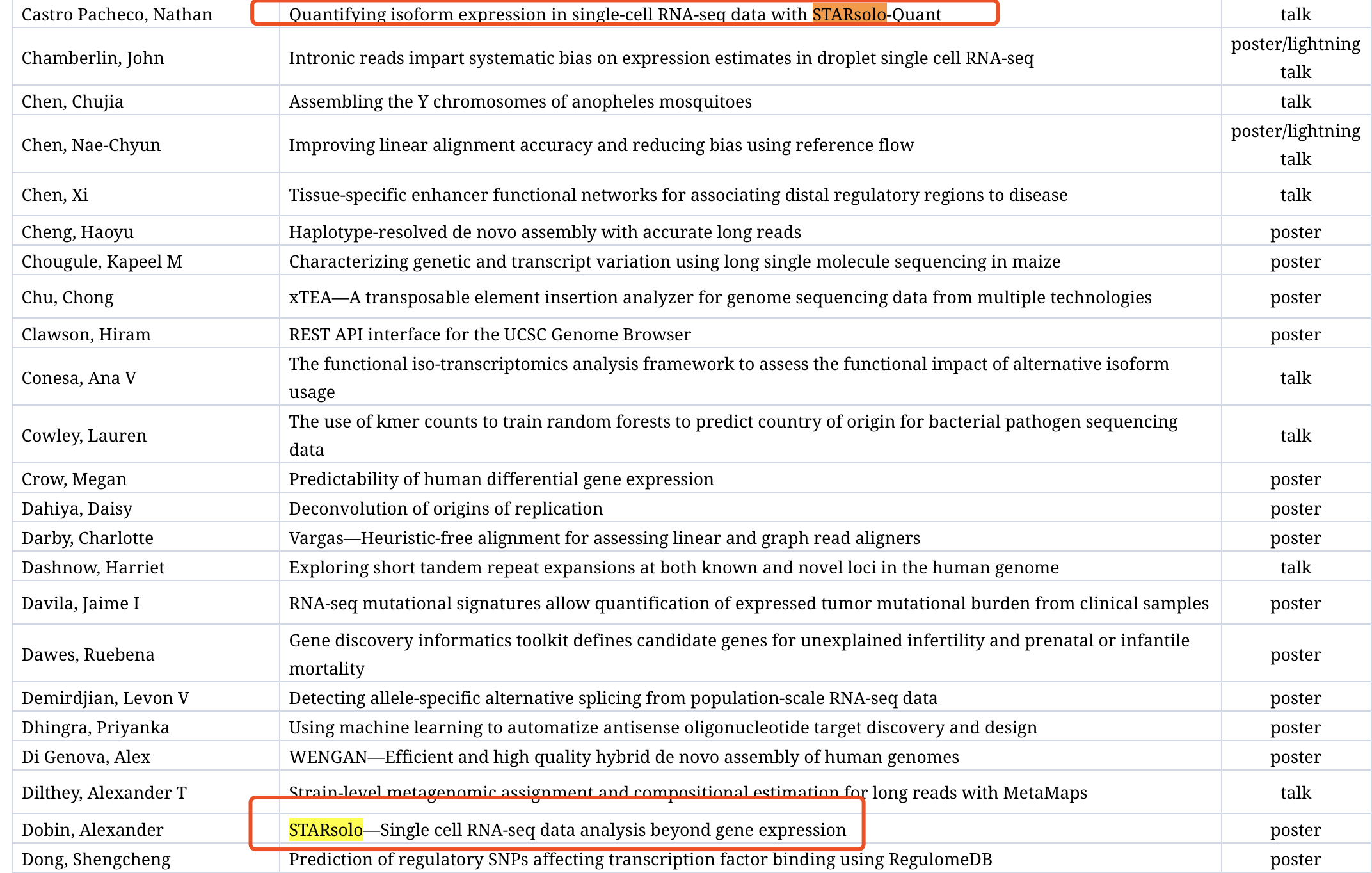
- Quantifying isoform expression in single-cell RNA-seq data with STARsolo-Quant
- STARsolo—Single cell RNA-seq data analysis beyond gene expression
一个poster,一个talk,还是很受大家关注的:
关于STARsolo
因为star软件是10X仪器做单细胞数据的御用软件,所以它自己也在2.7版本更新了STARsolo模块:
- STARsolo: mapping, demultiplexing and gene quantification for single cell RNA-seq.
可变剪切相比融合基因来说,可行度要高一点,期待大家的试用体验哈!
November 6 - 9, 2019