给学徒布置任务,根据我的教程使用salmon流程走一波airway这个转录组数据集,很快就出了结果,为了检查他数据处理的结果准确性,就把我两年前跑的结果给到他,然后让比较一下两个表达矩阵的相关性,结果出乎我意料!
不同流程的表达矩阵居然有批次效应
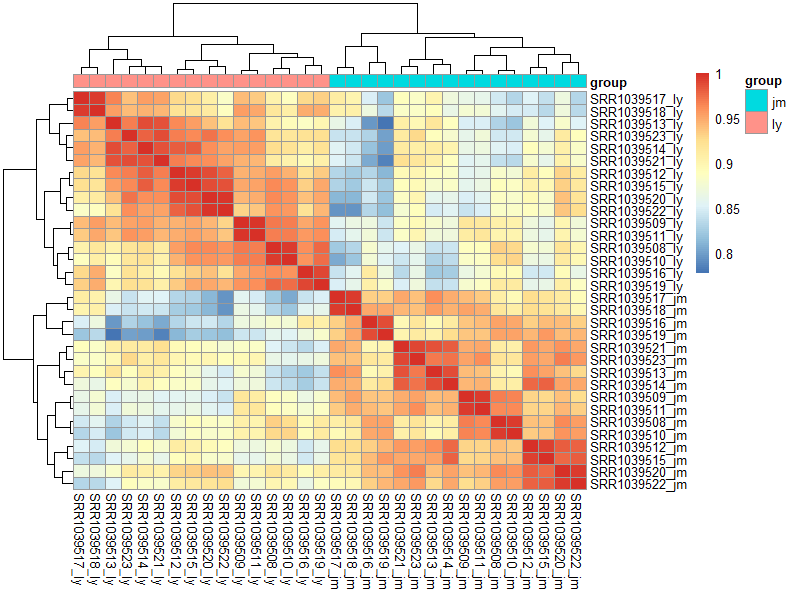
如下,可以看到我们两个人的表达矩阵,很清晰的分成了两个组:
主成分分析也是如此:
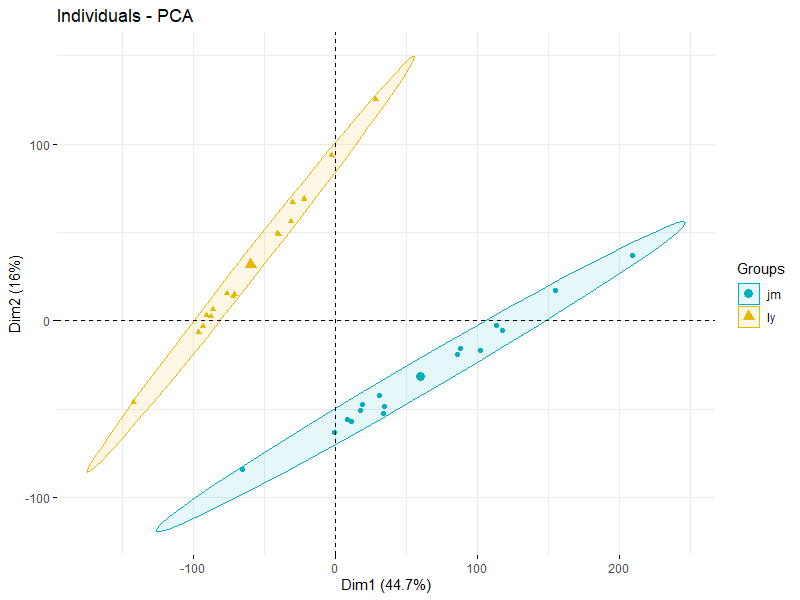
这个是不应该的,理论上来说,不同样本之间是有表达差异的,所以相关性不能太高,而同一个样本在不同流程理论上应该是不能变化太大的。
换一个数据集
起初,我怀疑是数据集的问题,所以让他继续跑了另外一个数据集,就是2018的果蝇的,同样的那个我也是有salmon流程结果,然后继续让他比较他今天的结果和我之前的结果的差异。
这个时候就清晰的看到,同样的一个样本,在salmon流程不同软件版本不同参考转录组得到的表达矩阵差异是很小的。
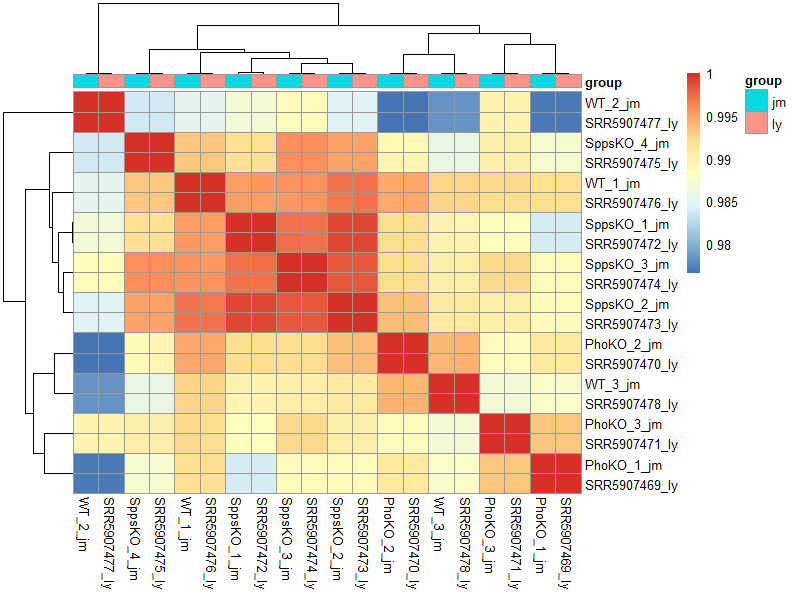
这个才是合理的,一个样本在不同流程表达矩阵需要几乎是一致的才行,如果换个流程就千差万别,那我们生物信息学数据分析也太不靠谱了。
那么前面的表达矩阵出了什么情况呢
随便检查一个样本的两次流程的表达量差异情况:
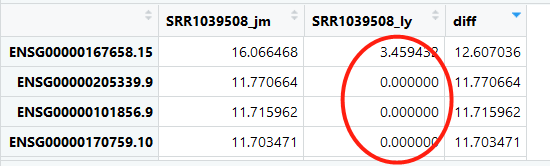
真的是很震惊啊,一个基因表达量差异之大,如云泥之别!
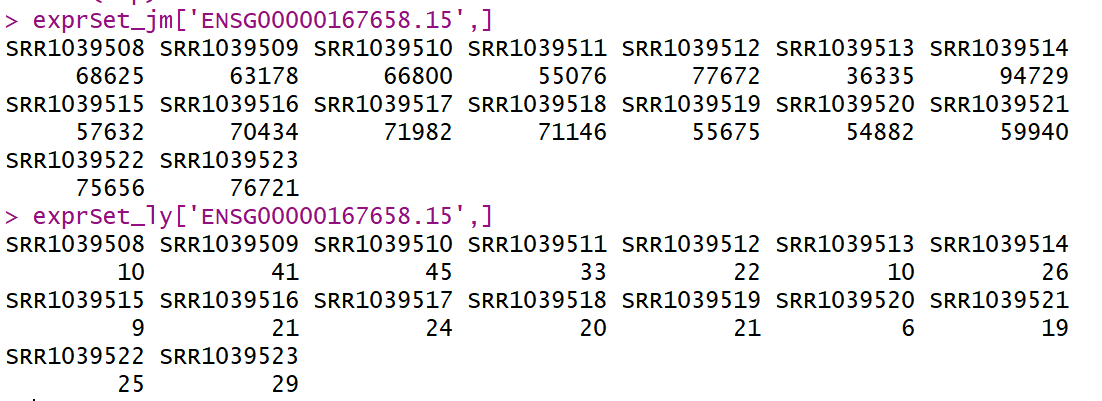
最简单的办法是直接载入bam到igv去查看该基因
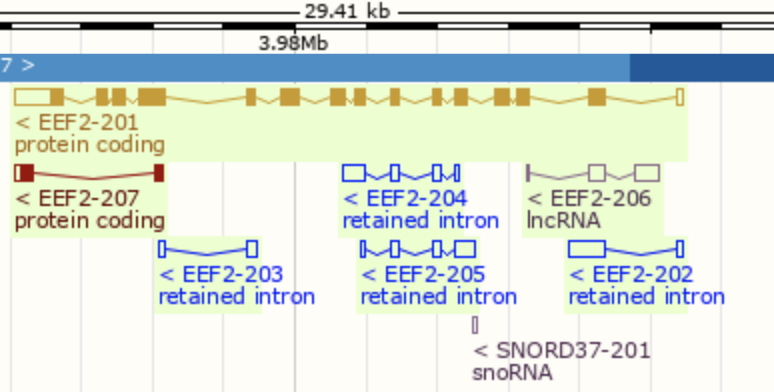
很简单的网页工具拿到其基因名字:https://www.ensembl.org/id/ENSG00000167658 是 EEF2 (HGNC Symbol) 所以IGV定位查看:
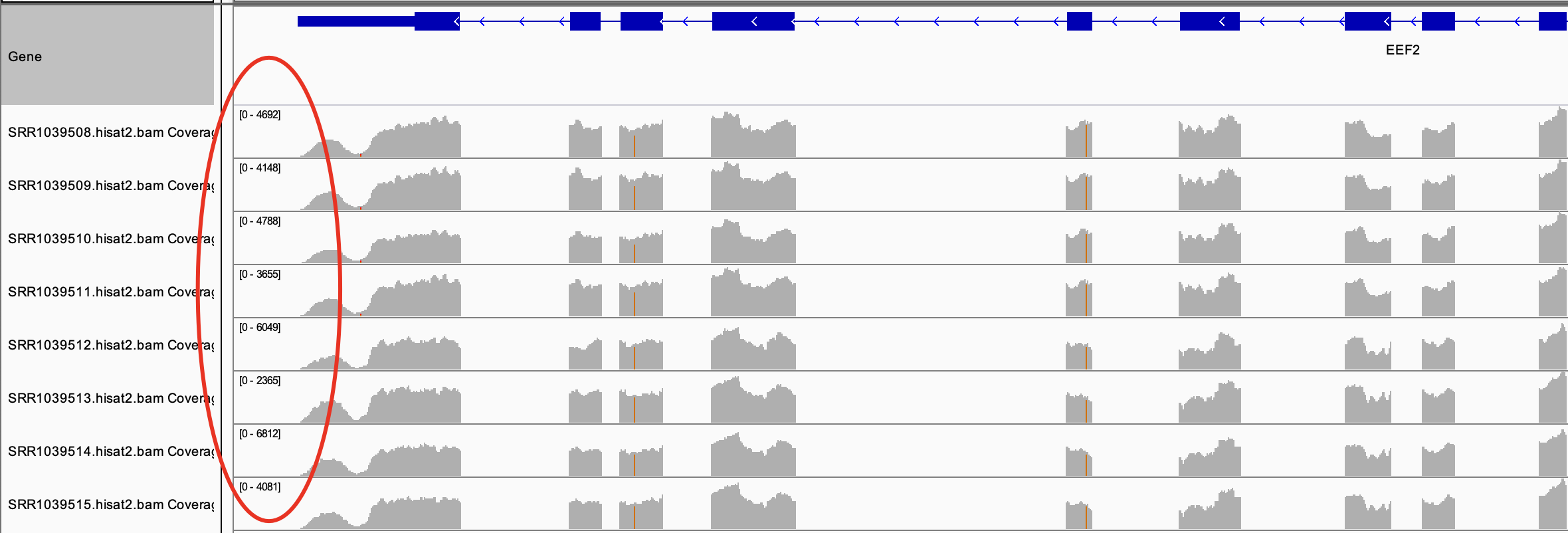
很明显,这么长的一个基因,这么高的表达量,所以学徒跑流程肯定错了,这个基因有很多转录本如下: