记得上半年参加某个单细胞会议,深圳某医院科研负责人(院长吧)豪气的说,大家尽管来我们医院,保底50万年薪,而且咱们这里单细胞平台全部搭建好了,以后只做单细胞,所有之前使用bulk转录组的实验统统换成单细胞。
当然了,人家院长只是为了吸引人才嘛,玩笑归玩笑,实际上bulk转录组还是有用处了,比如今天组会看到NC的这篇文献 June 2019 题目是Targeting enhancer switching overcomes non-genetic drug resistance in acute myeloid leukaemia 使用了非常多的NGS技术:

当时就镇住我了,毕竟里面居然还有我不会的NGS组学。
让我们言归正传,看看这篇文章是如何把单细胞可以跟bulk转录组结合起来的。
首先是2个病人的5个10X样品
我之前在单细胞天地分享过 两个样品的10x单细胞转录组数据分析策略 有趣的是本文完全就没有任何描述对他们的10X单细胞转录组数据处理,就一些冠冕堂皇的技术描述,也没有把数据上传到GEO。

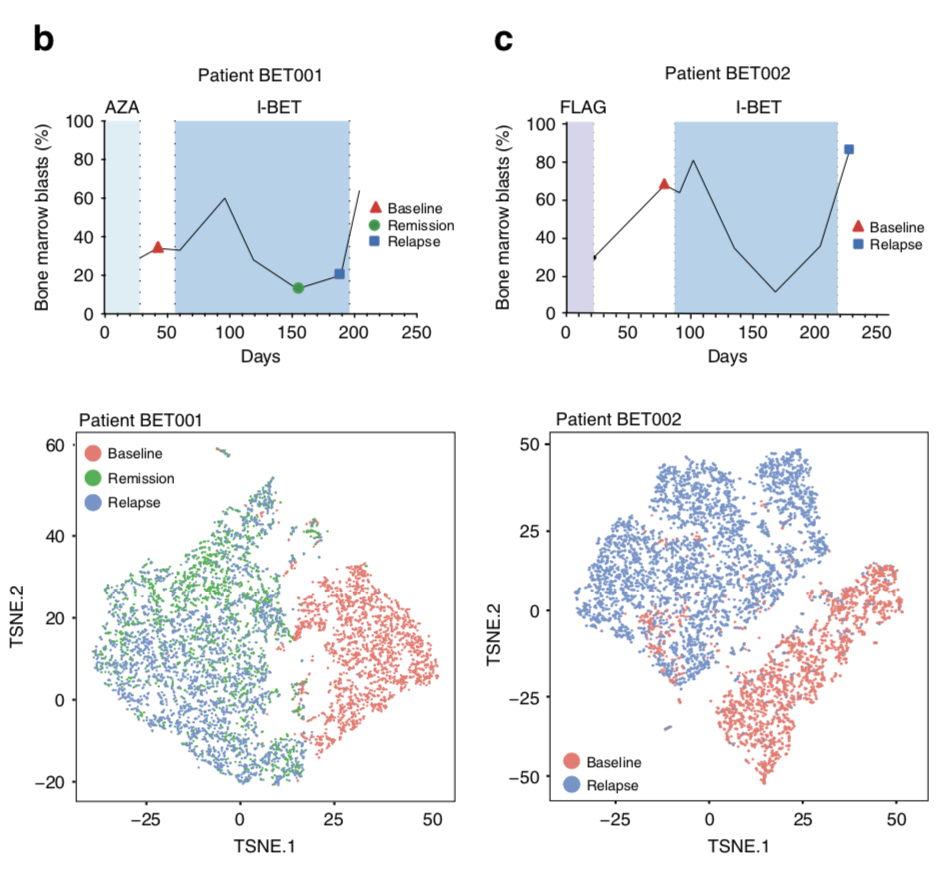
分析策略也很奇怪,可以看到第一个病人是t-SNE analysis of 7360 individual blast cells isolated from patient BET001 at baseline, remission and relapse 第二个病人是 t-SNE analysis of 6349 single blast cells isolated from patient BET002 at baseline and relapse
简单的分群后就没有任何描述,很诡异,也不上传数据,当然了,根据作者的生物学背景,挑选了一个基因集的部分基因可视化表达量,然后还看了看感兴趣的基因是否突变。
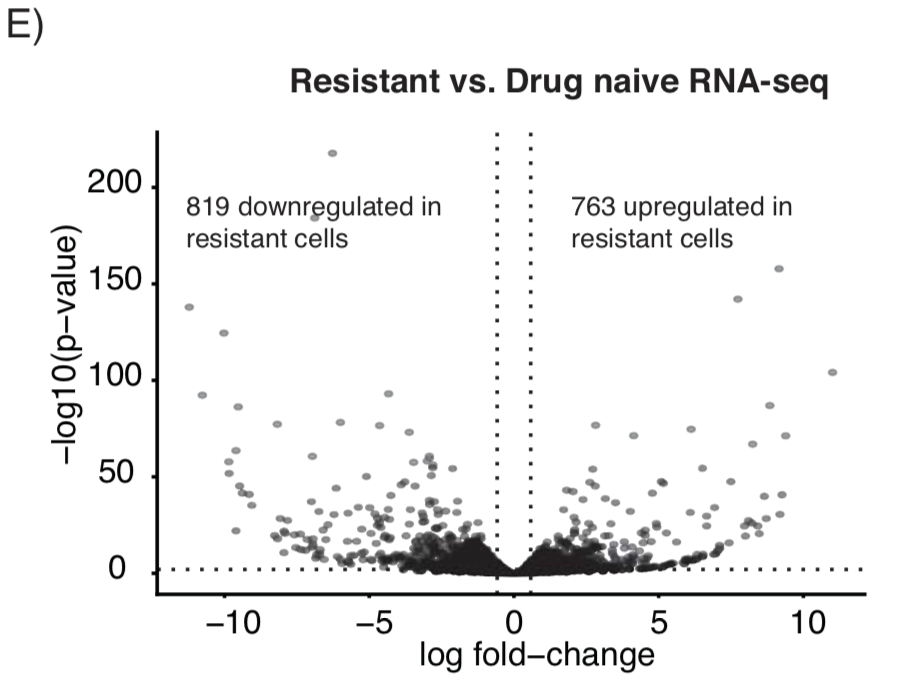
对bulk转录组数据找差异
比如 resistant versus drug naïve cell 的差异基因列表,就被拿去在单细胞转录组数据里面使用monocle看轨迹分析。
差异分析的经典火山图如下:
单细胞转录组还有CEL-SEQ
CEL-Seq(Cell expression by linear amplification and sequencing)恰如其名,是一种采用线性扩增的测序方法。线性扩增的主要优势是错误率比较低,不过线性扩增和PCR都存在序列偏好。CEL-seq技术于2012年发表在《Cell Reports》上,由以色列理工学院的Itai Yanai领导的研究团队开发。这种方法利用带有分子条形码的引物在单管中对每个细胞进行逆转录。在第二链合成后,将所有反应管的cDNA混合,接着进行PCR扩增。扩增后DNA的双端深度测序能够准确检测两条链的序列。2014年《Science》杂志发布的MARS-seq与CEL-seq很类似。
| gse | description |
| —————————————————————————————— | —————————————— |
| GSM3486893 | MLL-AF9_400nM IBET_scRNA-seq |
| GSM3486894 | MLL-AF9_600nM IBET_scRNA-seq |
| GSM3486895 | MLL-AF9_800nM IBET_scRNA-seq |
| GSM3486896 | MLL-AF9_1000nM IBET_scRNA-seq |
| GSM3486897 | MLL-AF9_1000nM IBET_9wks_scRNA-seq |
| GSM3486898 | MLL-AF9_DMSO_9wks_scRNA-seq |
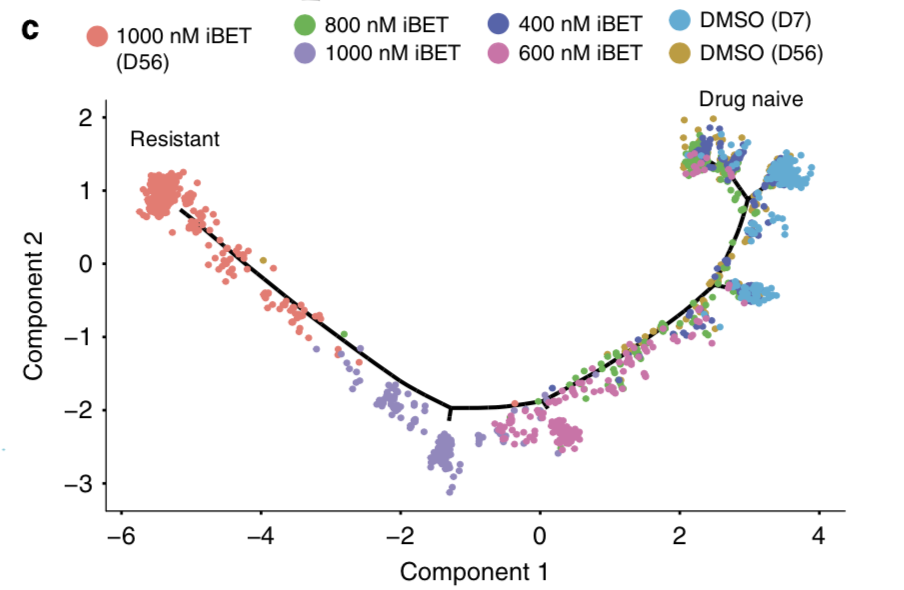
对这些单细胞转录组数据,结合resistant versus drug naïve cell 的差异基因列表,使用monocle看轨迹分析如下;
具体文章的其它科学意义,这里先不说了,反正这篇文章加入了我的200篇NGS文献推荐列表,在2020会详细解读。
后记
这个单细胞转录组结合bulk转录组的分析思路一般般吧,感觉有哪里不对,不过不影响我会把这个分析点当做是我们全网第二个单细胞课程的作业!