转录组已经走入寻常百姓家了,现在生命科学领域实验设计搞个转录组测序就跟PCR和WB一样频繁和普及,但并不是所有人都会分析,我在全国巡讲分享生物信息学经验的时候特别指出了两个容易混淆的知识点,但大伙类似的错误认知还有不少,这里再详细讲解一个,也顺便diss一些已经发表的文章。
理所当然的, 大家做完转录组,从全局表达矩阵,想看到下面的3张图:
- 左边的热图,说明我们实验的两个分组,normal和npc的很多基因表达量是有明显差异的
- 中间的PCA图,说明我们的normal和npc两个分组非常明显的差异
- 右边的层次聚类也是如此,说明我们的normal和npc两个分组非常明显的差异
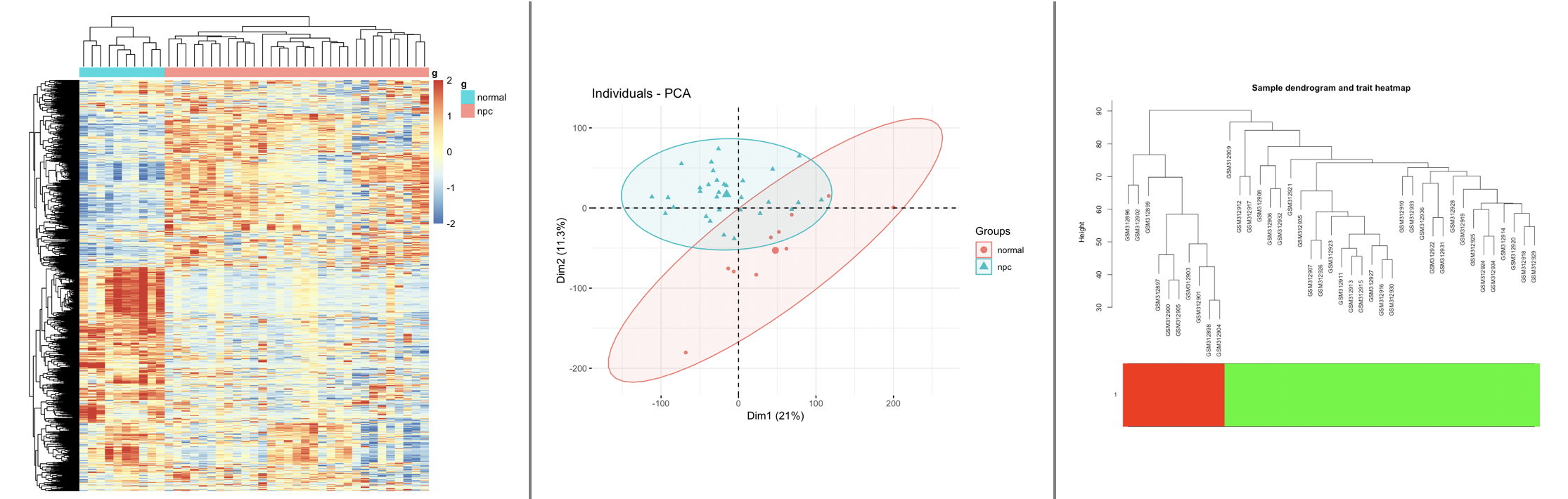
PS:如果你的转录组实验分析报告没有这三张图,就把我们生信技能树的这篇教程甩在他脸上,让他瞧瞧,学习下转录组数据分析。
PS: 示例的3张图来自于我的GitHub博客:https://github.com/jmzeng1314/GEO是不是转录组表达矩阵就应该按照表型信息如此泾渭分明呢?
诚然,有上面那样清晰可见的差异,这样的结果当然让人happy,但并不意味着所有的实验设计的结果都应该如此, 任何差异都应该是可以解释的,上图的normal和npc两个分组样本本来就是截然不同,它们的差异也就合情合理啦!
但是,我们来看看另外一篇文章发表在Neuropsychopharmacology. 2014 Aug;影响因子大概是7 ,题目是:A molecular profile of cocaine abuse includes the differential expression of genes that regulate transcription, chromatin, and dopamine cell phenotype. 有点长,但是文章故事很简单,就是两组人的转录组: - Subject inclusion in the cocaine cohort (n=10) was based on determination of cocaine abuse as the cause of death, a documented history of drug abuse
- Control subjects (n=10) died as a result of cardiovascular disease or gunshot wound, had no documented history of drug abuse, and tested negative for cocaine and other drugs of abuse
共20个人,每个人3次技术重复,所以是60个数据,但是如果我们天真的以为毒品上瘾与否的人,大脑的全局基因表达就应该是有翻天覆地的差异就错了,如下:
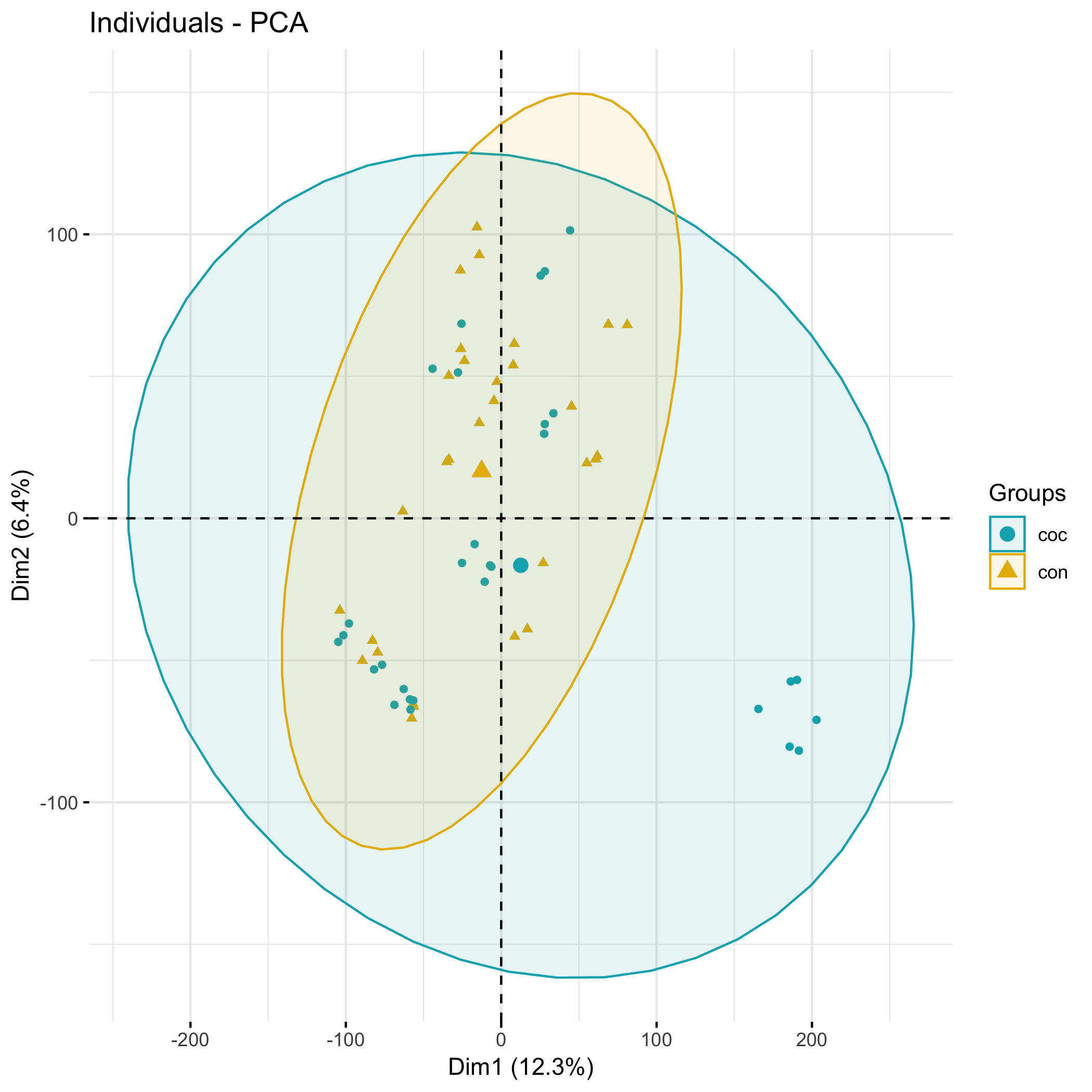
可以看到, control组合毒品组,基本上是无法区分开来的,在PCA里面,这个时候就需要仔细看热图:
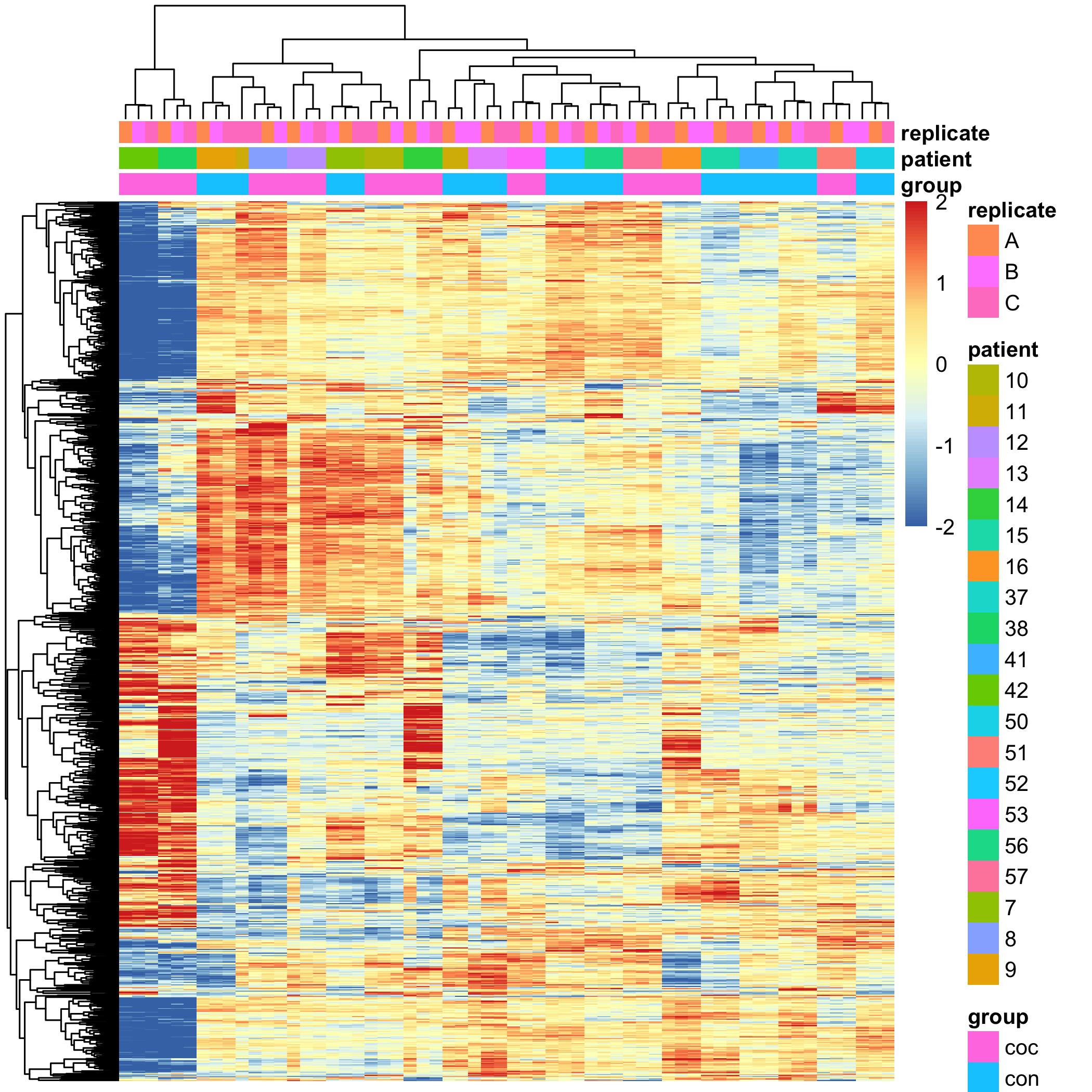
很明显,可以看到同一个人的3次技术重复差异非常小,这个符合预期,但是呢,人并没有按照毒品上瘾与否来区分,而是不同人之间的异质性非常高,而且中间还有3个病人,他的3次技术重复都出现了离群点,所以是需要去除的,这一点,文章做的很对!
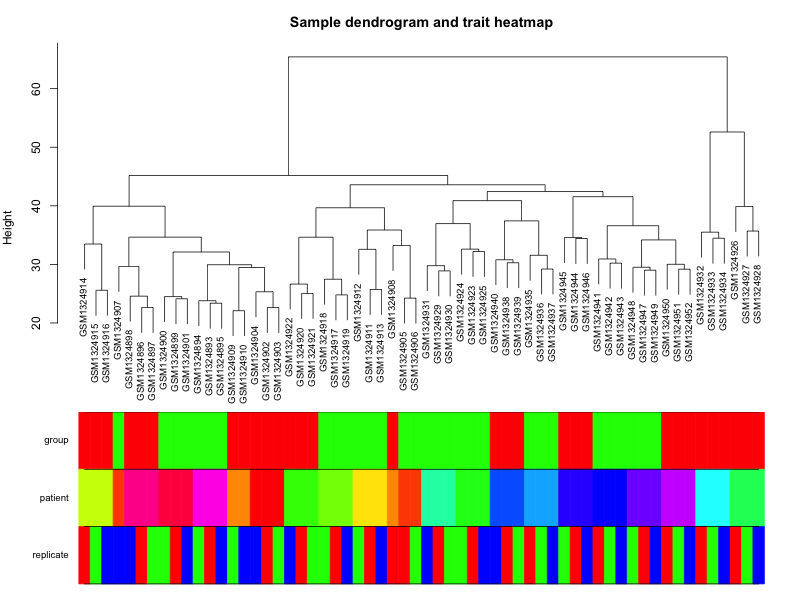
然后,文章就使用这样的表达矩阵和分组信息,去找差异基因了,找毒品上瘾与否不同组的人的差异表达基因,毫无疑问,这样的差异分析,即使把阈值调的再低,也没多少统计学显著性的基因能被找出来的。

文章里面就91个基因,很有趣的是他们使用qPCR实验验证了他们的芯片技术找到的差异是可靠的,当然, 这样他的生物学故事就足够solid了,这个是历史遗留问题,大家喜欢相信实验结果,搞得好像我们的ngs数据分析只能是一个引子一样。
实际上,这样的分析明显是有问题的, 既然不同的人差异这么大,理论上就可以把人当做是一个批次效应,使用北京大学李程课题组开发的sva包的combat函数,把这样的效应去除一下,接着再找差异。那个才更有可能是毒品上瘾与否的差异啊!全网最系统的表达芯片数据处理教程
表达芯片数据处理教程,早在2016年我就系统性整理了发布在生信菜鸟团博客:http://www.bio-info-trainee.com/2087.html
配套教学视频在B站:https://www.bilibili.com/video/av26731585/
代码都在:https://github.com/jmzeng1314/GEO 早期目录如下:
- 第一讲:GEO,表达芯片与R
- 第二讲:从GEO下载数据得到表达量矩阵
- 第三讲:对表达量矩阵用GSEA软件做分析
- 第四讲:根据分组信息做差异分析
- 第五讲:对差异基因结果做GO/KEGG超几何分布检验富集分析
- 第六讲:指定基因分组boxplot指定基因list画热图
- 第七讲:根据差异基因list获取string数据库的PPI网络数据
- 第八讲:PPI网络数据用R或者cytoscape画网络图
- 第九讲:网络图的子网络获取
- 第十讲:hug genes如何找
公众号推文在: - 解读GEO数据存放规律及下载,一文就够
- 解读SRA数据库规律一文就够
- 从GEO数据库下载得到表达矩阵 一文就够
- GSEA分析一文就够(单机版+R语言版)
- 根据分组信息做差异分析- 这个一文不够的
- 差异分析得到的结果注释一文就够
给你的作业
首先,这篇文章标题我给到你了,你应该是能找到数据集下载链接,走一波我的标准GEO代码,试图理解图表,然后说出你的想法。那么就做起来吧,把你的分析结果发邮件给我,发到我邮箱 jmzeng1314@163.com 并且写出你的故事,就有惊喜哦!
还有另外一个数据集,也是如此, 建议你花时间一起摸索一下,台湾人的研究,数据集GSE128502,分析也是错的。