发表于2018的NC,文章是:Unravelling subclonal heterogeneity and aggressive disease states in TNBC through single-cell RNA-seq 对6个TNBC病人总共测了 超过1500个单细胞 ,质控后还剩下1189个单细胞进入下游分析。使用的是FACS加上Smart-seq2 ,非常中规中矩的分析,所以就发了同样中规中矩的NC。
单细胞转录组细节
代码都公布在GitHub:https://github.com/Michorlab/tnbc_scrnaseq
数据都上传到了GEO: GSE118390.
过滤低质量细胞标准是:
- (i) small library size
- (ii) few expressed genes
- (iii) low total amount of mRNA.
表达矩阵的归一化也很复杂,使用了3个R包,主要是为了去除 technical variability and confounding factors - (1) transform the TPM values into relative counts with the Census algorithm (function relative2abs from the R package monocle
- (2) normalize the Census counts with the deconvolution strategy implemented in the R package scran
- (3) remove additional sources of unwanted variation in the scran-normalized Census counts with RUVSeq
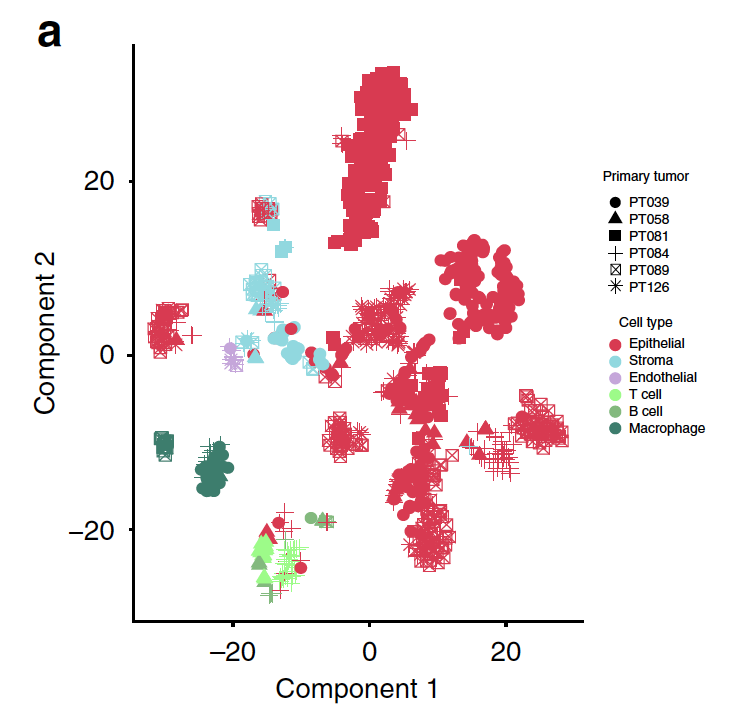
鉴别细胞类型
可以看到不同的上皮细胞(通常是肿瘤细胞)根据不同的病人分开了,而非上皮细胞(通常是免疫细胞)根据细胞类型分开的,而免疫细胞常见的种类在这里都有体现。
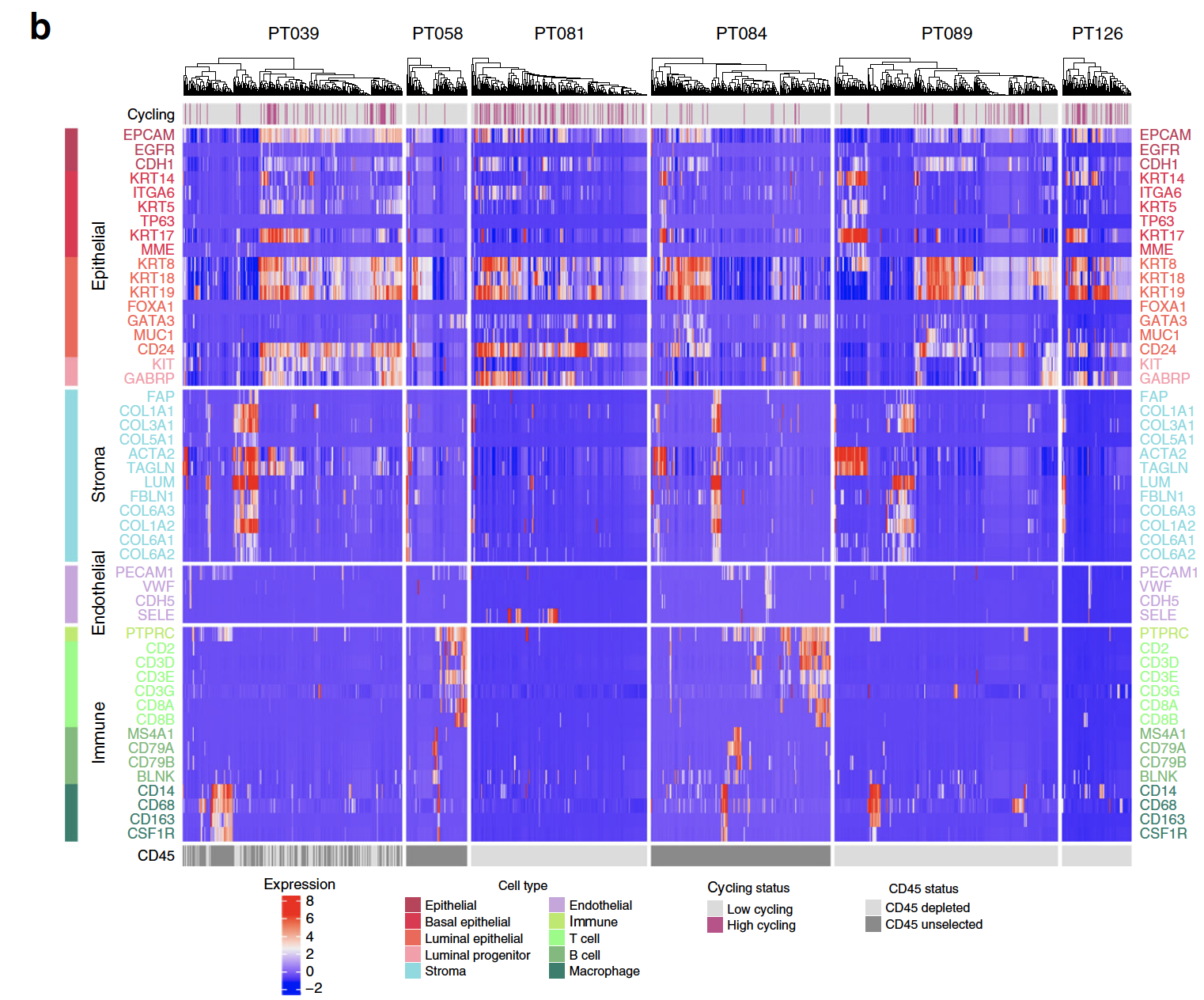
不同类型的细胞的marker基因是:
而且也是同样的方法来区分恶性的肿瘤上皮细胞,和免疫细胞,就是计算全局转录水平的拟CNV状态,可以看到肿瘤细胞通常是由CNV事件的,而正常细胞基本都是2倍体。
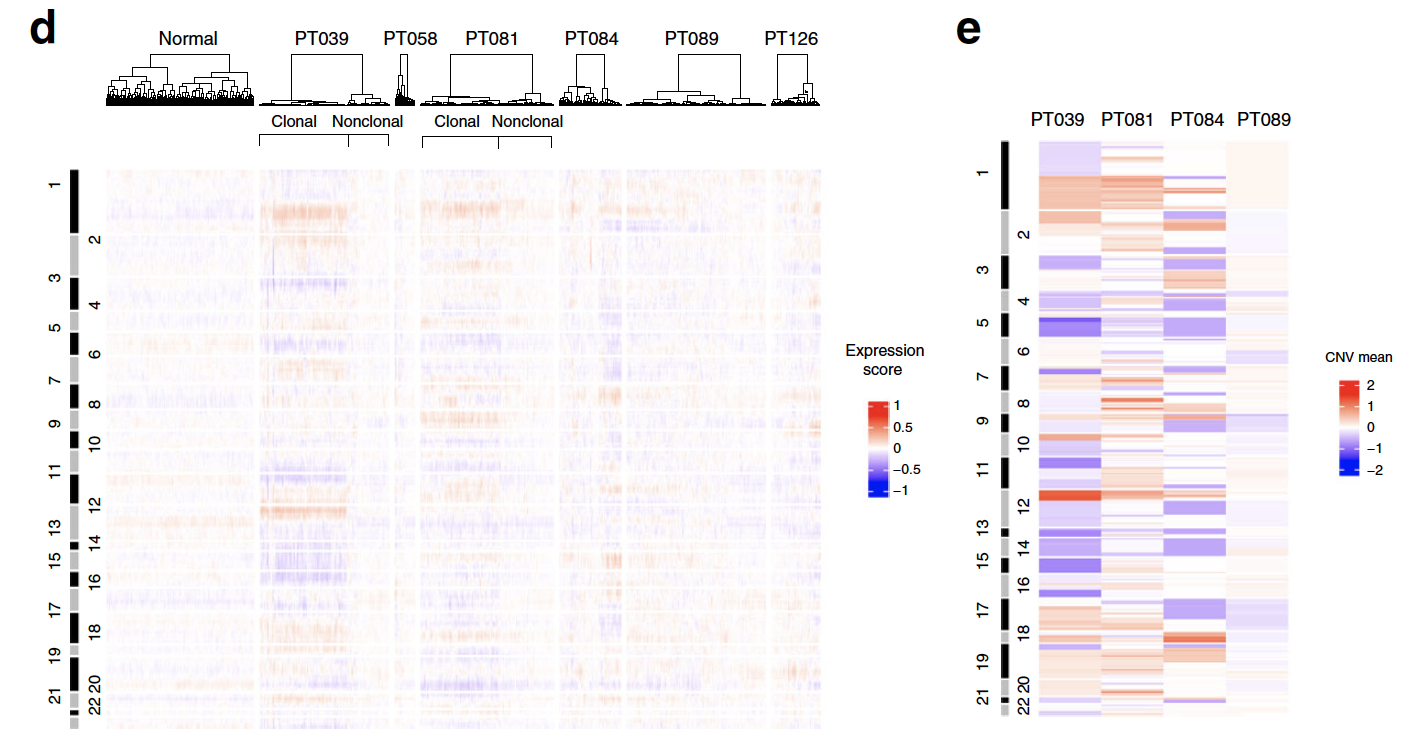
有了上面的分类信息,就可以把CNV正常的细胞单独看tSNE聚类情况:
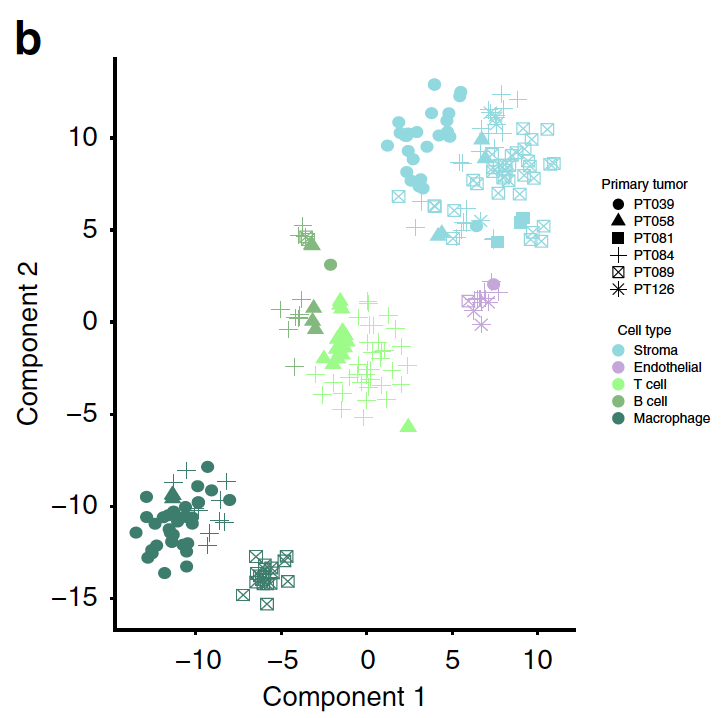
也可以把肿瘤细胞单独拿出来看,这里可以看到肿瘤细胞的肿瘤病人效应大于肿瘤细胞类型,而上面的正常细胞分群的时候,病人效应很容易就看不到了。
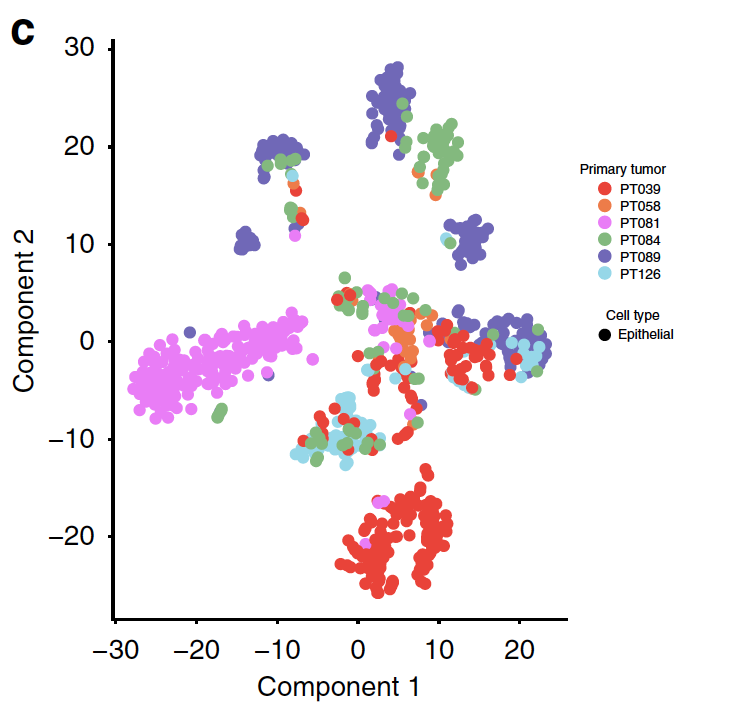
去除肿瘤细胞病人聚类效应
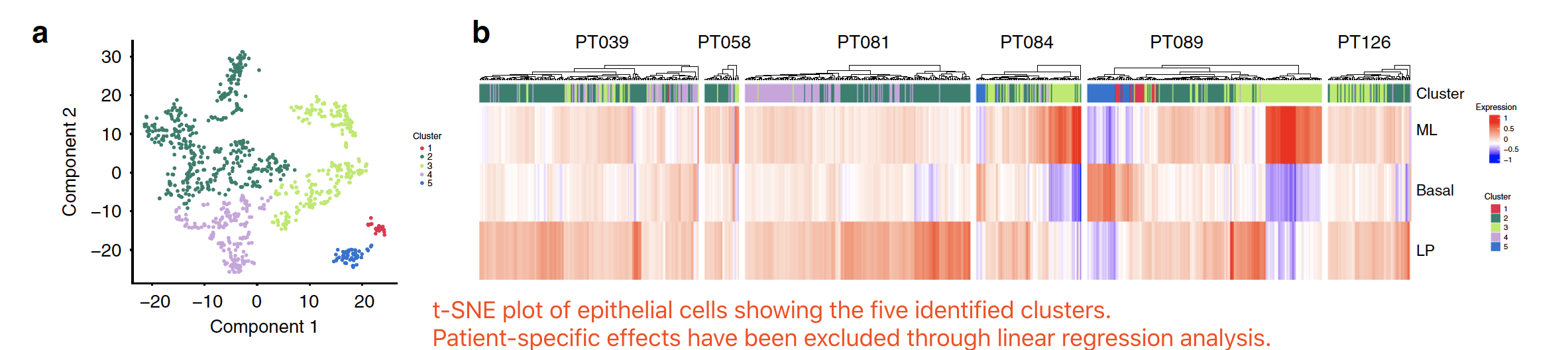
这个时候,作者并没有向肿瘤细胞的病人效应投降,而是通过线性回归去除了病人效应,这样6个病人的肿瘤细胞可以分成5组,如下:
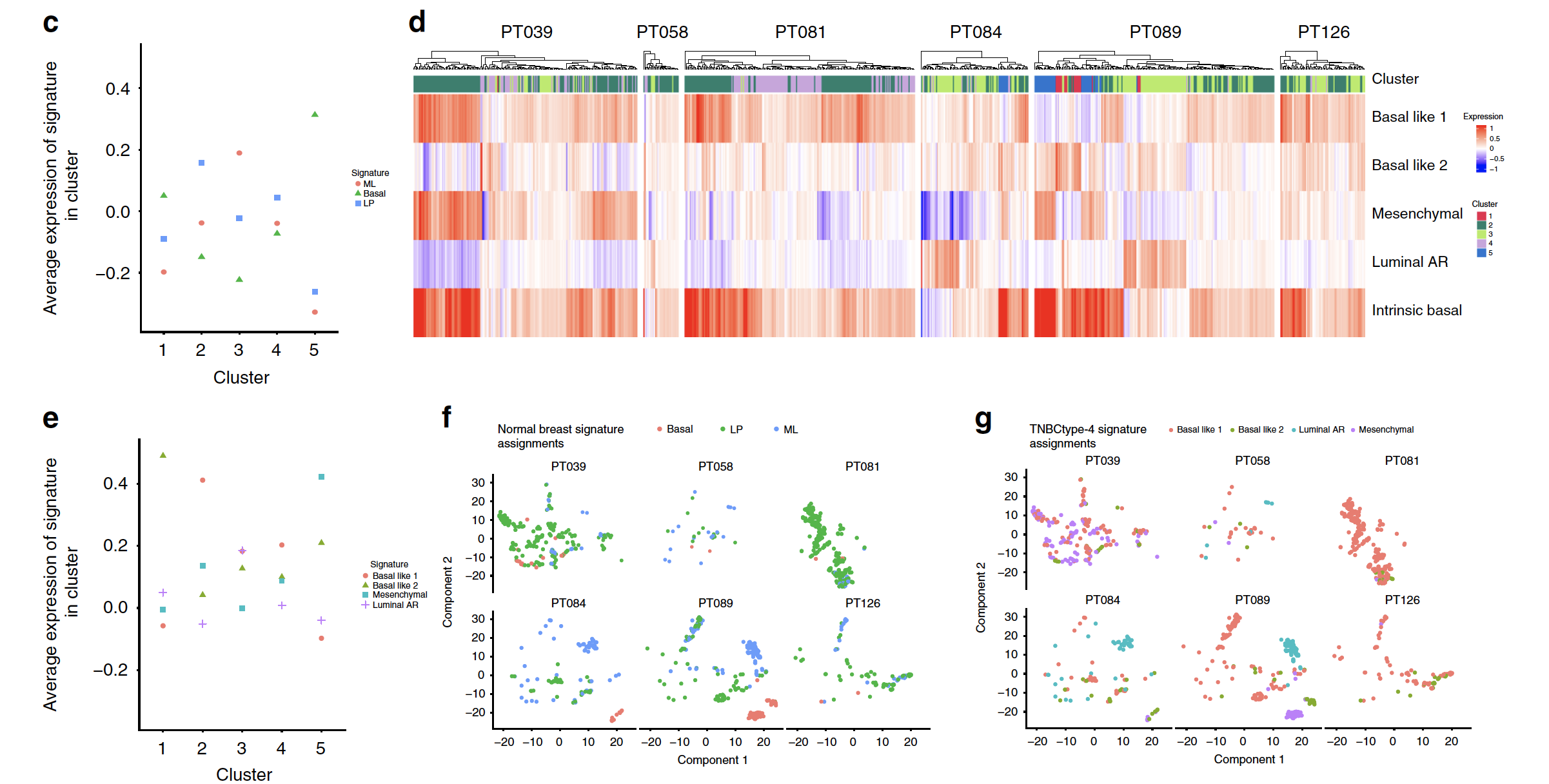
这5类细胞,就可以去和传统bulk转录组测序的TNBC数据分型进行比较,这里作者主要考虑的是four TNBCtype-4 subtype signatures 和 three normal breast epithelial subtypes signatures: ML (mature luminal), basal, and LP (luminal progenitor). 需要自行去查看查看文献,拿到指定的基因。
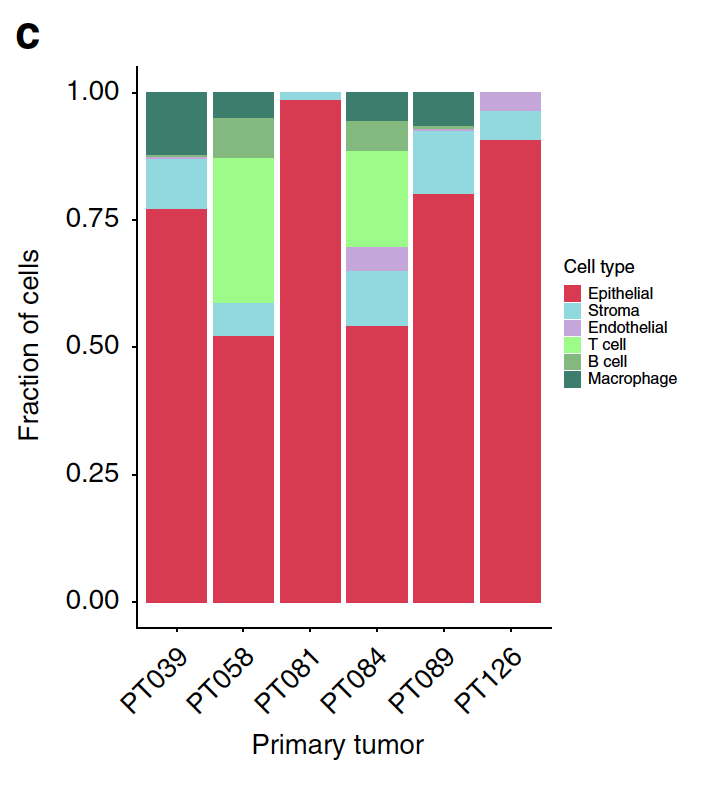
每个病人体内免疫细胞比例:
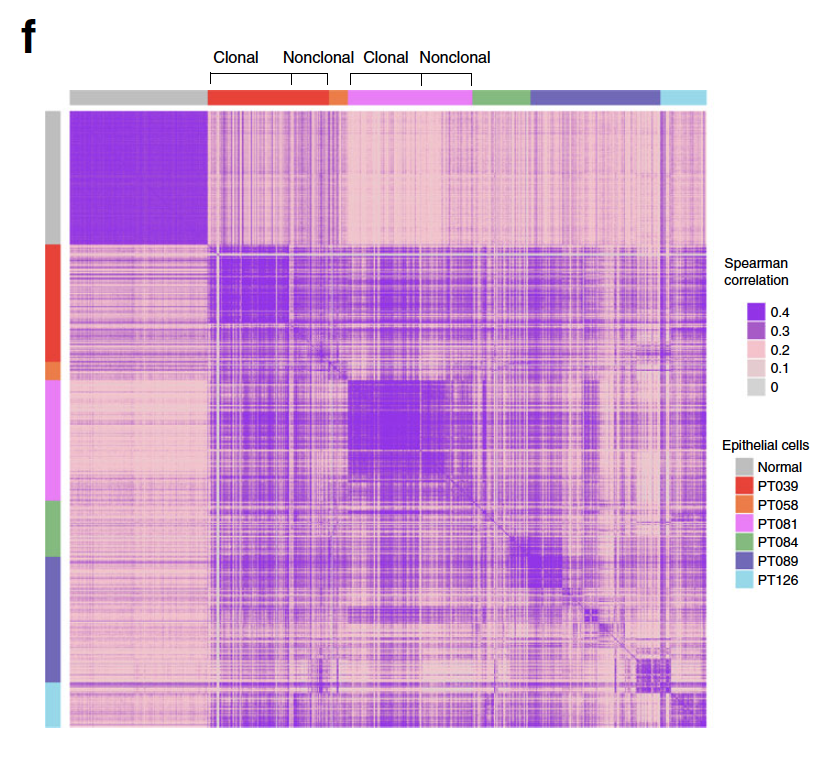
所有细胞之间的表达量的相关性,也是为了展现细胞恶性与否的分类,异质性
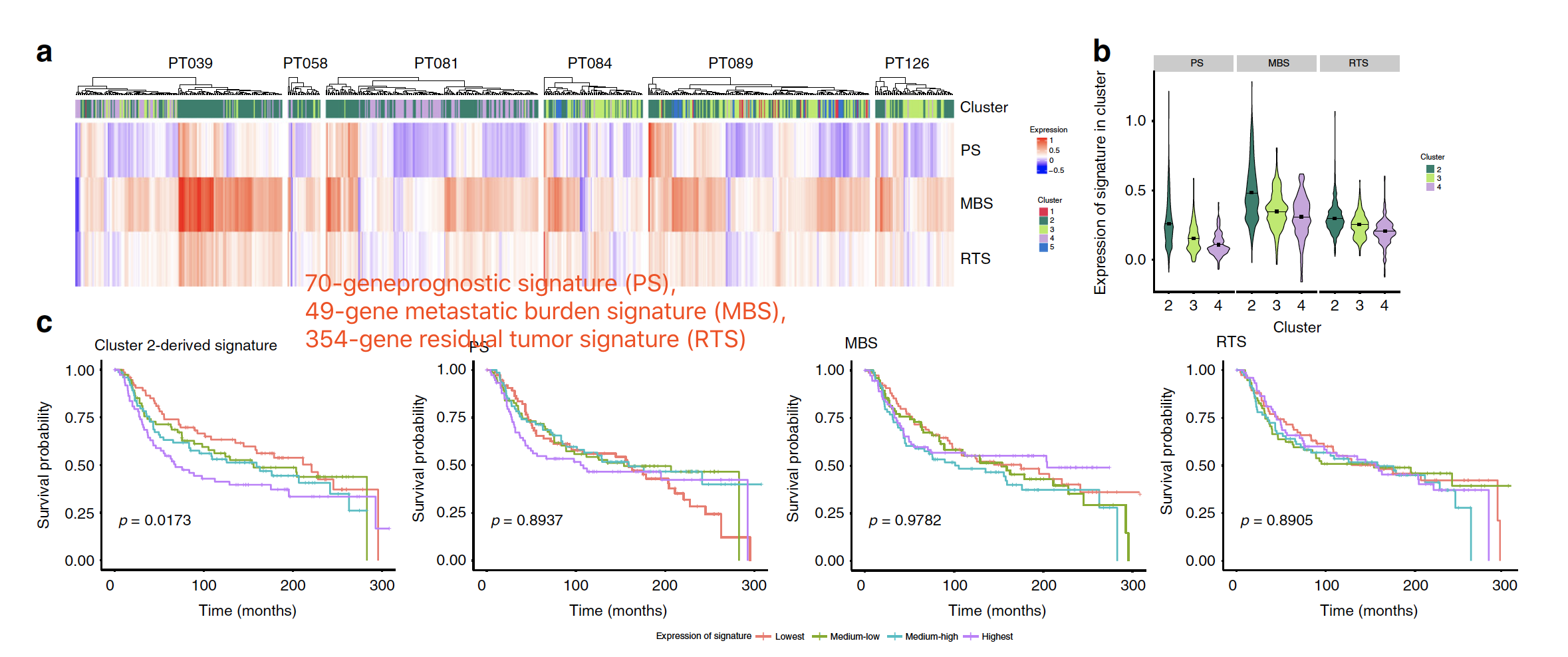
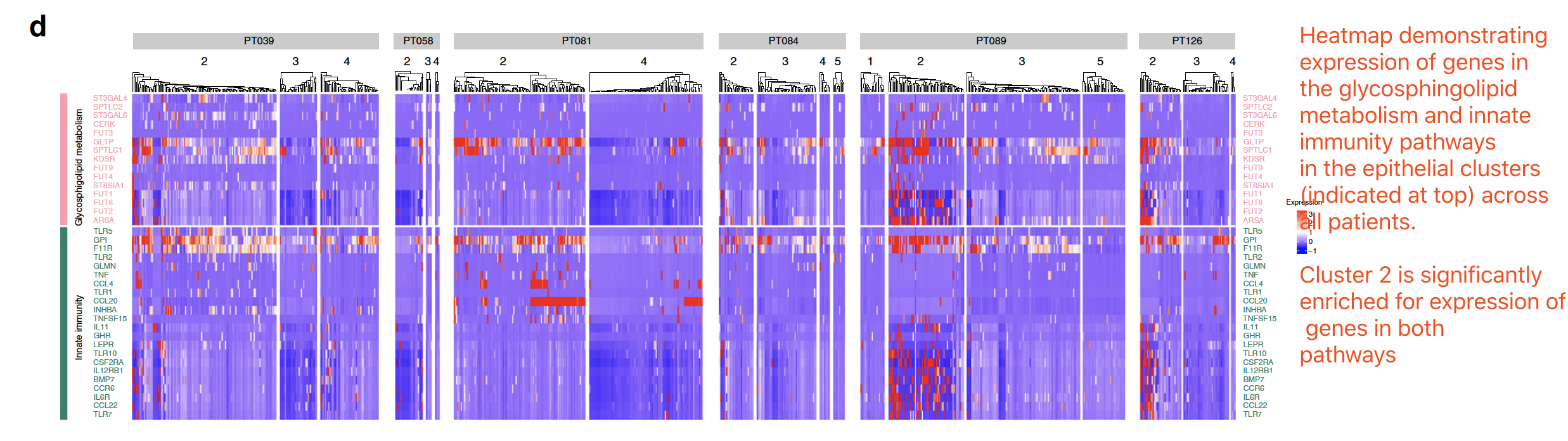
公共数据库结合分析 cluster 2 subpopulation
发现 cluster 2 subpopulation 特有的基因富集在 两个生物学意义的通路,所以热图可视化,
然后是看 3 个乳腺癌基因集: - 70-geneprognostic signature (PS),
- 49-gene metastatic burden signature (MBS),
- 354-gene residual tumor signature (RTS)
可以看到cluster 2 subpopulation 特有的基因集,可以在METABRIC队列显著区分生存,而那3个数据集并不能。