本次分享的文献发表于2018年11月30日,在Science 杂志,“Single-cell Multi-omics Sequencing and Analyses of Human Colorectal Cancer”, 该研究在国际上首次从单细胞分辨率、多组学水平深入解析了人类结直肠癌在发生和转移过程中,基因组拷贝数变异、DNA甲基化异常及基因表达改变的特点及相互关系。
Bian et al. used a single-cell triple omics sequencing (scTrio-seq) technique to examine the mutations, transcriptome, and methylome within colorectal cancer tumors and metastases from 10 individual patients.
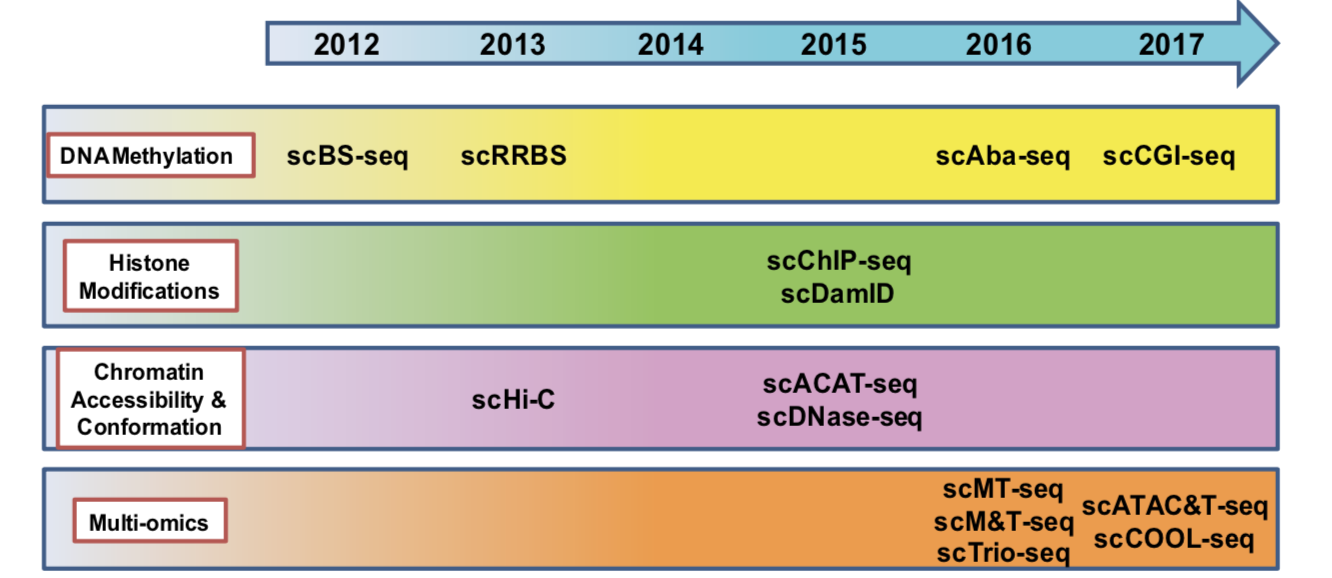
来自于大名鼎鼎的Tang Fuchou 课题组,他们在单细胞领域的技术开发,和发育学应用的多产是有目共睹的:
- scRRBS (Genome Research, 2013);
- scMicrofluidic-seq (PNAS, 2014);
- SUPER-seq (Genome Biology, 2015);
- scTrio-seq (Cell Research, 2016);
- scCLEVER-seq (Cell Stem Cell, 2017b);
- scCOOL-seq (Cell Research, 2017);
- MR-seq (Science Bulletin, 2017)
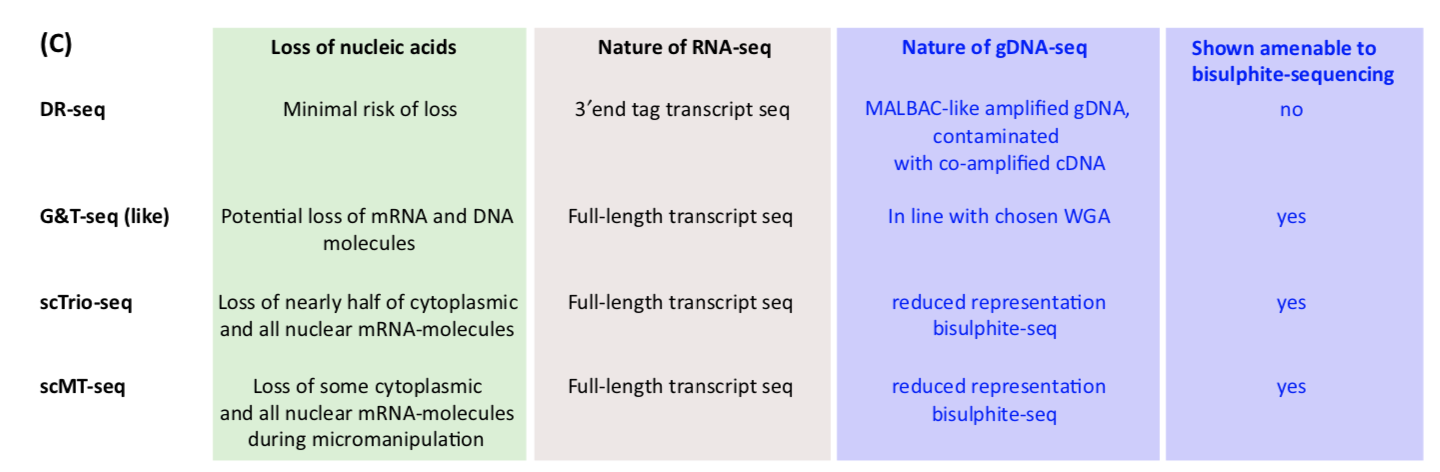
关于scTrio-seq
早在2016年,一种创新性的单细胞三重组学测序(triple omics sequencing)技术:scTrio-seq就被汤富酬研究组发表了,并利用它揭示出了肝细胞癌中的遗传、表观遗传及转录组异质性。这一重要的研究发布在2016年2月23日的《Cell Research》杂志上。
2018年11月30日,汤富酬研究组对该团队已发表的scTrio-seq(single-cell triple omics sequencing technique)技术进行了大幅度优化,极大地提升了对单个细胞转录组的检测效率和DNA甲基化位点(CpG位点)的覆盖度,达到与利用完整单细胞进行高精度转录组分析,或者利用完整单细胞进行高精度DNA甲基化组分析相近的效果。
有综述 Trends in Genetics, February 2017, http://dx.doi.org/10.1016/j.tig.2016.12.003 对一些类似的技术进行比较:
也有综述 Lo PK, Zhou Q (2018) Emerging Techniques in Single-Cell Epigenomics and their Applications to Cancer Research. J Clin Genom. 总结了单细胞技术在表观领域的进展:
参考:- http://www.nsfc.gov.cn/csc/20340/20343/33134/index.html
- http://jiangsu.china.com.cn/html/tech/rdzx/4430365_1.html
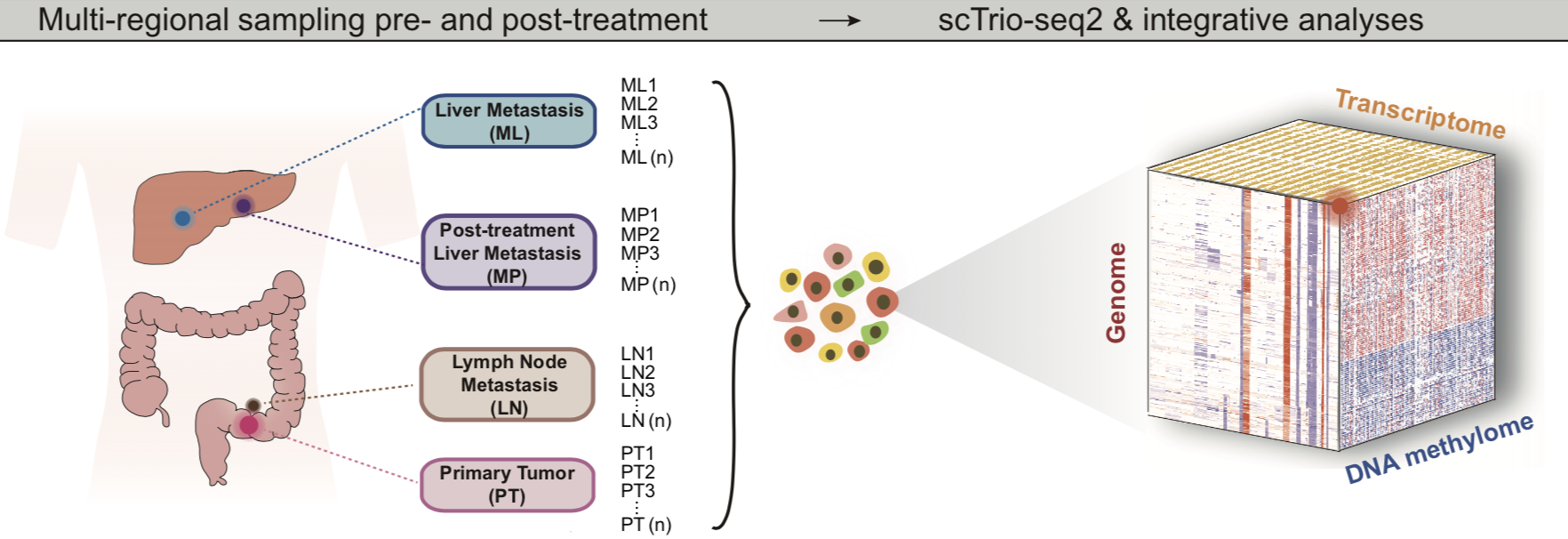
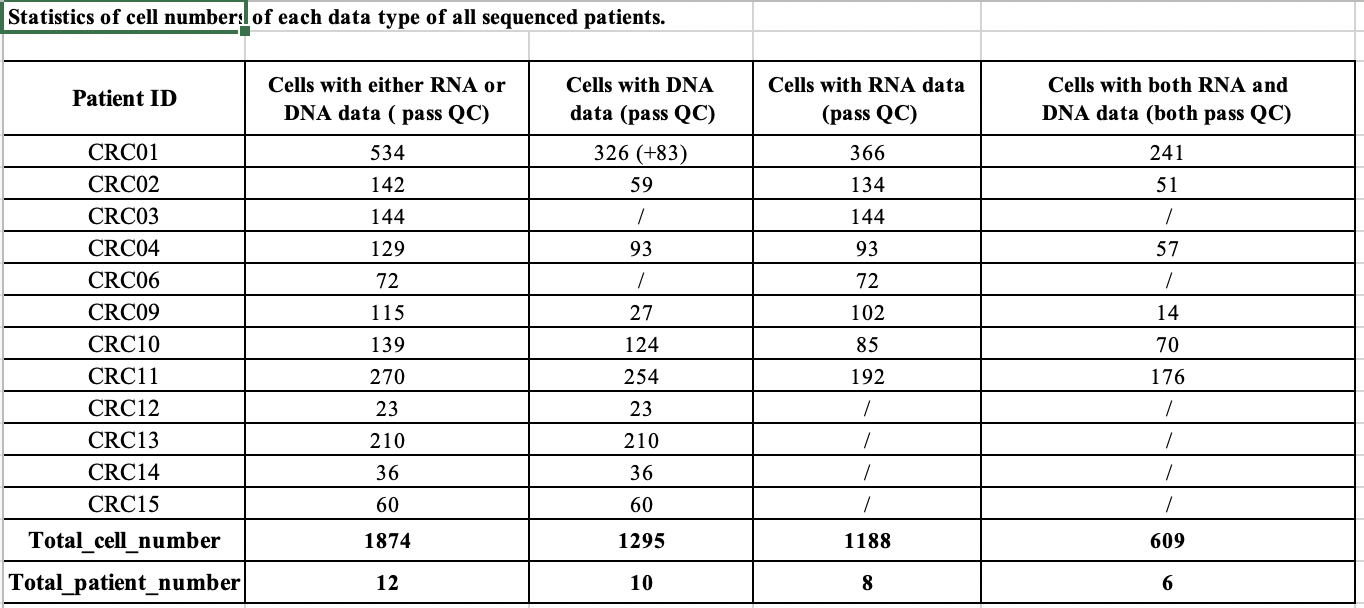
本研究纳入的病人队列
在文章附件有详细描述:
| Patient ID | Gender | Age | AJCC Stage | Primary Site |
| ————— | ——— | —— | ————— | —————— |
| CRC01 | Female | 50 | IV | Left Colon |
| CRC02 | Female | 56 | IV | Right Colon |
| CRC03 | Male | 60 | III | Rectum |
| CRC04 | Female | 71 | III | Right Colon |
| CRC06 | Female | 59 | III | Left Colon |
| CRC09 | Male | 71 | IV | Left Colon |
| CRC10 | Male | 63 | III | Left Colon |
| CRC11 | Female | 51 | III | Left Colon |
| CRC12 | Female | 49 | III | Rectum |
| CRC13 | Male | 51 | III | Left Colon |
| CRC14 | Female | 76 | III | Rectum |
| CRC15 | Female | 79 | IV | Right Colon |
可以看到实验设计还是蛮周全的,每个病人在不同治疗时间点,都取样了,而且每个样品都使用了scTrio-seq技术进行探索。
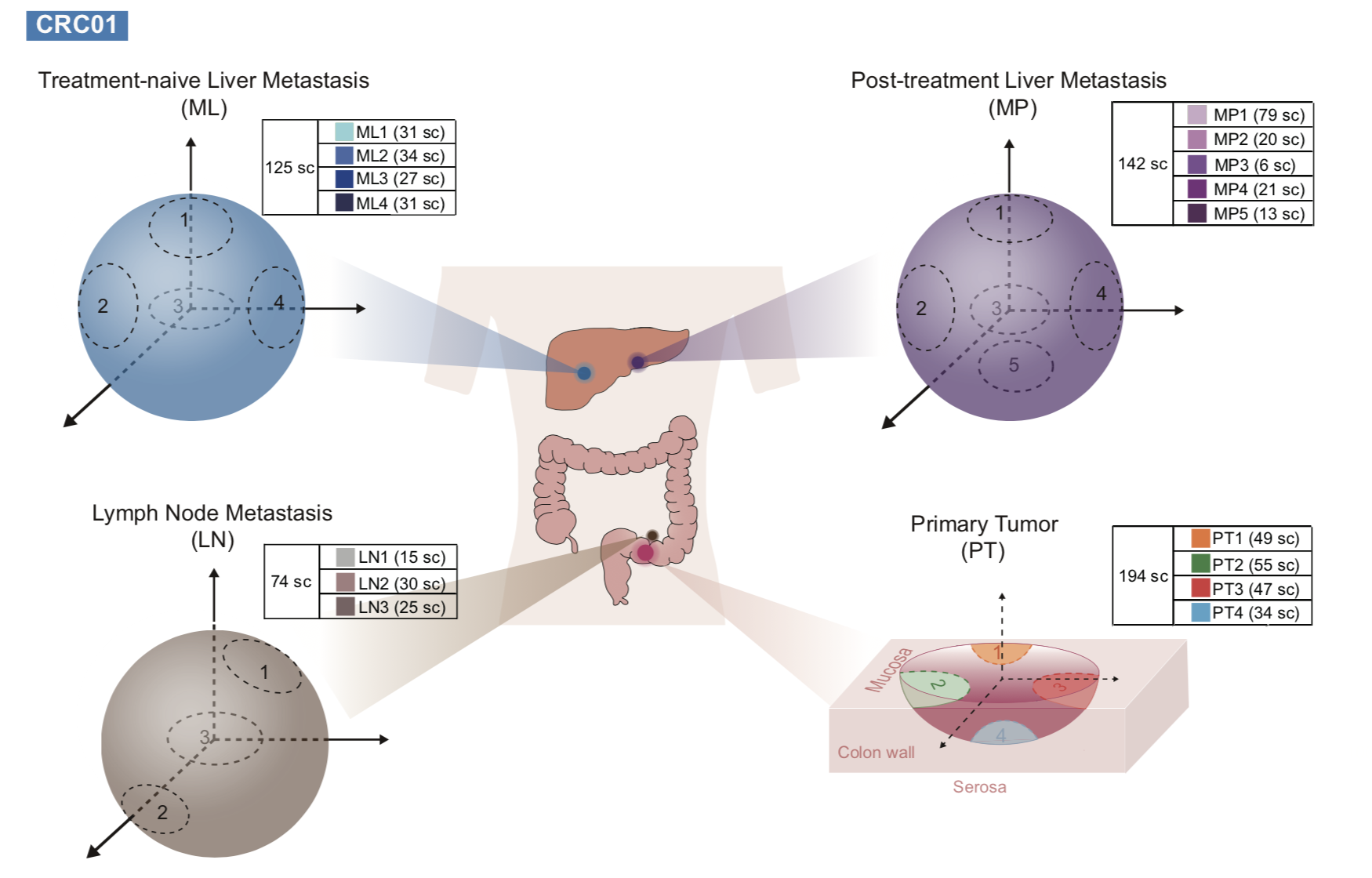
取样的空间策略如下:
所以细胞数量还是蛮可观的,毕竟不是10X那种商业化仪器一次性批量操作几千个细胞。
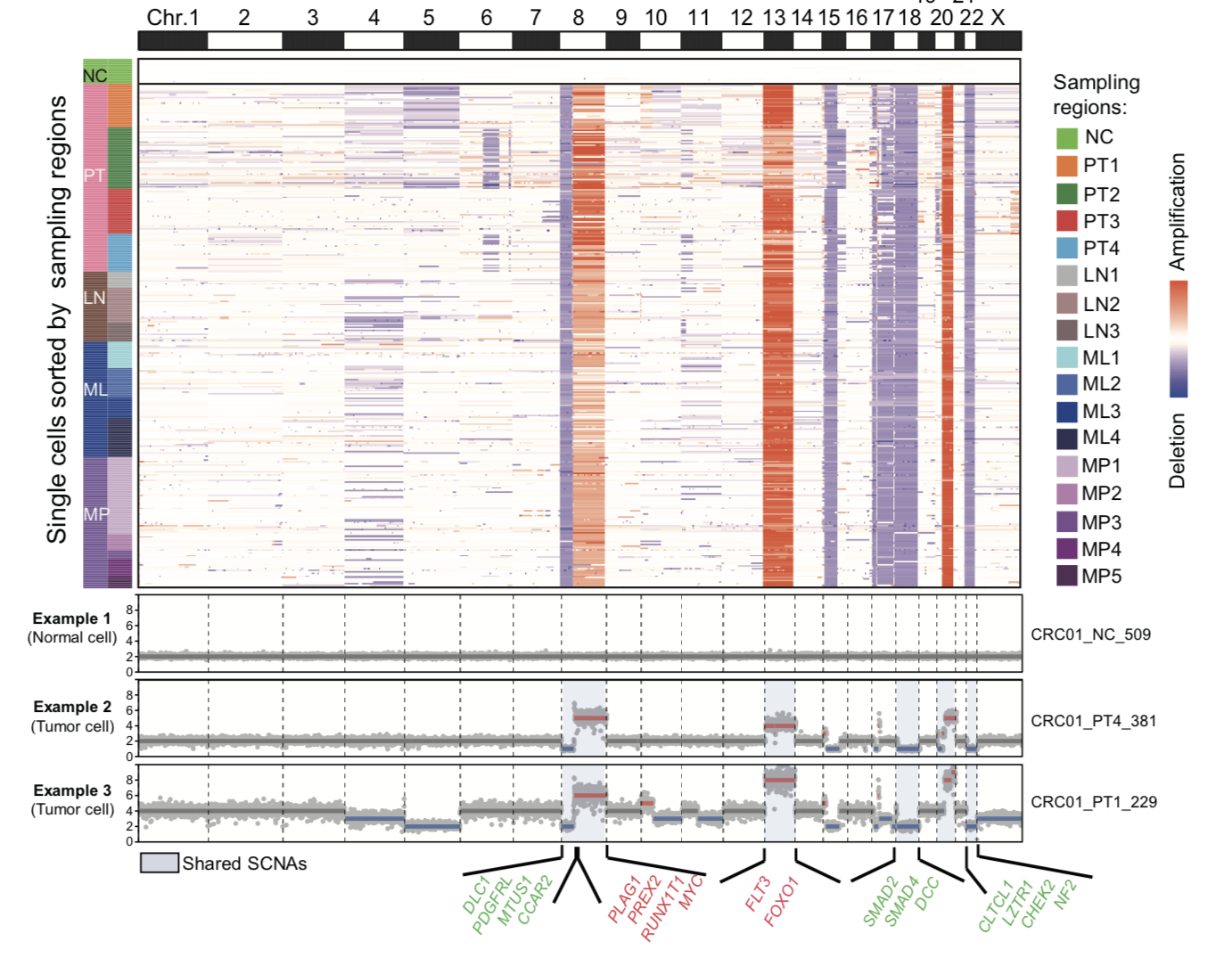
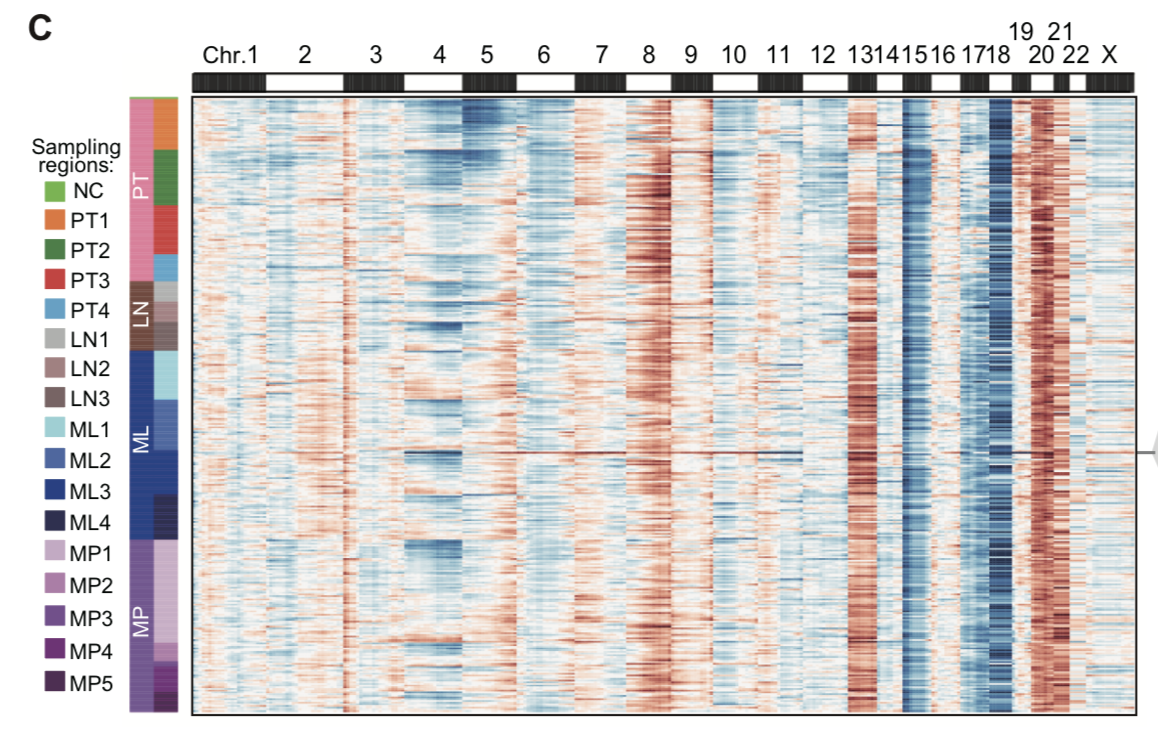
单细胞基因组数据推断拷贝数变异
对每个病人取4个不同时间点样品,每个时间点还选取了不同空间位置的多个细胞,单细胞基因组数据推断拷贝数变异,作者这里使用的是 https://www.nature.com/articles/nmeth.3578 文章里面,找single-cell copy-number variations (CNVs),大家可以试试看,是一个网页工具,非常容易使用。
可以看到,第一个病人的三百多个单细胞基因组数据,表明各个细胞的拷贝数变异非常接近。
单细胞转录组数据推断拷贝数变异
这个主要是基于broad开发的inferCNV软件所代表的算法,具体可以参考我以前在单细胞天地写的教程:
- 单细胞转录组数据分析CNV
- 使用broad出品的inferCNV来对单细胞转录组数据推断CNV信息
可以看到单细胞转录组数据推断的CNV和前面单细胞基因组数据推断的CNV还是很类似的。
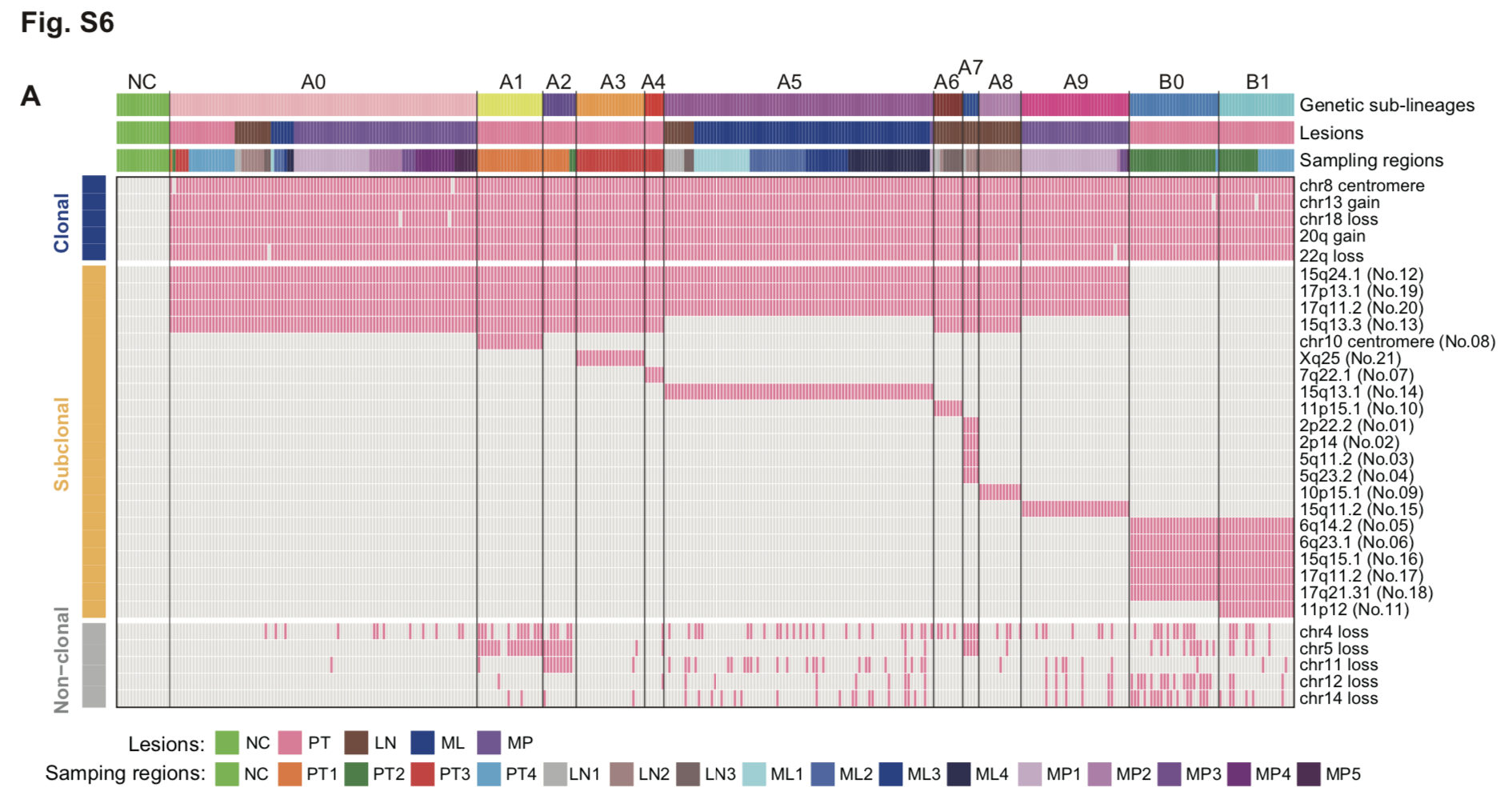
基于拷贝数变异的细胞分群
总共是3种拷贝数变异事件:
- Clonal SCNAs were those shared by nearly all obtained single cancer cells.
- Sub-clonal SCNAs had intra-chromosomal breakpoints and were private to only a subset of the cancer cells.
- Some whole-chromosome-level SCNAs, which occurred frequently among the cancer cells, but were not consistent with genetic lineages, were non-clonal.
可以很清晰的看出来。
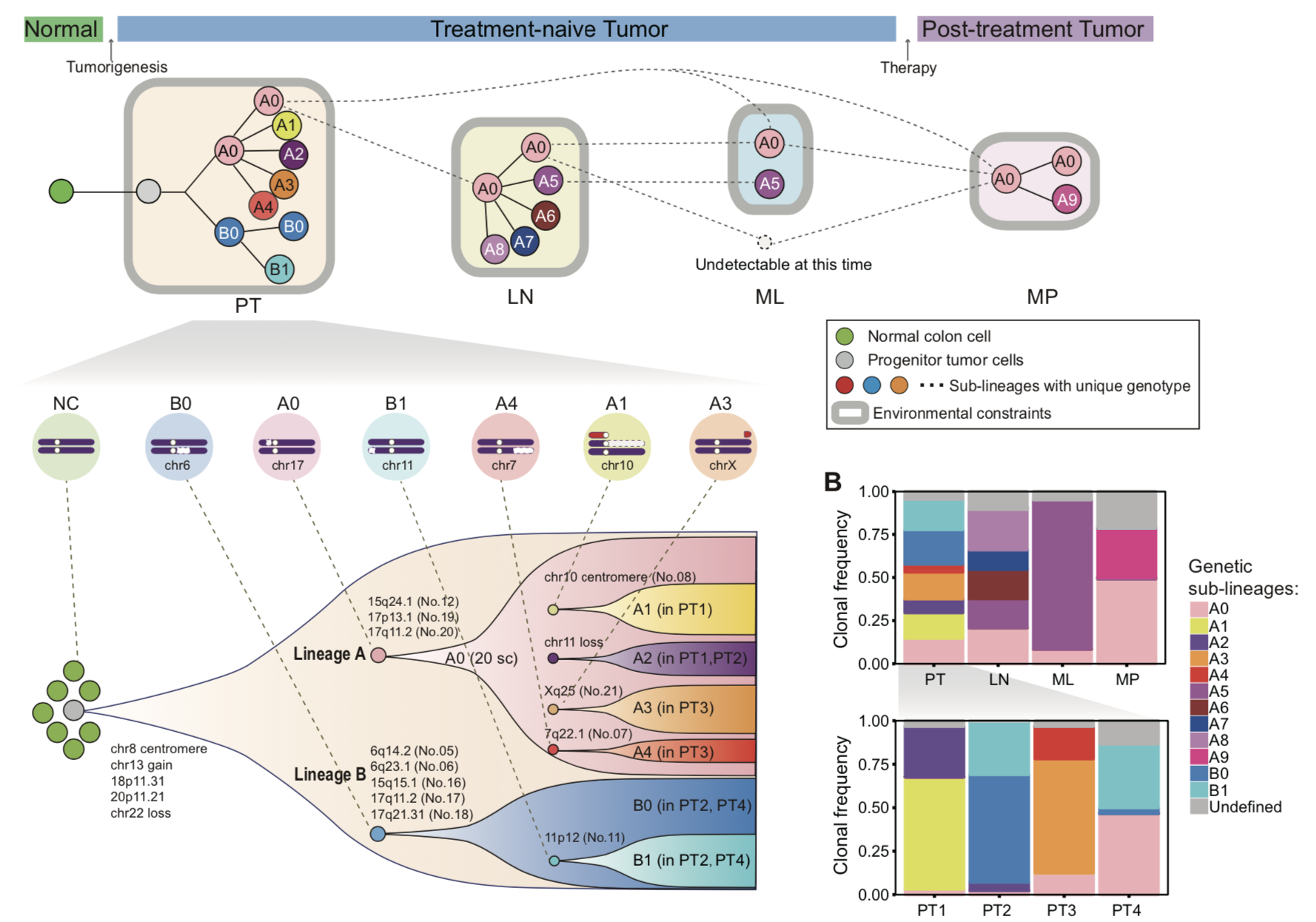
构建肿瘤进化树
本实验设计非常合理,从病人选取,不同疾病进展时期选取,不同空间位置的肿瘤细胞选取上面都有考究,所以可以合理的构建肿瘤进化树。
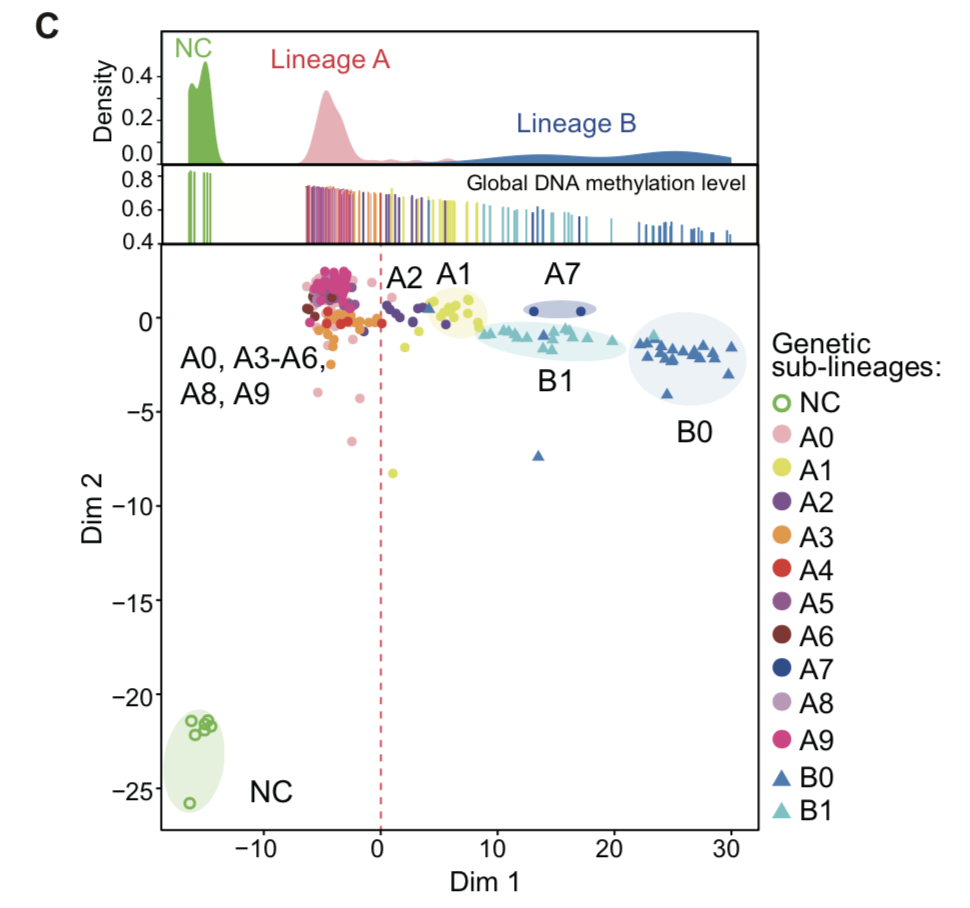
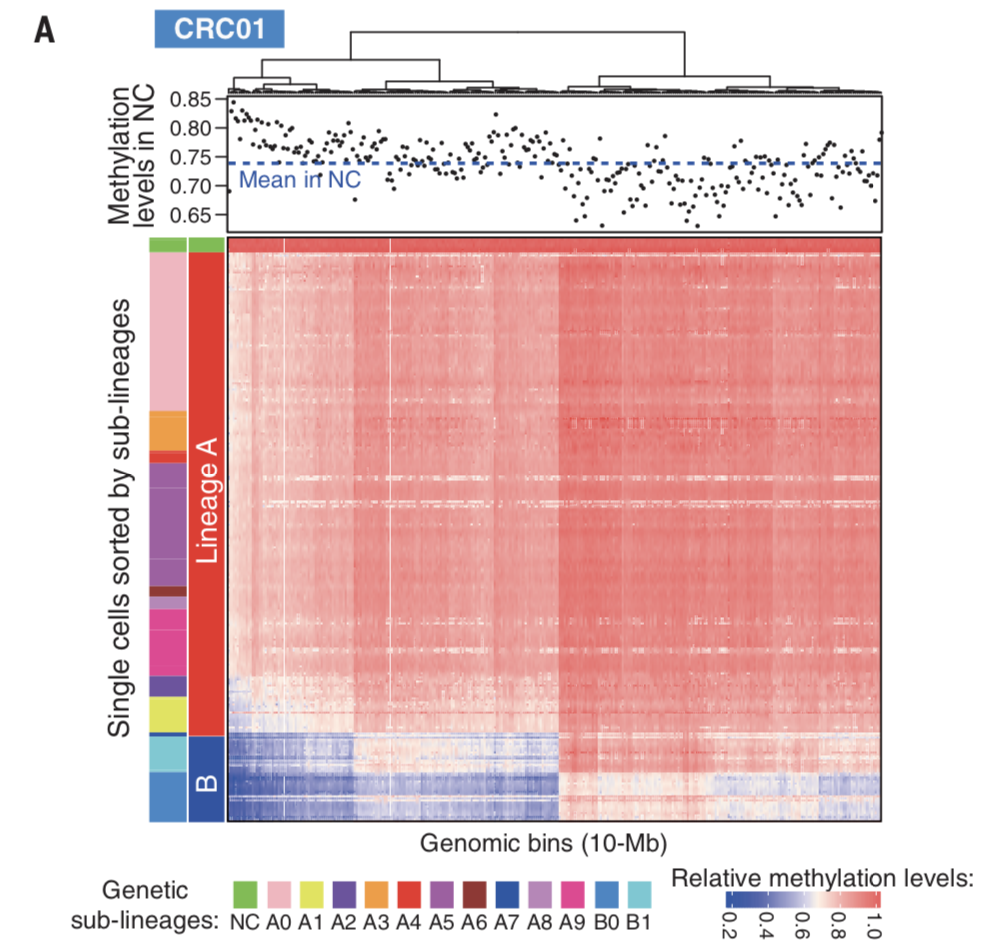
甲基化信号分群
对多个病人的所有细胞使用MDS算法,分辨率是 DNA methylation levels (1-kb tile)
热图展现如下:
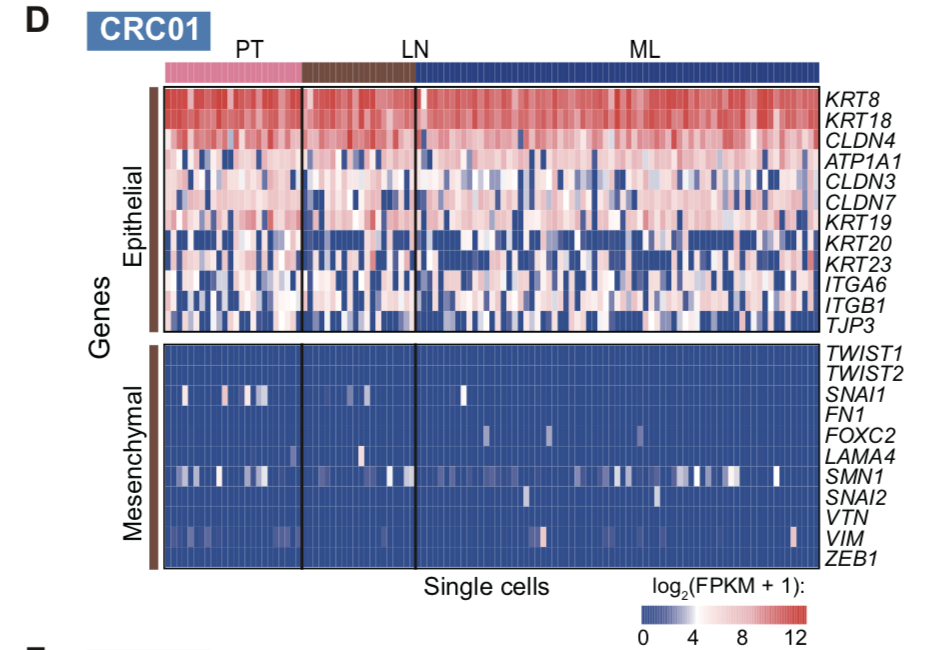
EMT基因的表达量热图
既然是 The EMT-related genes’ expression levels ,同理也可以展现甲基化信号的变化。
后记
因为本文是技术方法,所以并不需要有很强的生物学故事,只需要证明这个技术可靠,有用即可,所以作者对自己技术的数据产生了大量的图表,但是最后的结论是他们开发的单细胞多组学技术,可以辅助探索结直肠癌疾病发生发展和转移过程的分子机制。