为了分析不同类型、组织起源肿瘤的共性、差异以及新课题。TCGA于2012年10月26日-27日在圣克鲁兹,加州举行的会议中发起了泛癌计划。参考:https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6000284/ 为此我也录制了系列视频教程在:TCGA知识图谱视频教程(B站和YouTube直达)
发表于 PLoS Genet. 2018 Sep 的研究,Pan-cancer inference of intra-tumor heterogeneity reveals associations with different forms of genomic instability. 系统性的探索了32种癌症的接近6000个肿瘤病人数据的肿瘤内部异质性情况,值得注意的是作者这里使用PhyloWGS算法的结果来代表肿瘤内部异质性,所有的生物学意义的结论都是基于这个假设。
文献解读属于100篇泛癌研究文献系列,首发于:http://www.bio-info-trainee.com/4132.html
PhyloWGS算法得到的克隆数量
PhyloWGS算法这里就不介绍了,用法很简单,但是需要原始的测序数据,所以这里也没有办法演示,但是作者提供了每个样本的PhyloWGS算法得到的克隆数量数据下载。
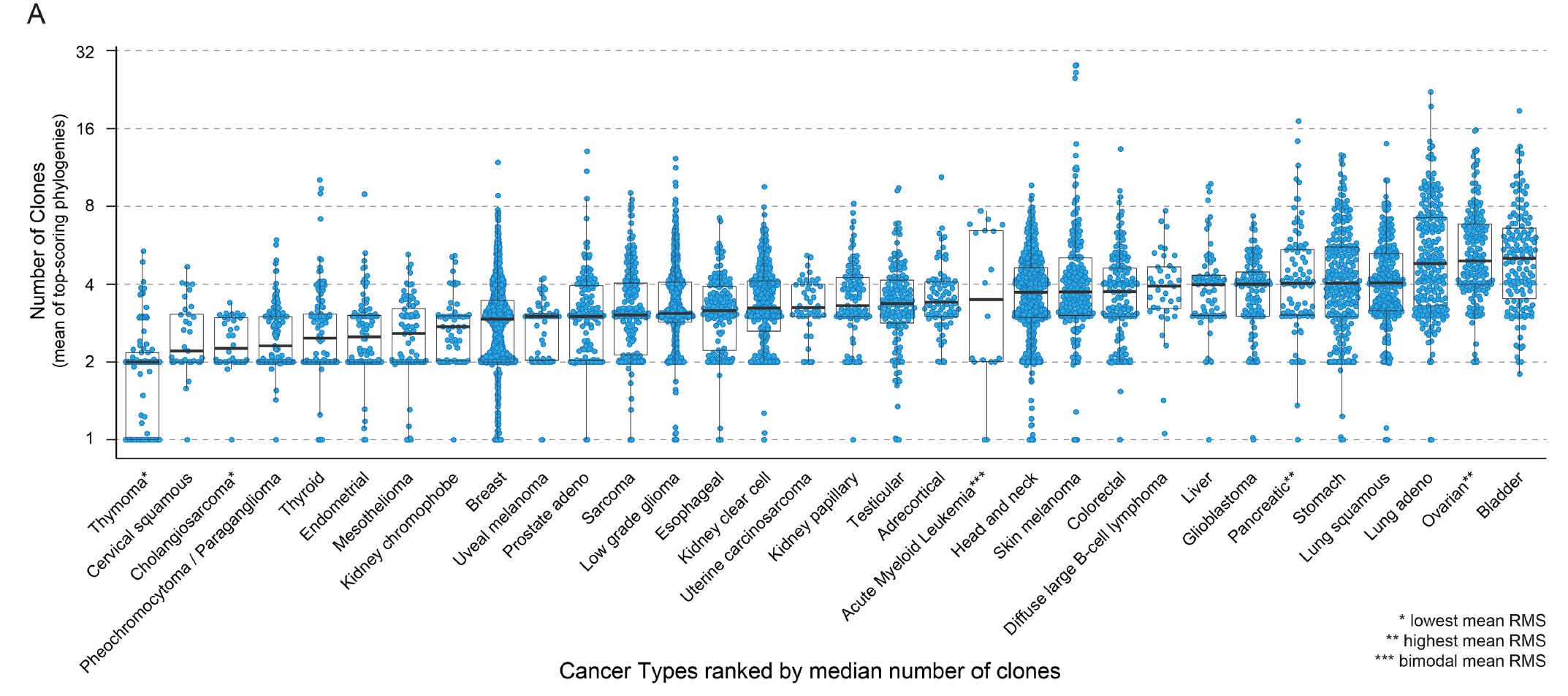
虽然PhyloWGS本来是为WGS测序数据设计的,但是软件作者说明了其在WES数据也可以使用,
可以看到PhyloWGS算法得到的克隆数量这个指标是跟一些因素相关的,包括purity (B) inferred by ABSOLUTE, mean number of reads per mutated sites (RMS) (C), number of mutations (D) and number of copy number altered segments
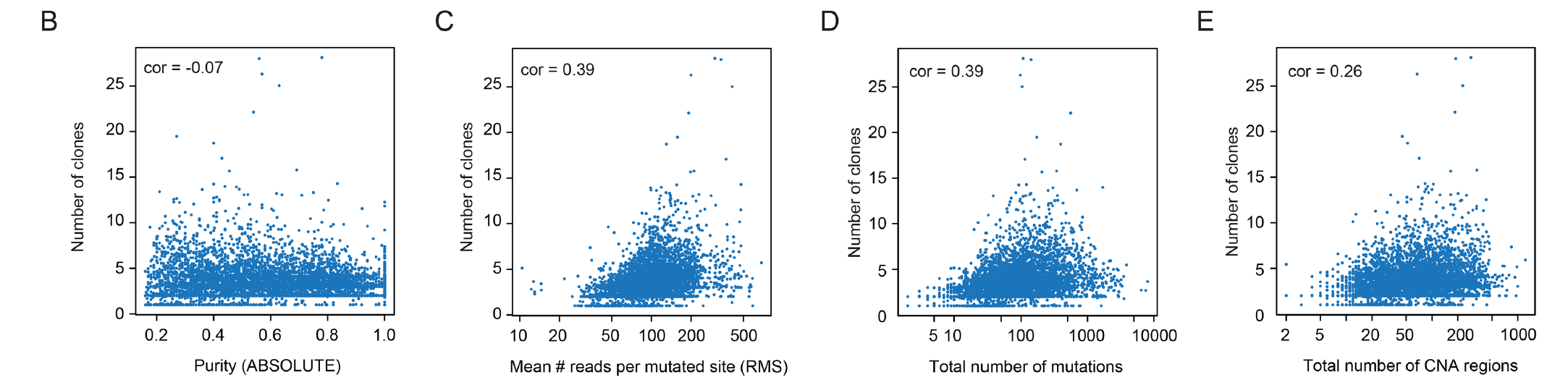
假设PhyloWGS算法得到的克隆数量就代表着肿瘤内部异质性,那么这个肿瘤内部异质性就手动突变数量和拷贝数变化情况的影响。
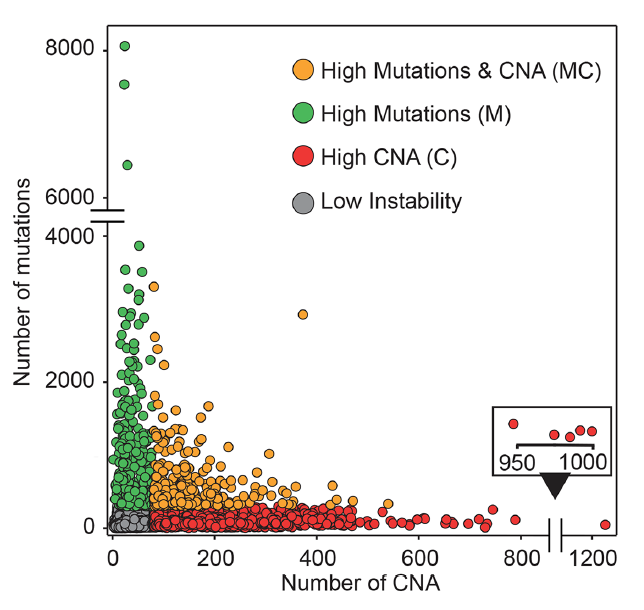
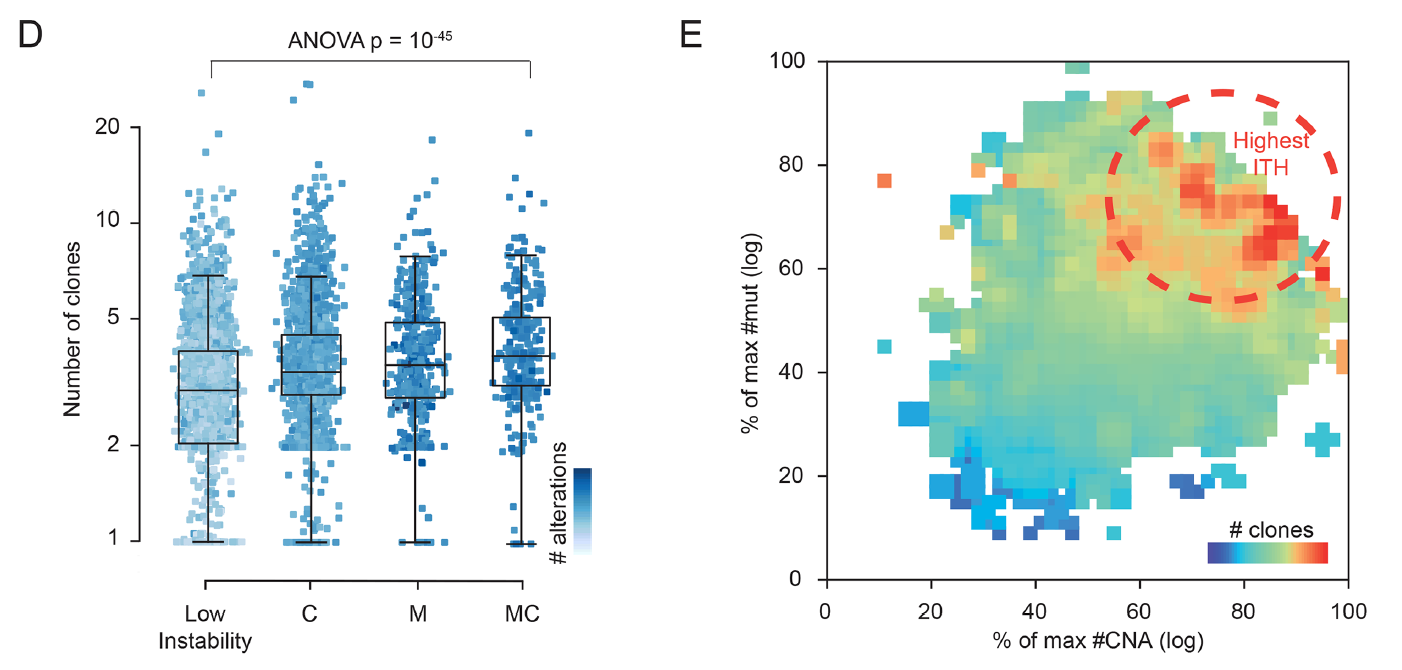
根据突变数量和CNV情况对样本进行分类
通过阈值可以分成4组:
- low numbers of mutations (<300) and CNA (<80) (gray),
- high number ofmutations (>300) and low number of CNA (<80) (M class, green)
- high number of CNA(>80) and low number of mutations (<300) (C class,red),
- high numbers of both mutations (>300) and CNA (>80) (MC class, orange).
它们的相关性如下:
PhyloWGS和EXPANDS的比较
可以看到EXPANDS推断的克隆数量也是在根据突变数量和CNV情况对样本分成的4组是有差异的。
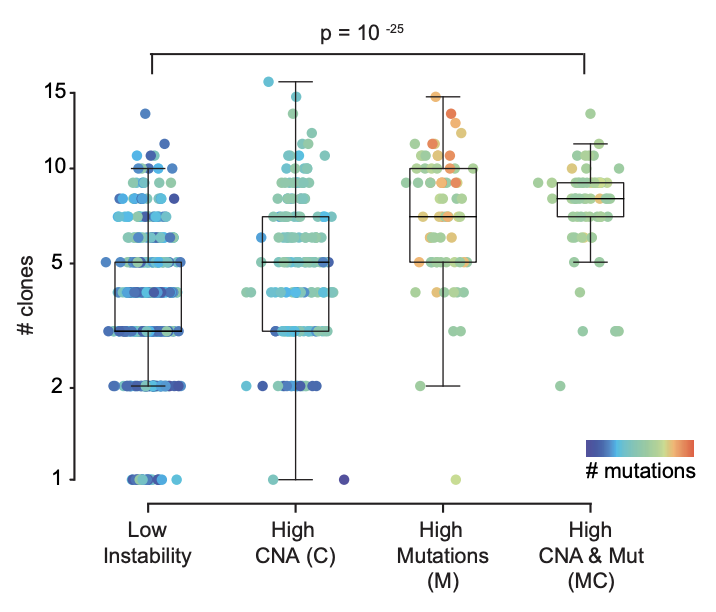
基因组不稳定性影响着肿瘤内部异质性
如下图可以看到,根据突变数量和CNV情况对样本分成4类,它们这4类的肿瘤内部异质性显著差异,排序后可以通过热力图展示。
不同克隆结构代表的异质性
前面是直接使用PhyloWGS算法得到的克隆数量代表肿瘤内部异质性,实际上,即使不同病人都是5个亚克隆, 它们的克隆结构也可以不一样,如下所示:
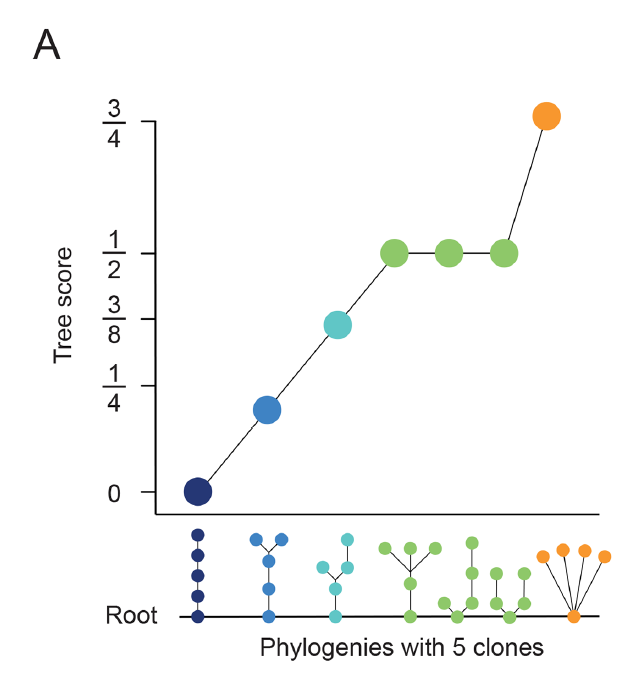
这个时候,需要引入 Tree scores的概念。亚克隆的异质性
这个时候需要对PhyloWGS算法有一定的理解,特别是软件的输出结果的理解。
分析结果下载
S1 Table. Properties of the tumor samples.
- Column 1—Tumor sample name
- Column 2—Tumor type
- Column 3—Tumor subtype
- Column 4—Mean number of reads per mutated site
- Column 5—Number of mutations
- Column 6—Number of copy number altered segments
- Column 7—Top scoring phylogenies mean number of clones
- Column 8—Top scoring phylogenies mean Tree score
- Column 9—Top scoring phylogenies mean number of clonal mutations
- Column 10—Top scoring phylogenies mean number of subclonal mutations
- Column 11—TCGA curated tumor sample purity
- Column 12—ABSOLUTE inferred tumor sample purity.
理论上,使用这个表格的数据,我们可以重现作者的分析结果。后记
从流程图来看,本研究并不复杂,也很容易复现出来, 关键是如何提出还有如何挑选数据集。
本文献解读属于100篇泛癌研究文献系列,首发于:http://www.bio-info-trainee.com/4132.html