看过我TCGA肿瘤数据库知识图谱的小伙伴都只是如何在任意癌症查询指定感兴趣基因的表达量,并且对样本进行分组比较,网站是:https://xenabrowser.net/heatmap/
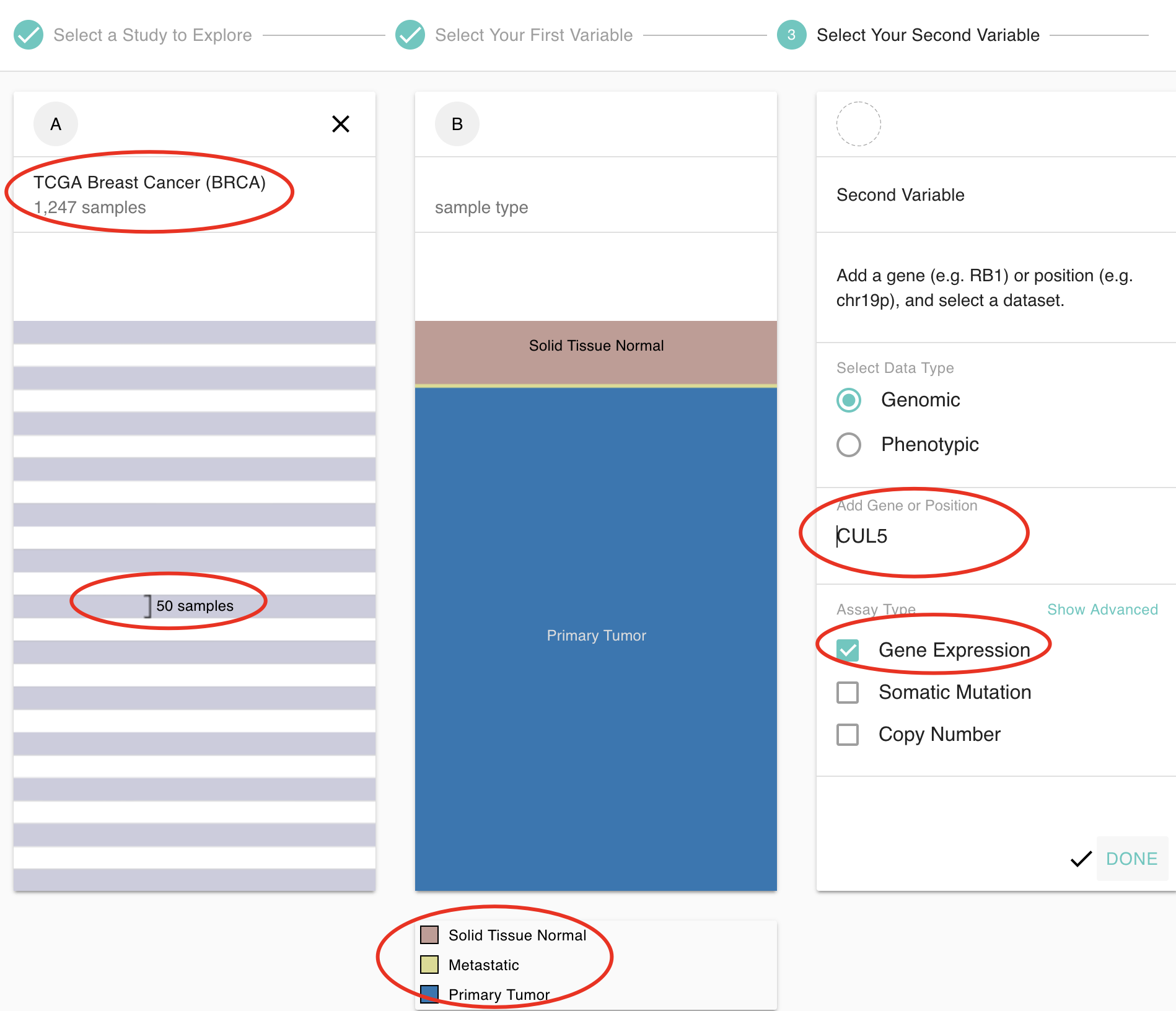
根据视频教程拿到数据,很容易可视化如下:
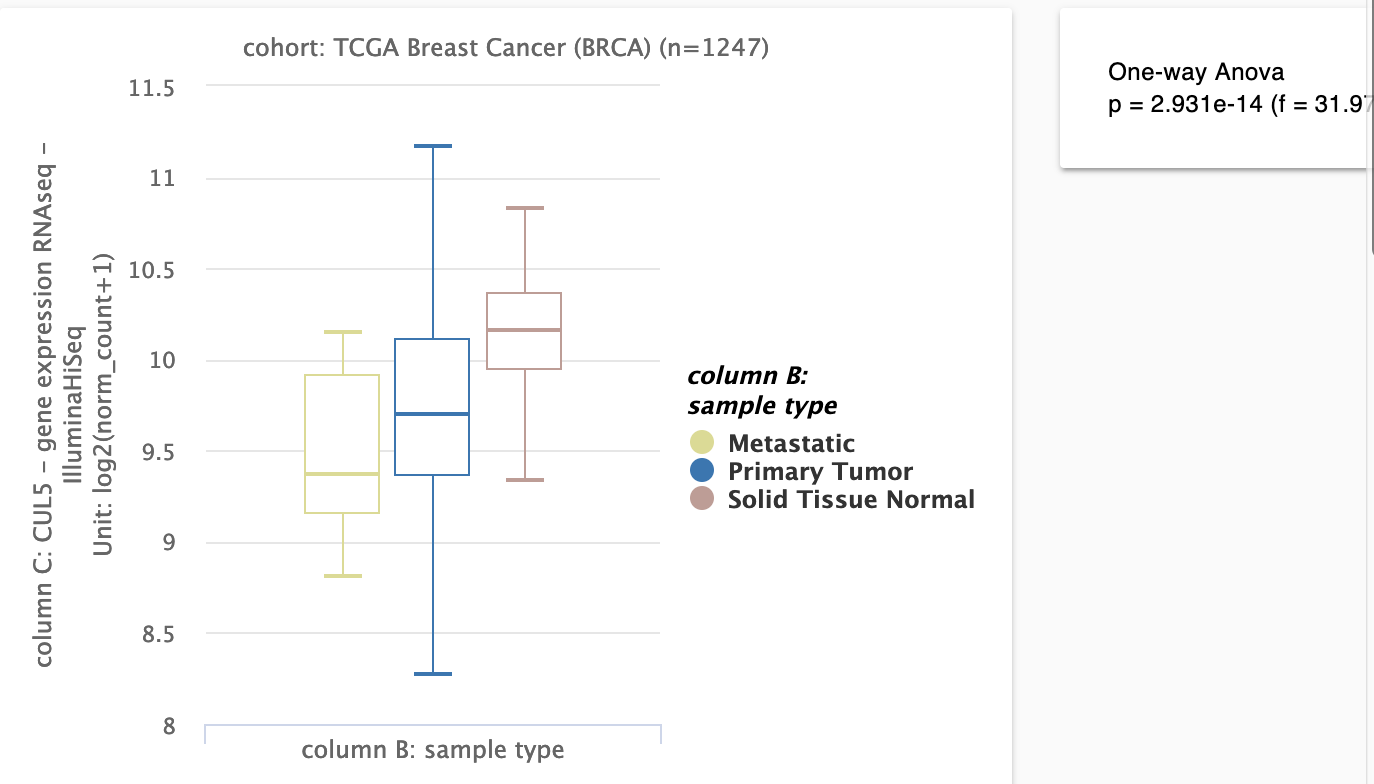
可以得出结论,我们感兴趣的基因(这里是CUL5)在乳腺癌的正常组织及癌症组织(原位和转移)表达量,使用
单因素方差分析,得到了统计学显著的结果。
定义
单因素方差分析是两个样本平均数比较的引伸,它是用来检验多个平均数之间的差异,从而确定因素对试验结果有无显著性影响的一种统计方法。
- 因素:影响研究对象的某一指标、变量。
- 水平:因素变化的各种状态或因素变化所分的等级或组别。
- 单因素试验:考虑的因素只有一个的试验叫单因素试验。
了解数据
数据文件可以下载,然后读入R里面进行可视化,代码如下:
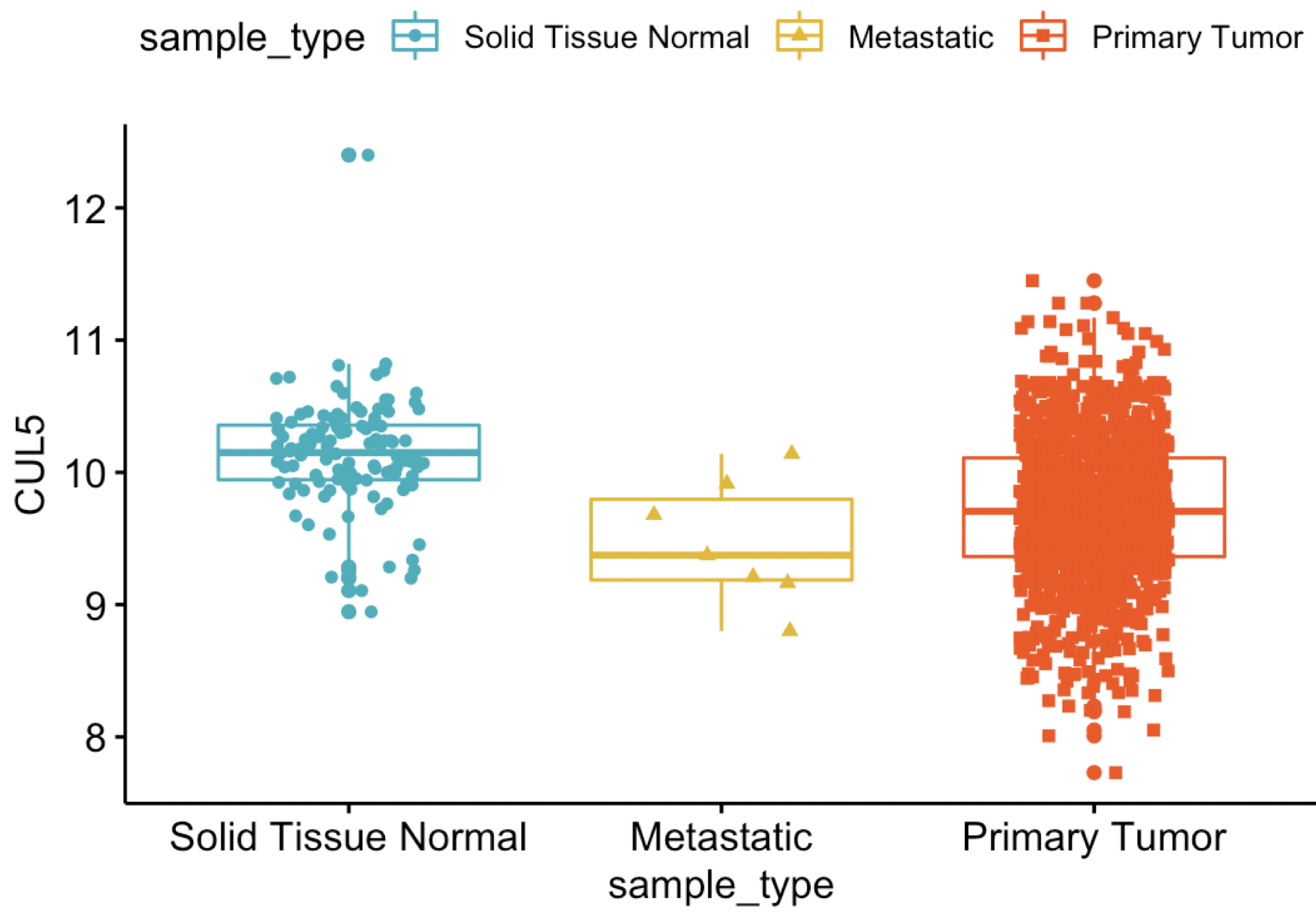
rm(list = ls()) options(stringsAsFactors = F) library(ggpubr) df=read.table('CUL5-BRCA-type.tsv',header = T,sep = '\t') colnames(df) p = ggboxplot(df, "sample_type", "CUL5", color = "sample_type", palette =c("#00AFBB", "#E7B800", "#FC4E07"), add = "jitter", shape = "sample_type") p这个文件
CUL5-BRCA-type.tsv如果你没有看我的TCGA肿瘤数据库知识图谱可能不知道如何下载,可以发邮件给我找我申请这个测试数据 ( 邮箱: jmzeng1314@163.com )
可以看到,比网页工具出图要好看:
但是,这个时候还没有进行统计分析,可以添加的统计学检验包括:
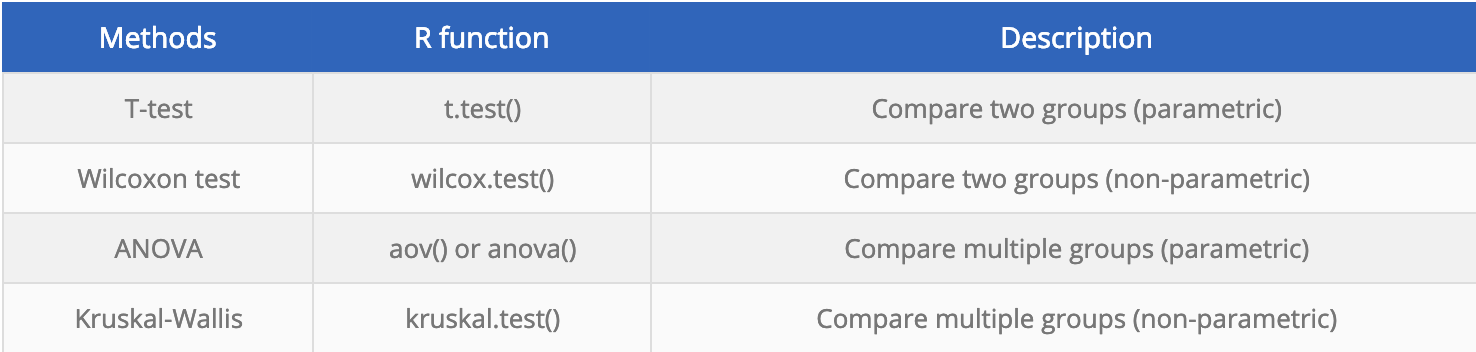
代码也很简单:p+stat_compare_means(method = "anova", label.y = 10)+ # Add global p-value stat_compare_means(label = "p.signif", method = "t.test", ref.group = ".all.") # Pairwise comparison against all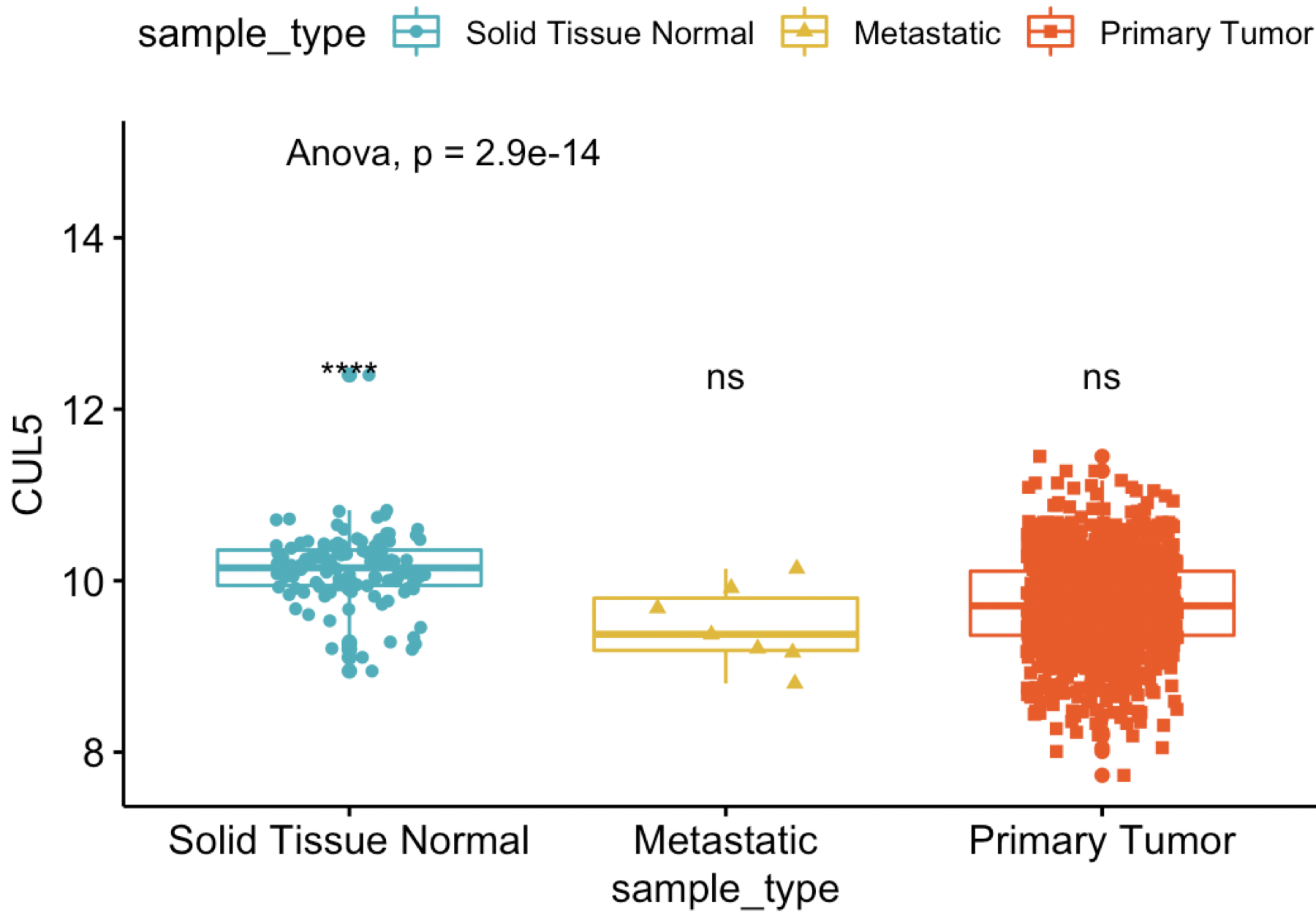
可以看到,跟网页工具结果一模一样,而且出图更漂亮,下面我们就手把手带领大家完成这个分析,把这个ggpubr一步就完成的工作拆解开来。第1步:计算各组内样本均值
lapply(split(df,df$sample_type),function(x) mean(x$CUL5))第2步:计算所有样本均值
mean(df$CUL5 )第3步:计算各组内部误差平方和
tmp=lapply(split(df,df$sample_type),function(x) sum((x$CUL5-mean(x$CUL5))^2) ) sse = sum(unlist(tmp))第4步:计算各组间误差平方和
tmp=lapply(split(df,df$sample_type),function(x) nrow(x)*(mean(x$CUL5) - mean(df$CUL5 ))^2 ) ssb = sum(unlist(tmp))第5步:计算各组内部均方误
mse=sse/(nrow(df)-length(unique(df$sample_type)))第6步:计算组间均方误
msd=ssb/length(unique(df$sample_type))-1第7步:计算F比率
f= msb/mse f第8步:查找F临界值
df1=(length(unique(df$sample_type))-1) df2=(nrow(df)-length(unique(df$sample_type))) qf(0.05,2,1215)差别可知这里的F值是0.05,远小于我们真实情况,所以非常显著了。
第9步:判断是否显著
1-pf(f,2,1215)现在我们已经知道了,在选定的显著水平为0.05时候,这个F统计是显著的,但是仍然是不知道哪组之间不一样, 所以可以选择tukey检验
第10步:进行tukey检验,多重比较
J·W·图凯(Tukey)于1953年提出一种能将所有各对平均值同时比较的方法,这种方法现在已被广泛采用,一般称之为“HSD检验法”,或称“W法”。 Tukey (John Wilder Tukey) for multiple comparisons
主要应用于3组或以上的多重比较。比如说一共有4组数据,两两比较产生6个统计值,Tukey test用于生成一个critical value来控制总体误差(Familywise error rate,FER);与Tukey test相类似的是Dunnett test,它是控制多对一比较(即3组同时和一个参照组比较)的FER。
这个多重比较算法还蛮多的,参考:https://zhuanlan.zhihu.com/p/44880434