是深圳华大基因牵头的10X单细胞转录组测序项目,本研究关注的是乳腺癌病人的肿瘤微环境 的细胞类型,尤其是免疫细胞如何作用于癌症细胞。尤其关心那些与肿瘤病人预后或者治疗情况相关的肿瘤浸润性淋巴细胞,T细胞功能障碍(也被称作耗竭)在癌症中很常见,为了更好地理解T细胞功能障碍,通常需要分析肿瘤浸润性淋巴细胞(tumor-infiltrating lymphocyte, TIL),这个时候单细胞转录组技术就派上用场了。
肿瘤浸润T细胞疗法
肿瘤浸润性淋巴细胞(tumor infiltrating lymphocyte, TIL)指的是从肿瘤组织中分离出的浸润淋巴细胞,1986年,Rosenberg研究组首先报道了TIL细胞。TIL细胞表型具有异质性。一般来说,在TIL中,绝大多数细胞是CD3阳性的。在不同肿瘤来源的TIL细胞中,CD4+ T细胞、CD8+ T细胞的比例有差异,但大多数情况下以CD8+T细胞为主。
作为一种针对实体瘤的免疫疗法,它的基本机制很简单:从患者身上分离一定量的新鲜肿瘤组织,这里面往往存在着一定量浸润但“休眠”状态的T细胞,当然还有带有突变的癌细胞,以一定方式在体外筛选并扩增出对肿瘤细胞有特异杀伤性的T细胞,然后以几百亿的数量级回输到患者体内。最神奇的结果就是,如果回输的T细胞具有理想的高特异性和足够的持续性,基本所有肿瘤病灶都会得到(可能长期的)控制或缩减。
纳入病人
本次单细胞转录组纳入了 15个病人,总共五万多单细胞,是商业的10x技术。
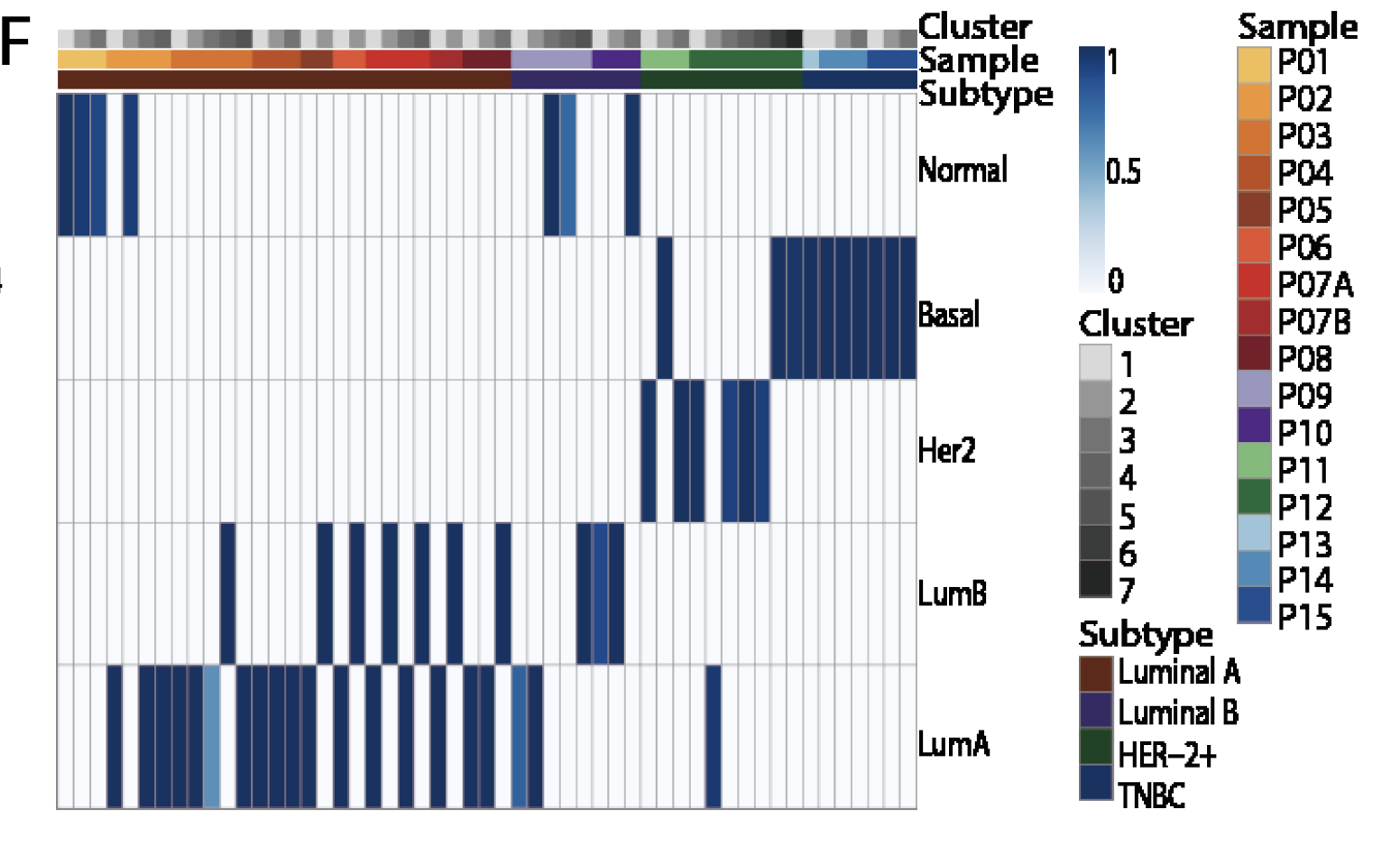
- luminal A (P01, 5 P02, P03, P04, P05, P06, P07A, P07B and P08)
- luminal B (P09 and P10)
- HER2+ (P11 and P12)
- TNBC (P13, P14 and P15).
单细胞建库是:Chromium Single-Cell Instrument (10X Genomics) to generate single-cell gel bead-in-emulsions (GEMs). 有趣的是测序阶段使用的是 BGIS EQ-500 。 同时做了少量的wgs和wes数据,使用的也是 BGIseq Human Exome V4 Kit (BGI) 自己的wes捕获芯片。首先区分是否是上皮细胞
直接使用 Seurat 进行分类即可,可以区分上皮细胞和非上皮细胞,这里研究者主要关心的当然是那些非上皮细胞,也就是肿瘤微环境的那些免疫细胞。
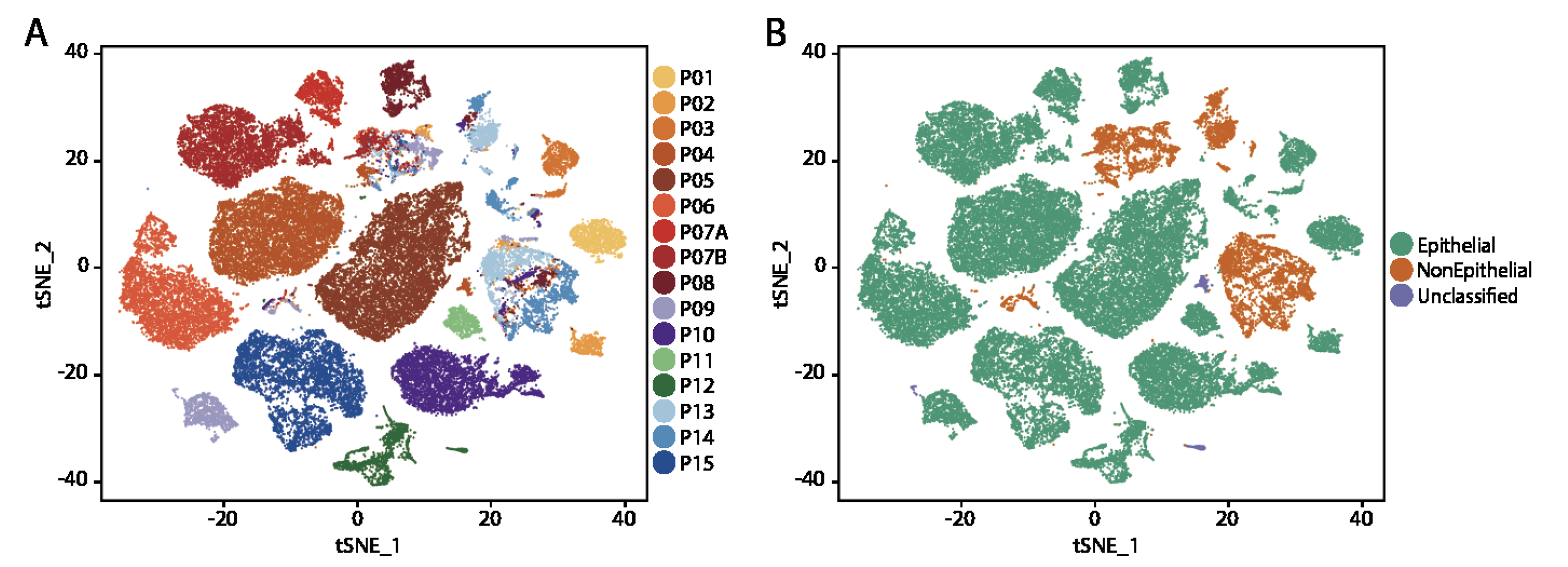
免疫细胞和CAFs的分群情况
可以比较容易区分成有生物学意义的细胞类型
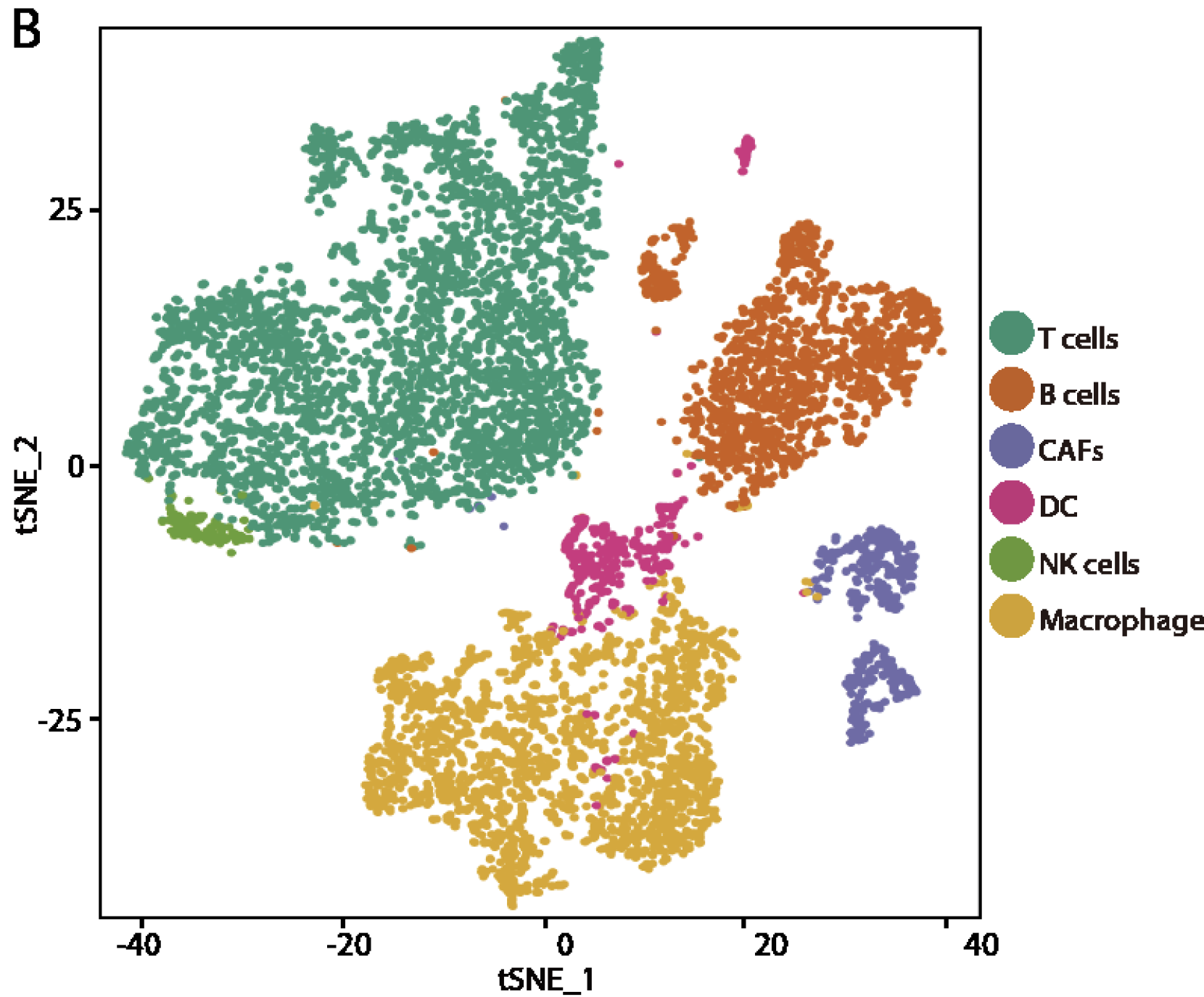
当然,上面的分类太粗犷了,更加细致的分类是: - 9 clusters of T cells
- 5 clusters of TAMs
- 5 clusters of B cells
- 2 clusters of DCs
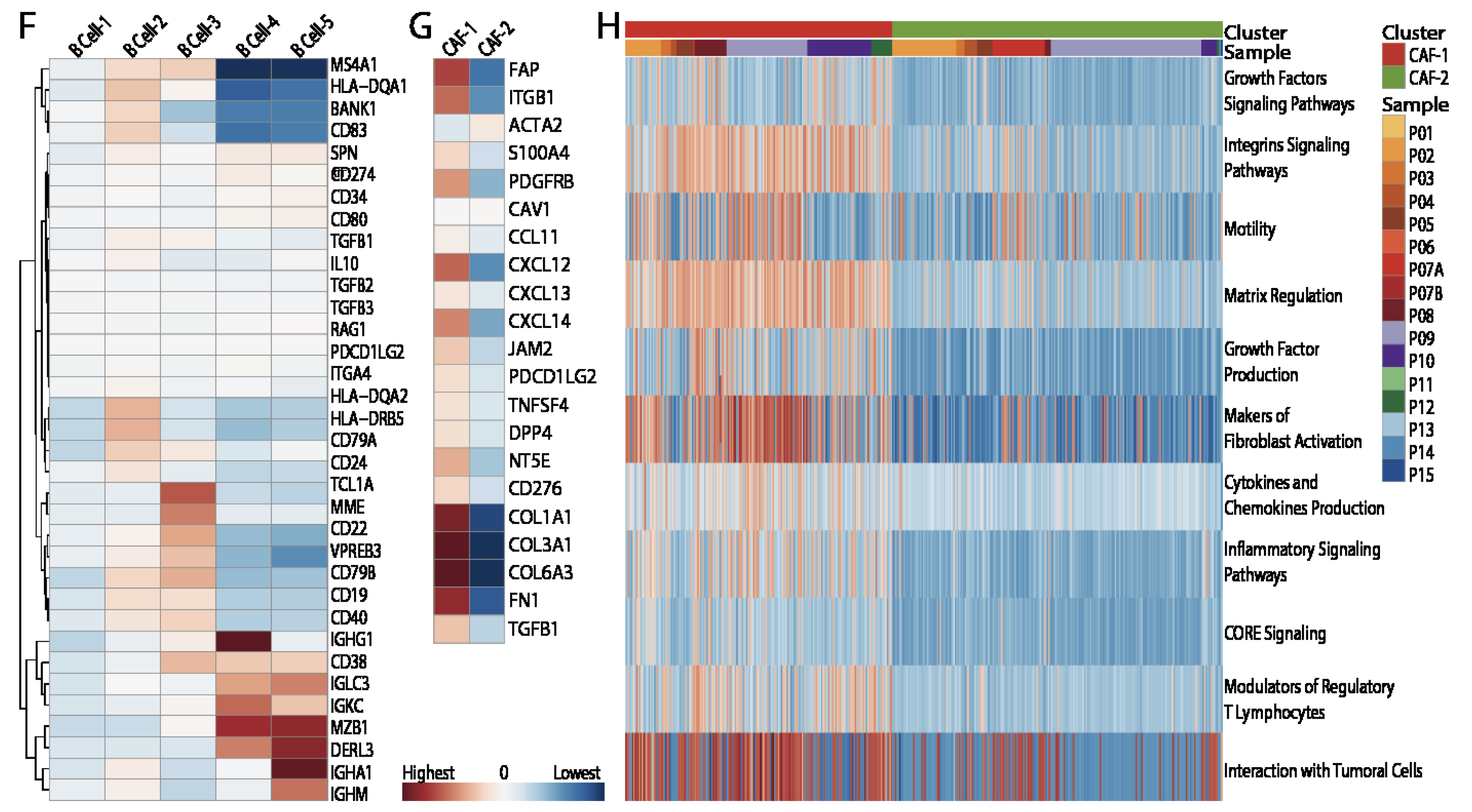
- 2 clusters of CAFs
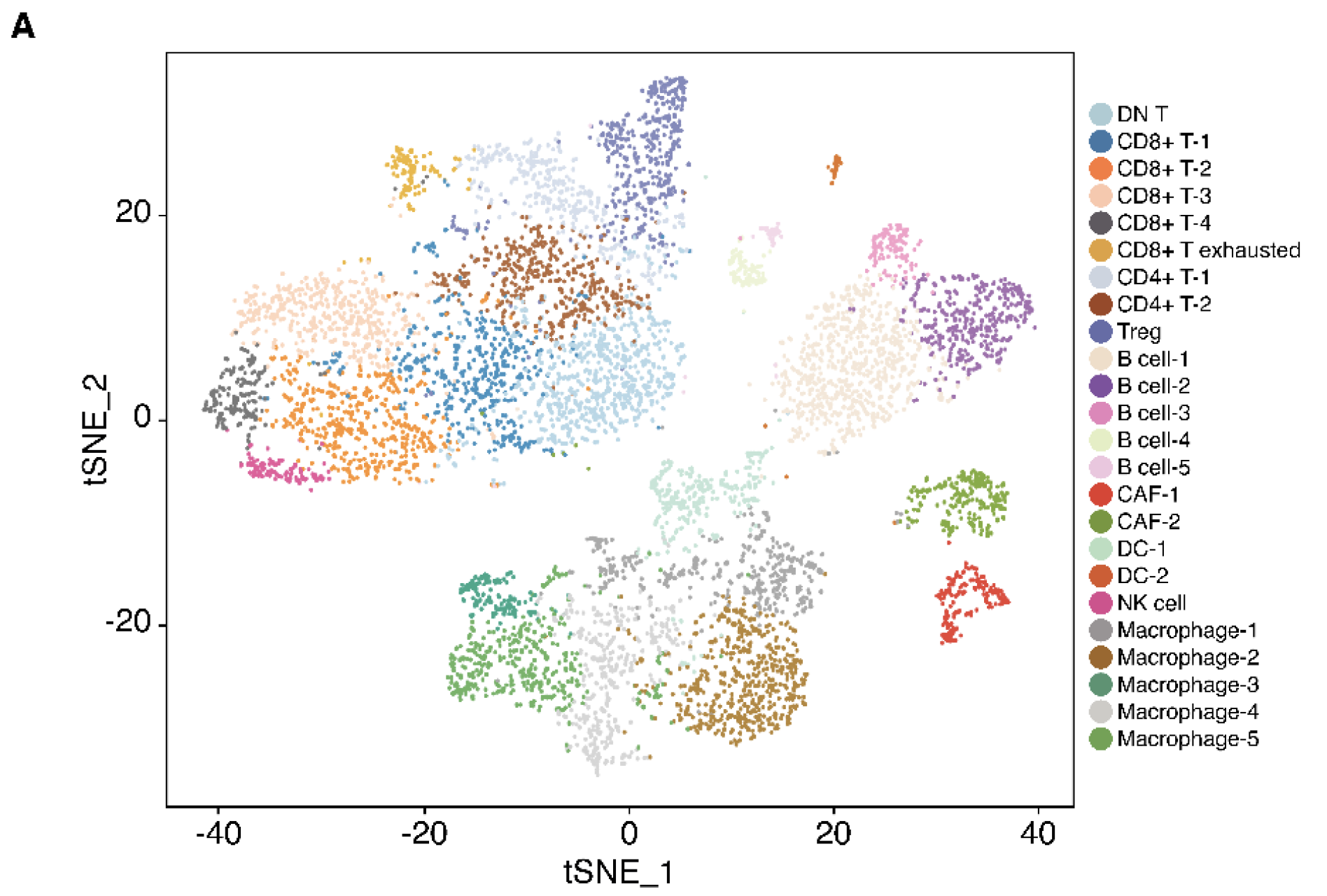
每一群细胞都可以去仔细解读,看看是哪些有意义的生物学基因集在背后。
而且如果以病人来上颜色,可以看到是

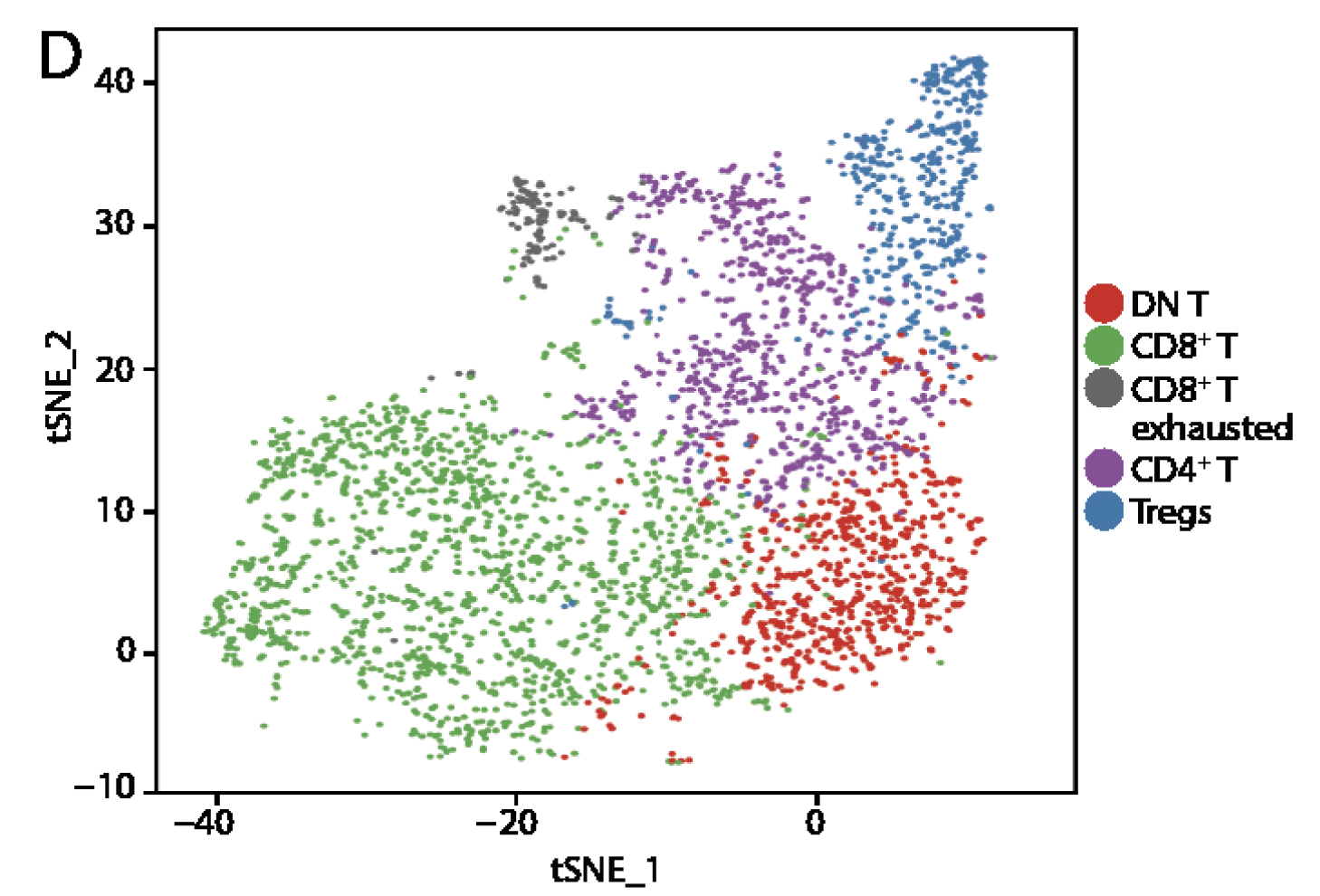
根据生物学marker把T细胞进行细分
可以分成:
- CD4+ T cells
- regulatory T cells (Tregs)
- CD8+ T cells
- CD8+ exhausted T cells
如图,这个时候它们就没有前面细胞类型那样的差异明显了。
上皮细胞根据CNV情况分成恶性与否
对单细胞转录组计算全局CNV我已经多次讲解了,很容易区分恶性细胞与否,不过之前的数据都是在SMART-SEQ2这样的覆盖度尚可的单细胞转录组,第一次看到在10X的单细胞转录组做这个分析。

肿瘤细胞依然是不同的病人自成一类
这个问题不管是在哪个单细胞转录组文章都存在,就是不同病人的肿瘤细胞的单细胞转录组视频的表达量都是没办法聚在一起的,每个病人的肿瘤细胞都是自己跟自己聚在一起。
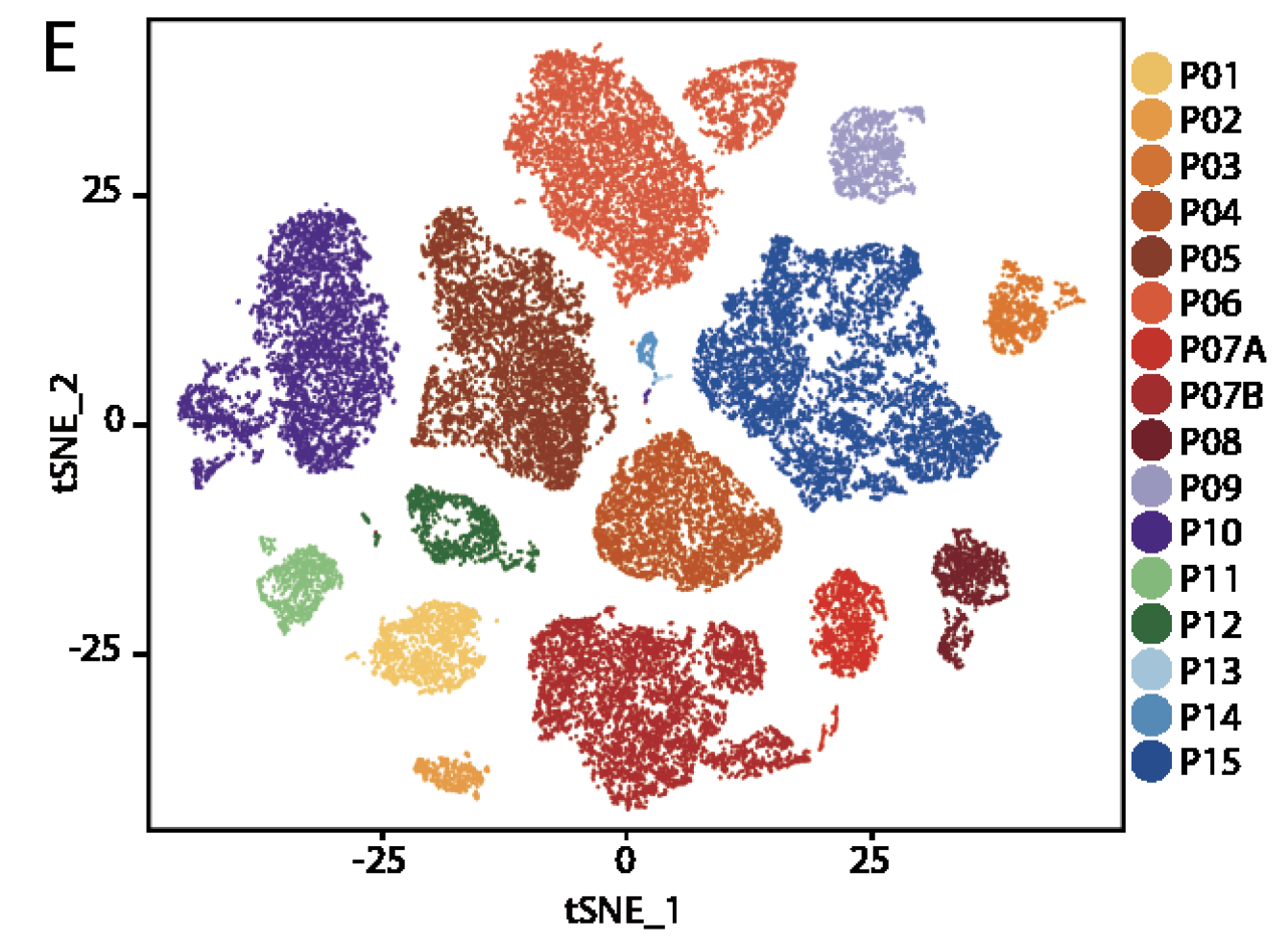
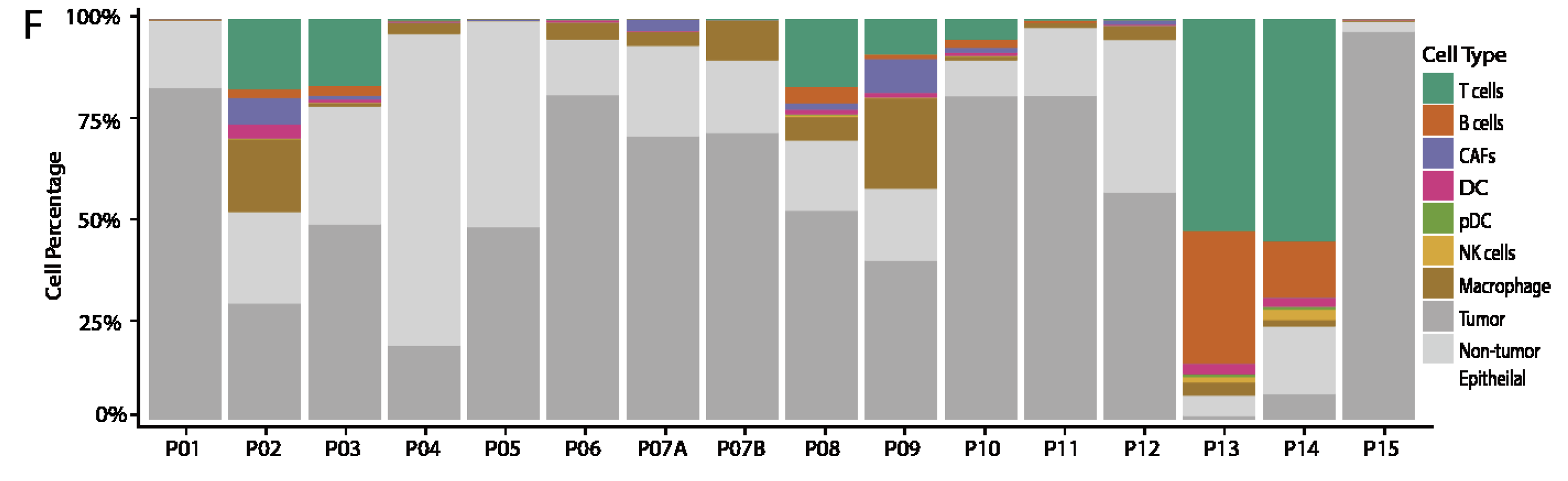
每个病人的不同类型细胞比例不一样
可以看到异质性很高,总体上来说,TNBC组的两个病人的TILs比例非常高,而且以T,B细胞为主,而luminal B组的macrophage细胞含量很高。
每个肿瘤病人的肿瘤细胞异质性非常高
前面说到了不同病人的肿瘤细胞无法聚在一起,只能是按照不同病人来分开,但是同一个病人的肿瘤细胞其实内部异质性也很高,如下:
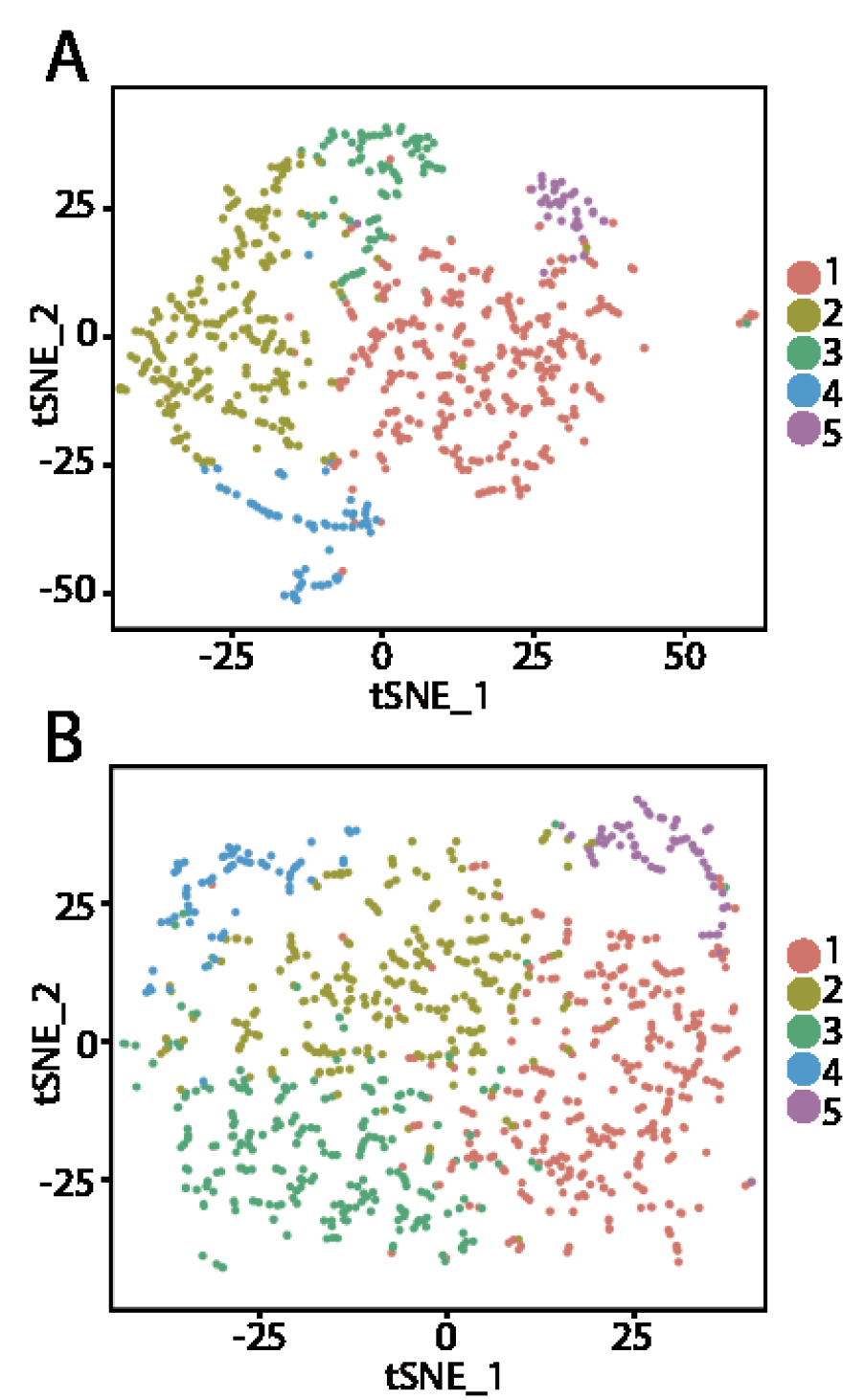
这两个病人的细胞仍然是可以进行分组,只不过是没有确切标准。病人体内的不同组别的肿瘤细胞的差异表达情况
既然可以强行分组,就可以强行找差异表达基因,进行热图可视化:
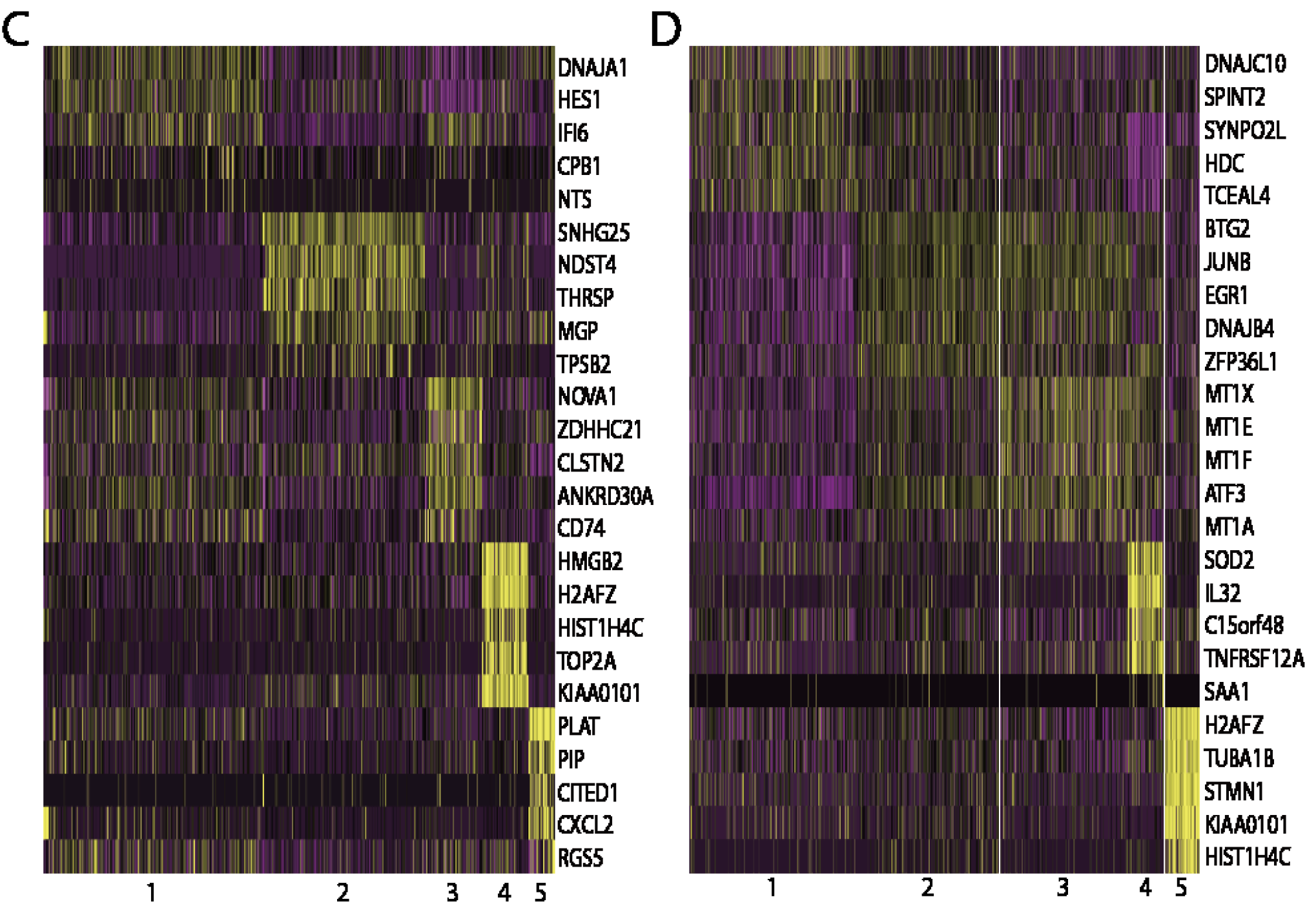
不同病人的基因特征不一样
这个时候需要一定的生物学背景知识来挑选感兴趣的基因集,使用GSVA算法即可,15个病人的,每个病人的肿瘤细胞都可以分成1~5组,最后算出每个组别的GSVA值,热图如下。
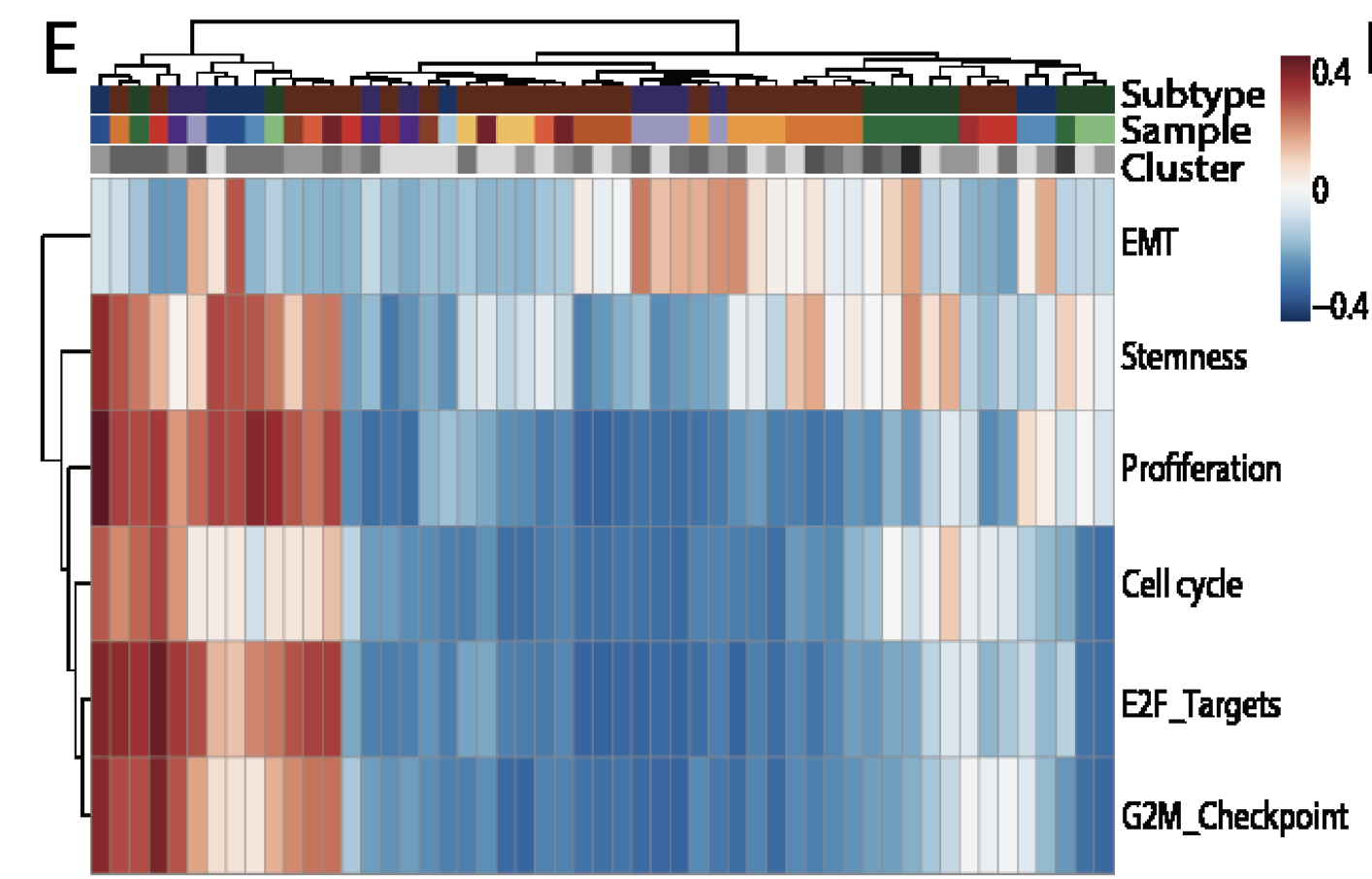
不同病人的PAM50分类异质性也有
可以看到6个luminal A的病人里面的细胞被PAM50分类器归到了LUMINAL B组。
两种不同功能的CAFs
第一组的CAFs可能是跟乳腺癌的肿瘤免疫侵袭有关系。
后记
本文因为是丢在了预印本,所以并不是正式的文章写作,所以都没有discussion部分,只是最后一段提到了作者的研究揭示了乳腺癌病人的肿瘤细胞是如何与肿瘤相关的巨噬细胞和肿瘤细胞的成纤维细胞相互作用,从而逃脱了免疫细胞对肿瘤细胞的清除。
总体上来说,从实验设计上面和分析层面都没有出彩之处,鉴于乳腺癌领域的单细胞文章已经如此之多,这个从数据量角度来说是CNS级别的文章,现在前途堪忧。