看自己感兴趣的基因在自己研究的癌症的预后相关性是高频需求,其实就是拿到基因在癌症病人的表达信息,然后就可以根据表达量高低对病人进行分组,最后这个分组是否统计学显著的把病人的生存情况区分开来。
但是我没有想到,同样的基因在同样的癌症的生存分析结果,在不同的网页工具里面居然是千差万别。
oncoln
首先我们看看http://www.oncolnc.org
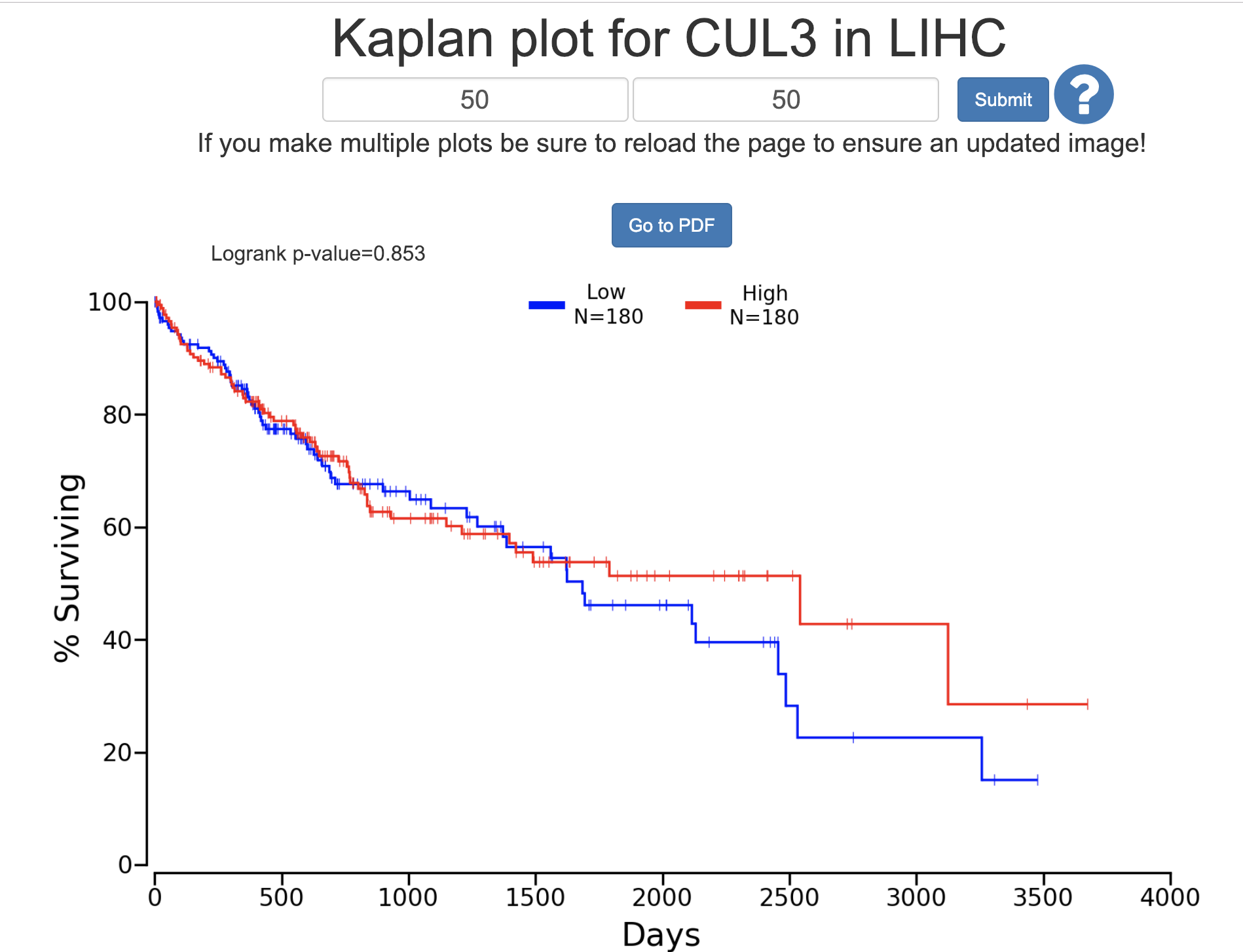
很明显这个基因在这个癌症,如果是按照表达量分成高低两个组别, 那么生存分析是不显著的。
kmplot
但是我们再看看 http://kmplot.com/analysis
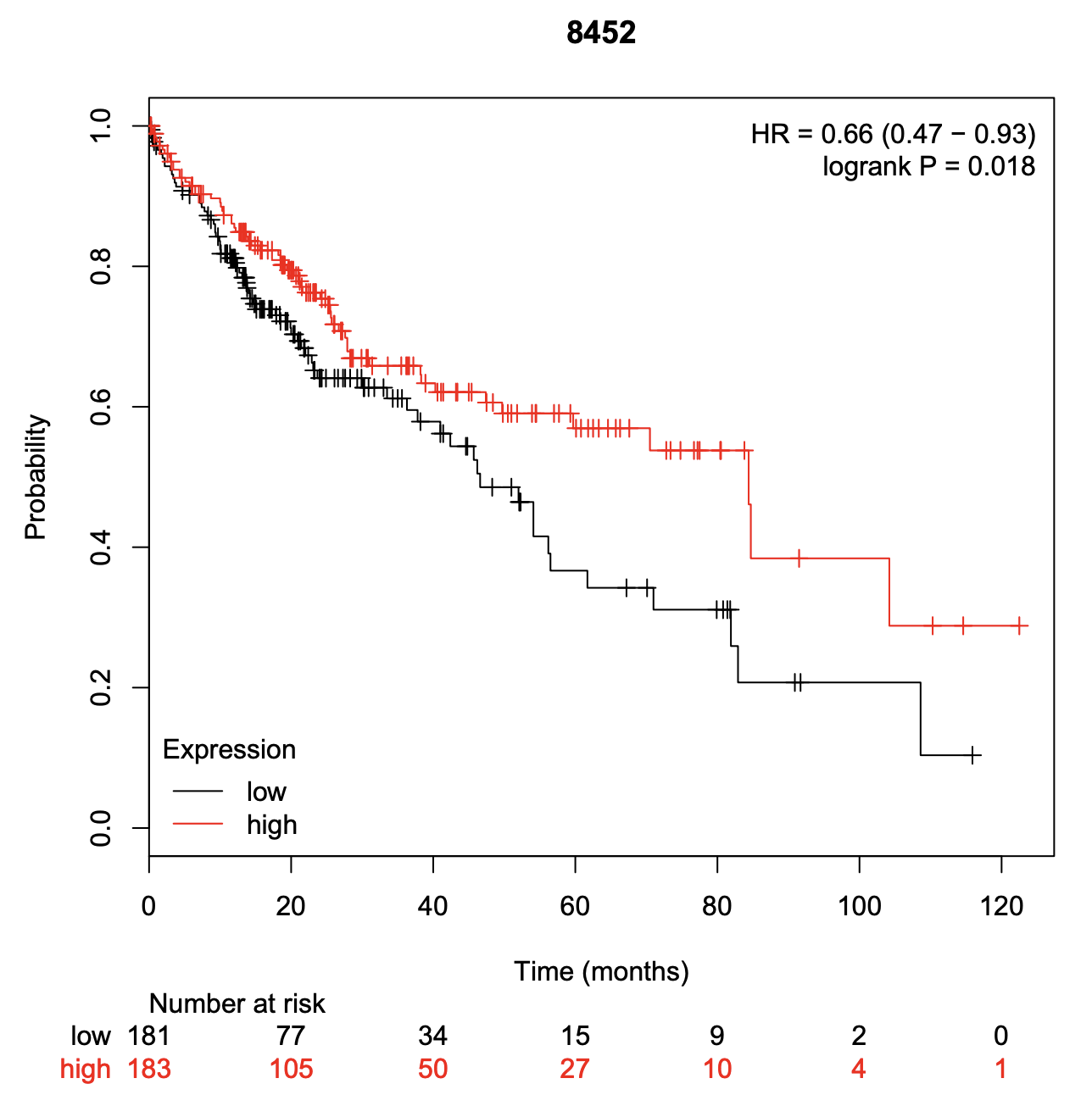
神奇的事情发生了,同样是按照表达量分成高低两个组别,生存分析结果那是非常的显著啊
很困惑。
GEPIA
再看看:http://gepia.cancer-pku.cn/detail.php?gene=CUL3
这款网页工具稍微有点不一样,可以选择OS和DFS,这里稍微介绍一下临床试验终点(End Point),比如大家熟知的 OS、PFS、ORR 还有 DFS、TTP、TTF……不同的终点服务于不同的研究目的。
- 总生存(Overall survival,OS)定义为:从随机化开始至(因任何原因)死亡的时间。被认为是肿瘤临床试验中最佳的疗效终点,当患者的生存期能充分时,它通常是首选终点。
- 无病生存期(Disease-free survival,DFS)定义为:从随机化开始至疾病复发或(因任何原因)死亡之间的时间。DFS 最常用于根治性手术或放疗后的辅助治疗的研究,目前是乳腺癌辅助性激素治疗、结肠癌辅助治疗、以及乳腺癌的辅助化疗的主要审批基础。
我们先看看 OS的结果吧:
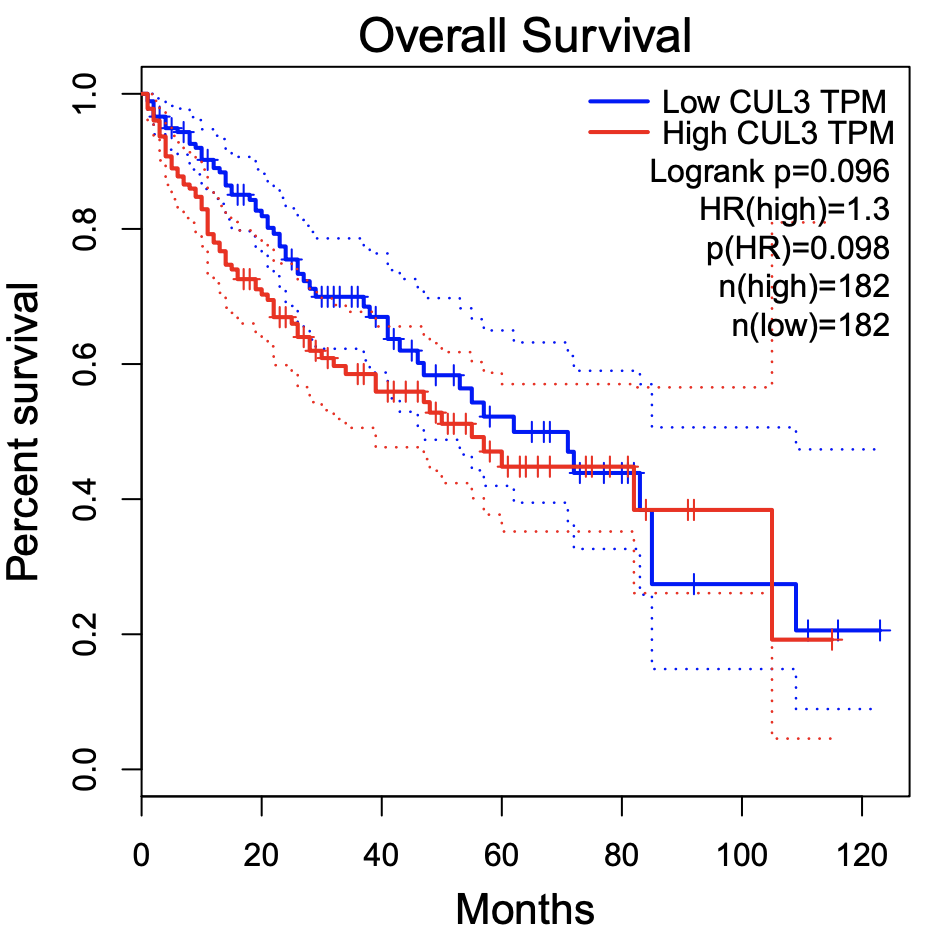
跟前面介绍的两个数据库网页工具结果都不一样,我有点头大,但还是切换了DFS再看看:
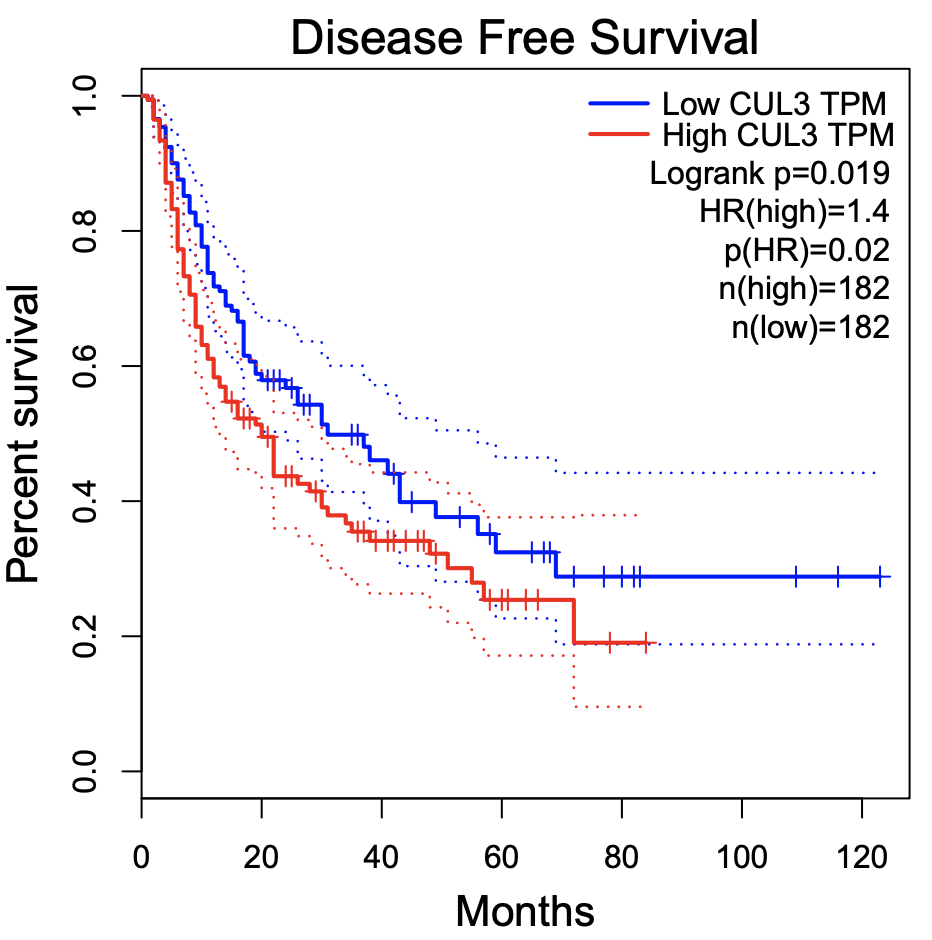
发现这个时候的生存分析输出的图跟前面的KMPLOT工具几乎是一模一样的,这个时候我思考的结果是既然有两个数据库一致,那么我们就会认为第三者,也就是oncolnc是错的,但是为什么它会错呢?
我继续探索
在R里面重新画oncolnc数据
在oncolnc网页工具里面可以下载其生存分析的数据,我首先怀疑是不是该工具自己绘图错误,所以在R里面重新绘制,代码是:
rm(list=ls())
options(stringsAsFactors = F)
# http://www.oncolnc.org/kaplan/?lower=20&upper=20&cancer=LIHC&gene_id=8452&raw=CUL3&species=mRNA
f='LIHC_8452_50_50.csv'
a=read.table(f,
header = T,sep = ',',fill = T)
colnames(a)
dat=a
colnames(dat)
library(ggstatsplot)
ggbetweenstats(data =dat,
x = Group, y = 'Expression')
ggbetweenstats(data =dat,
x = Status, y = 'Expression')
library(ggplot2)
library(survival)
library(survminer)
table(dat$Status)
dat$Status=ifelse(dat$Status=='Dead',1,0)
sfit <- survfit(Surv(Days, Status)~Group, data=dat)
sfit
summary(sfit)
ggsurvplot(sfit, conf.int=F, pval=TRUE)
如图:
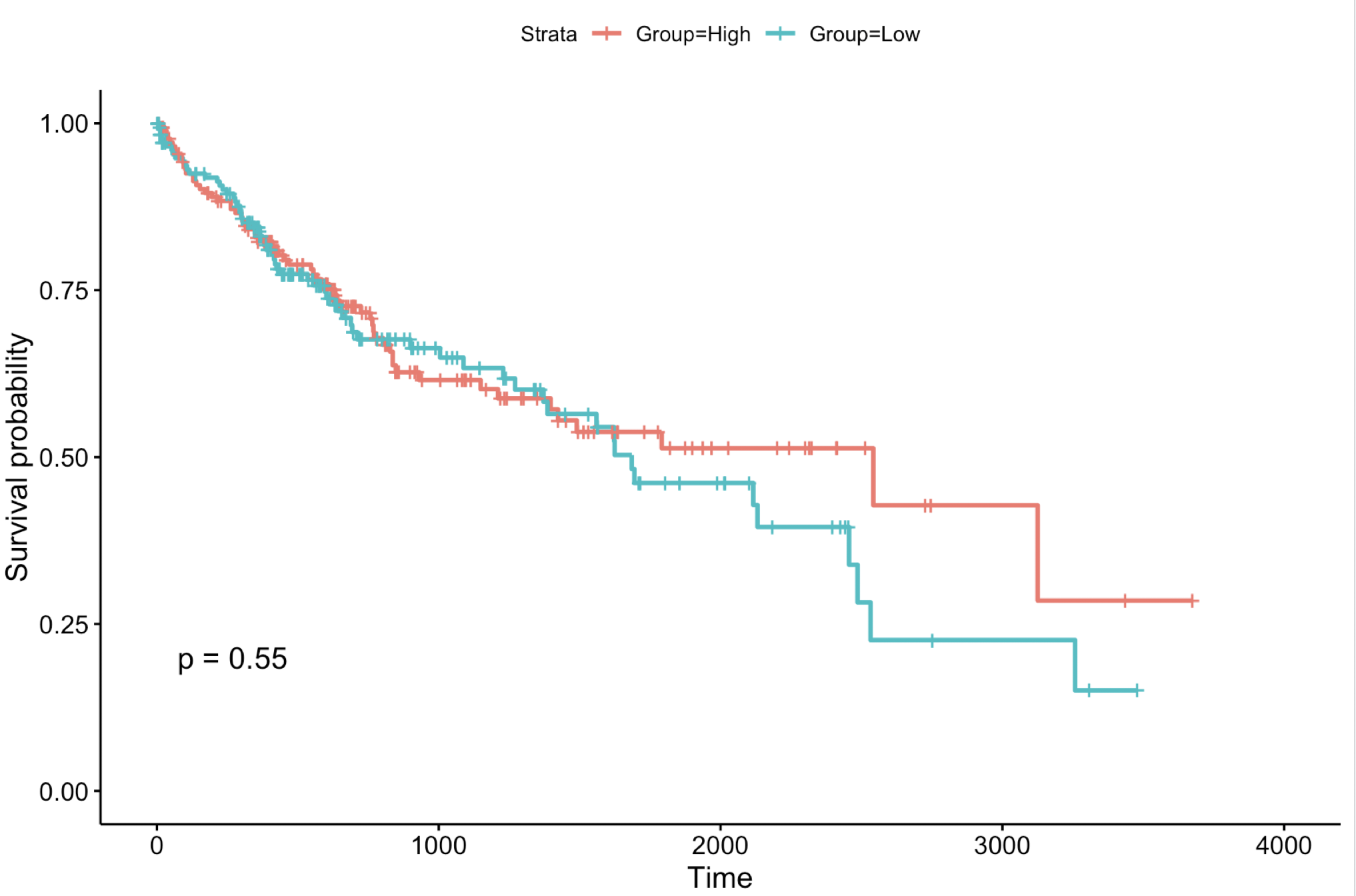
很有趣,的确是P值被扰动了,但都是不显著的,所以应该不是其网页工具绘图问题,就应该是该网页工具使用的数据源和另外两个不一样。
既然提到了TCGA数据源,我就必须看看cbioportal和ucsc的xena数据源了,同样的道理,下载它们,然后在R里面比较:
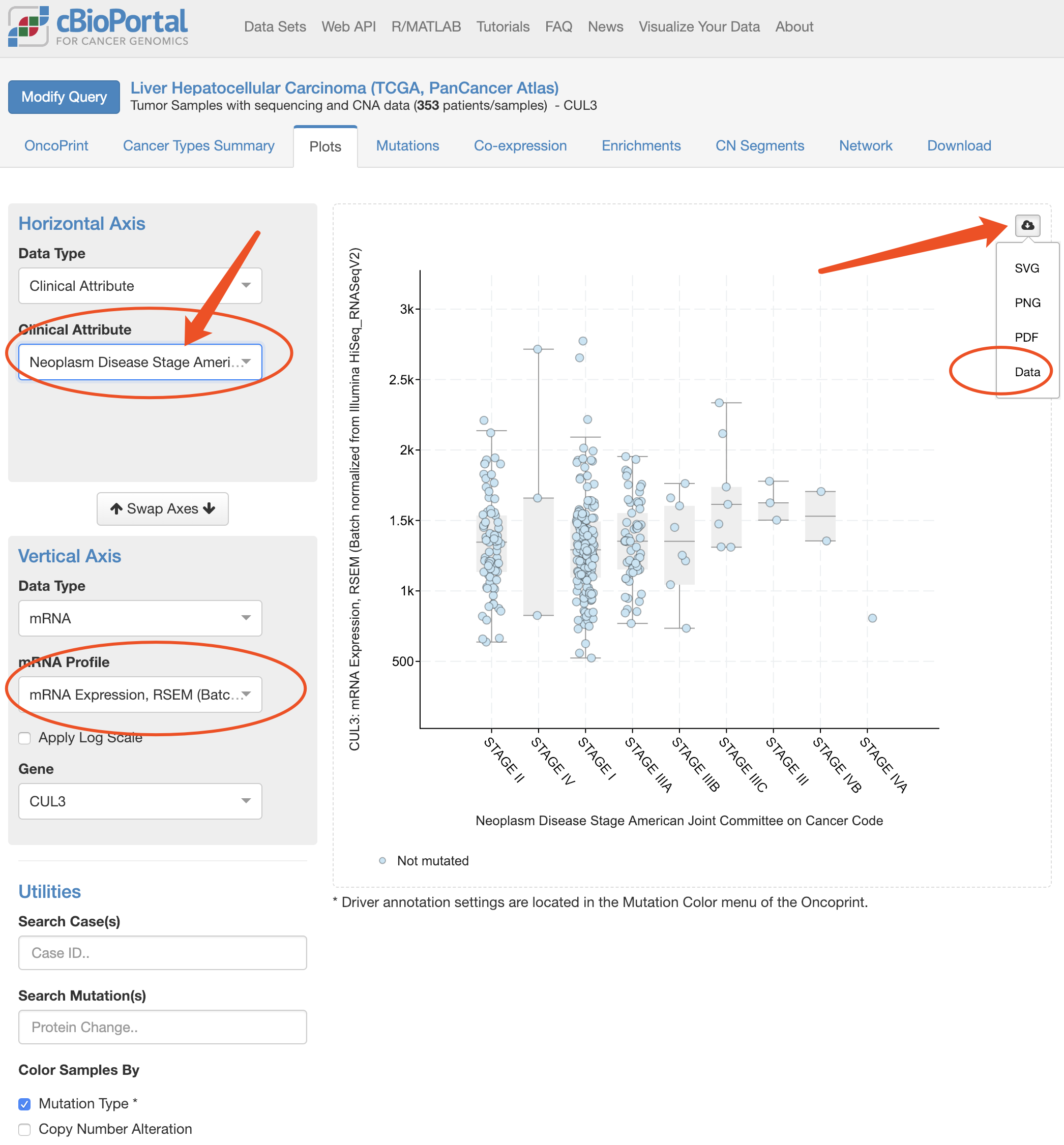
这些数据源导入到R里面,代码是:
rm(list=ls())
options(stringsAsFactors = F)
# http://www.oncolnc.org/kaplan/?lower=50&upper=50&cancer=LGG&gene_id=93663&raw=ARHGAP18&species=mRNA
# http://www.oncolnc.org
f='LIHC_8452_50_50.csv' ##
a=read.table(f,
header = T,sep = ',',fill = T)
colnames(a)
dat=a
colnames(dat)
head(dat)
## http://www.cbioportal.org
b=read.table('plot-LIHC_cbioportal.txt',
header = T,sep = '\t',fill = T)
colnames(b)=c("Patient","stage","rsem")
b[,1]=substring(b[,1],1,12)
d=merge(a,b,by='Patient')
# https://xenabrowser.net/datapages/
xena=read.table('TCGA-LIHC.survival.tsv.gz',
header = T,sep = '\t',fill = T)
xena=xena[grepl('01A',xena[,1]),]
head(xena)
e=merge(d,xena,by.x='Patient', by.y = 'X_PATIENT')
par(mfrow=c(2,1))
plot(e$Days,e$X_TIME_TO_EVENT)
plot(e$Expression,e$rsem)
神奇的事情又发生了,cbioportal和ucsc的xena数据源与前面那个oncolnc是一致的。不管是时间点记录,还是表达量记录。