本研究是乳腺癌领域的第一个类器官研究,发表于Cell. 2018 Jan 作者是 N Sachs , 到2018年底已经被引用超过50次啦,文章题目是:A Living Biobank of Breast Cancer Organoids Captures Disease Heterogeneity.
研究者收集了 超过150例乳腺癌患者样品,然后声称其团队成功的制作了超过100个类器官。
From 155 tumors, we established 95 BC organoid lines that readily expanded.
而且NGS数据分析结果表明类器官比较好的保留其配对肿瘤样品的CNV和SNV特征。
分析流程很常规,就是想办法提供organoids的成功率,并且从各个角度来证明:
- histology
- whole genome DNA sequencing (WGS)
- RNA sequencing (RNA-seq)
- drug testing
全基因组分析
上游分析得到somatic的SNVs流程我在生信技能树多次介绍过,这里作者采取的是其实验室内部数据分析流程,但是大同小异。数据上传到了 EGAS00001002158 需要申请授权才能下载。
数据分析流程详细描述如下:

全基因组测序数据得到的CNV信息,有两个匹配比较好的organoids如下:

事实上这样的展现方式并不好,需要仔细看两个CNV图的相似性,如果仔细看,其实很多地方并不匹配。
其它的配对的类器官就不值一提,所以作者仅仅是挑选部分基因来看organoids及其primary肿瘤的CNV的匹配情况,如下:
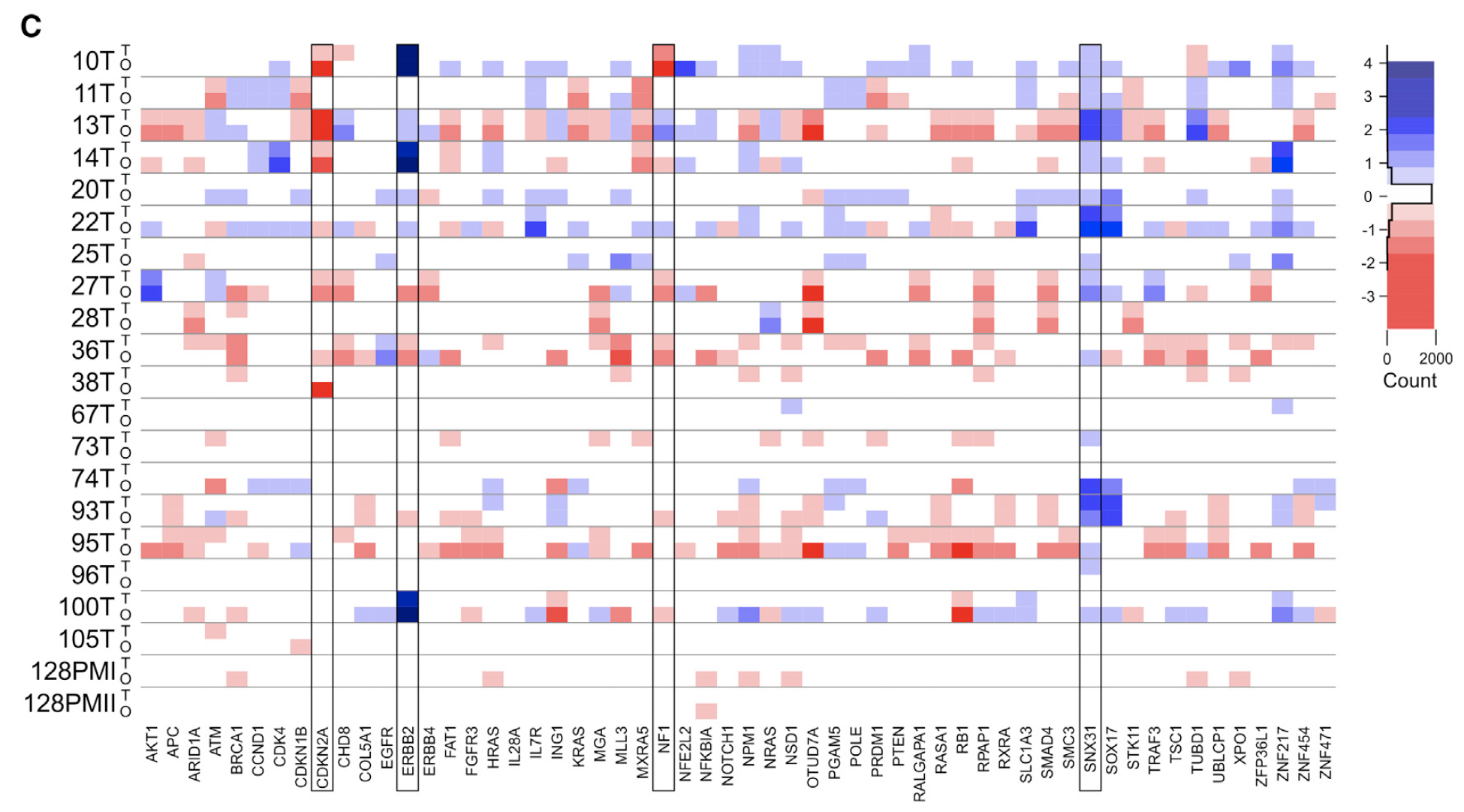
即使是作者只挑选部分基因来展示CNV的匹配性,而且作者也声称匹配性很好,可是他并没有给什么统计学指标证明匹配很好。
对SNV也是如此,挑选部分基因秀,如下:
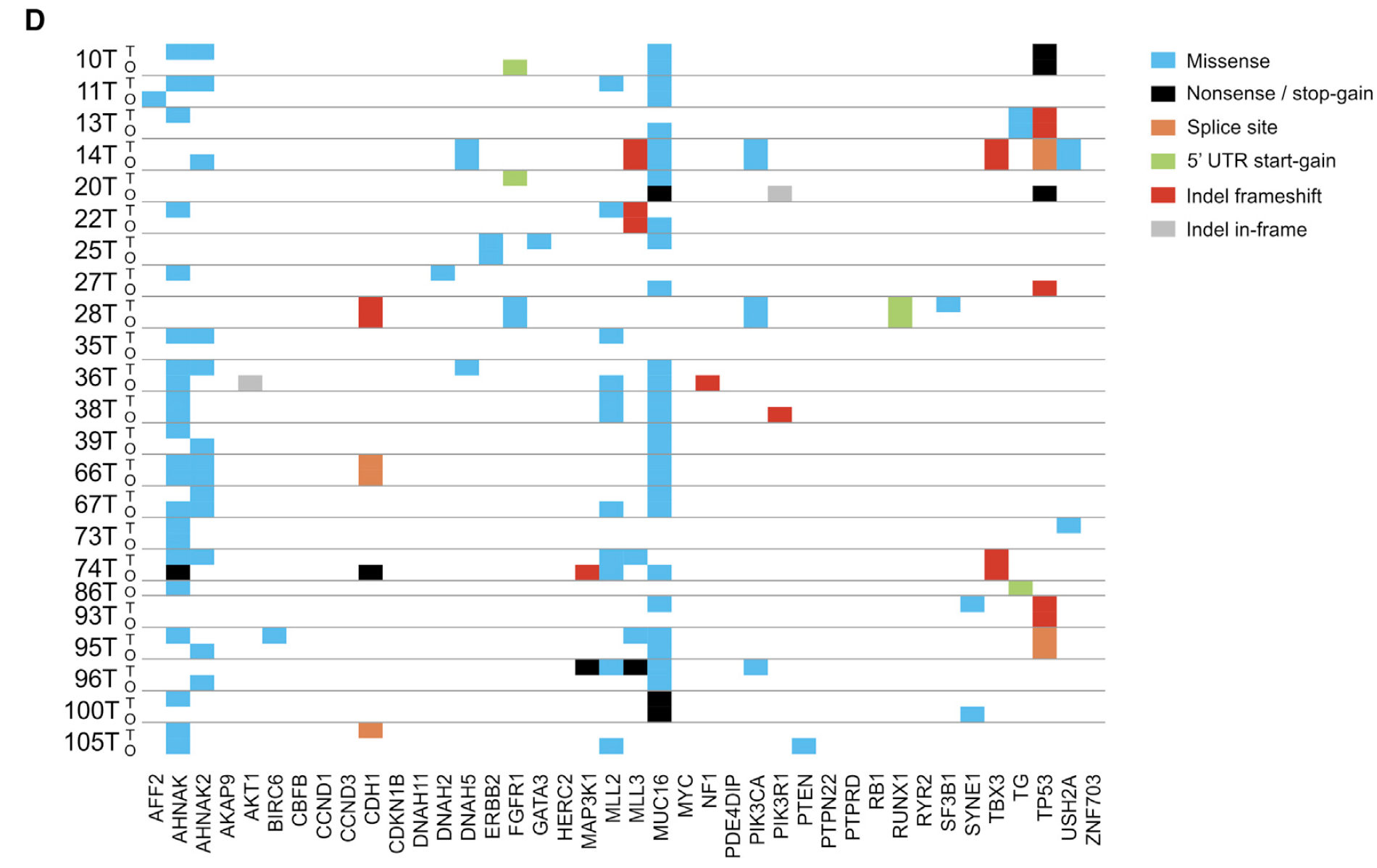
可以肯定的是organoids的SNV不可能百分百与其primary肿瘤的somatic SNV一致,所以作者比较的是突变的总数,以及突变的特征频谱,如下:
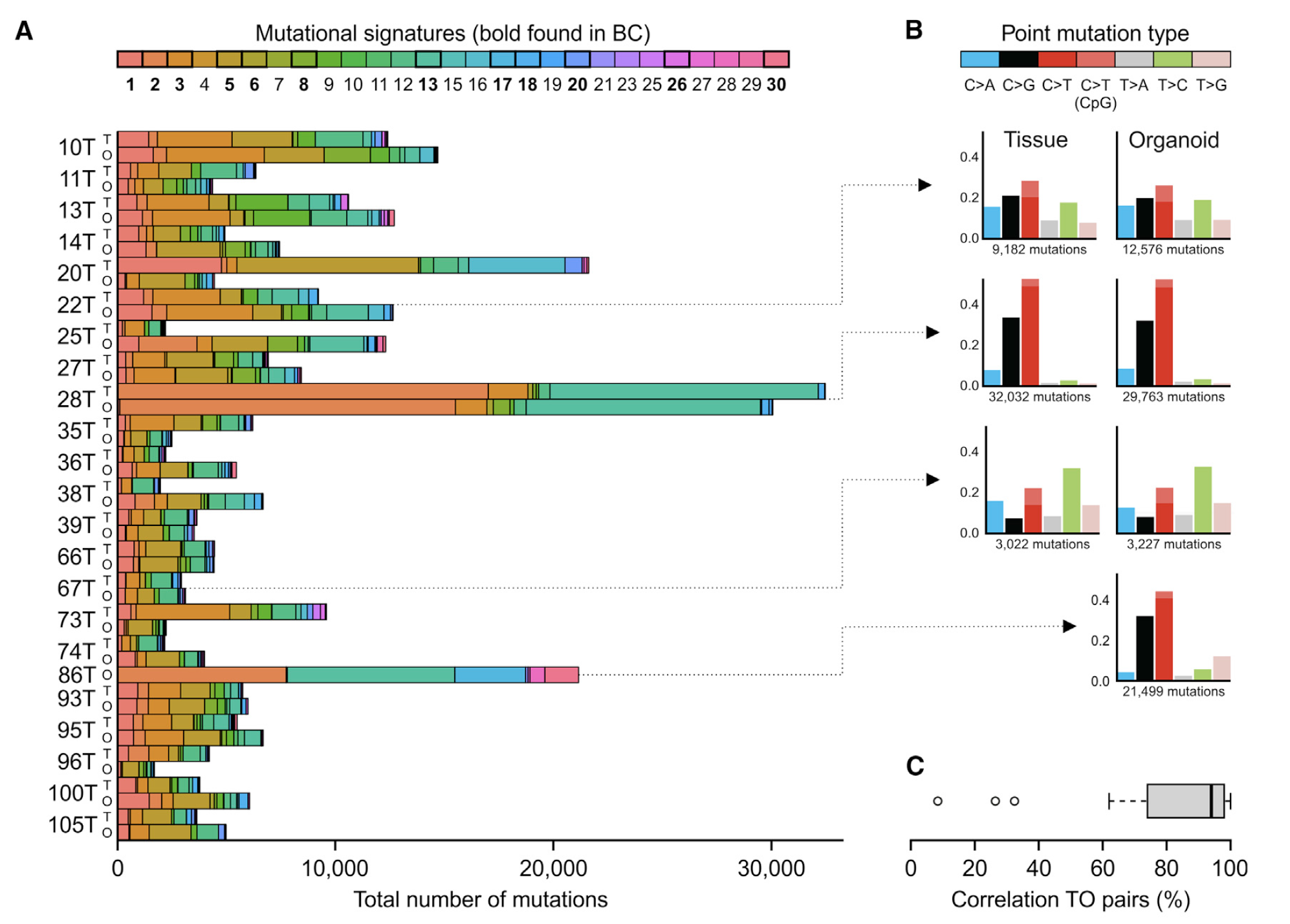
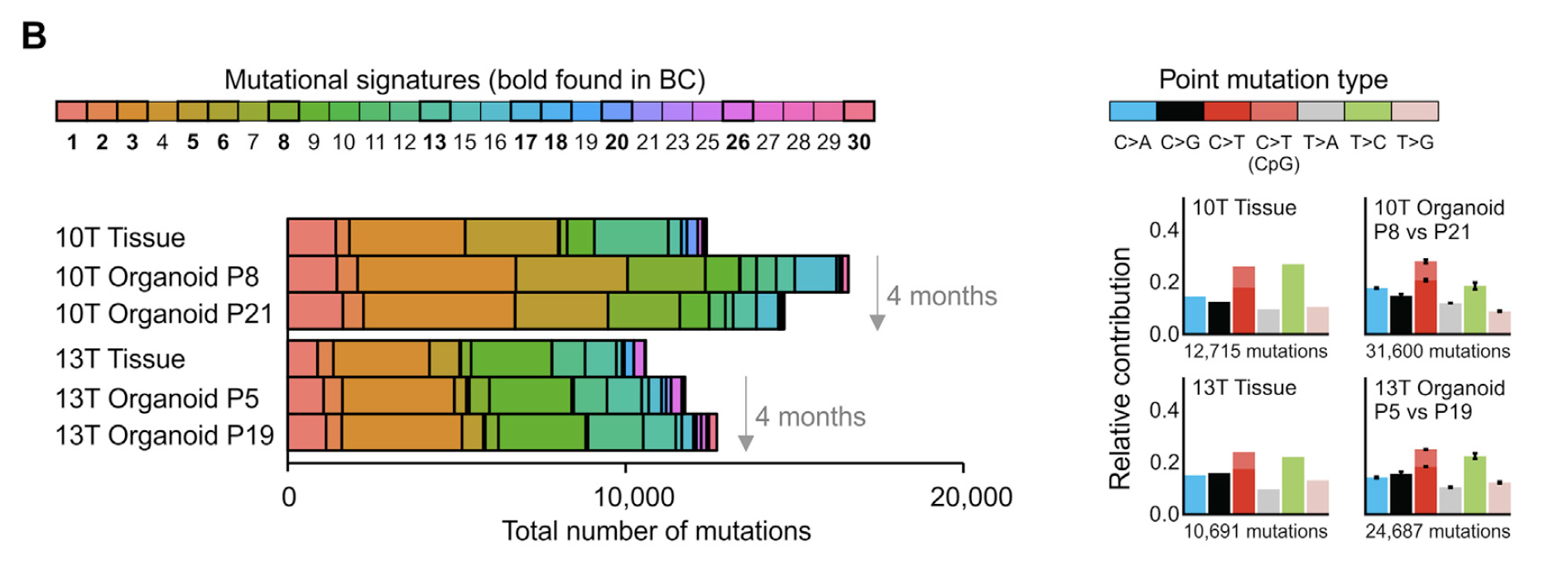
从DNA变异情况看organoids不同培养时期的情况
同样,首先是CNV情况:
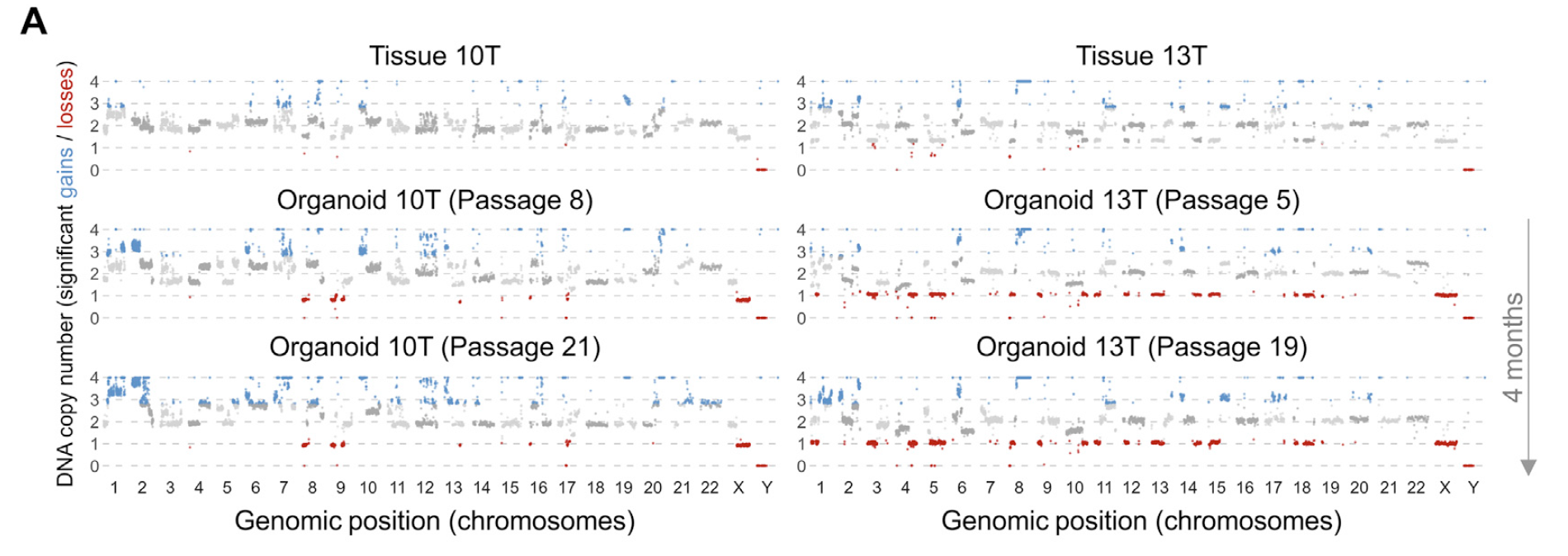
可以看到是类似的,可以得出结果organoids保留了其肿瘤的分子特性。
然后看SNV的,首先是突变数量和6突变频谱如下:
然后看96突变频谱:
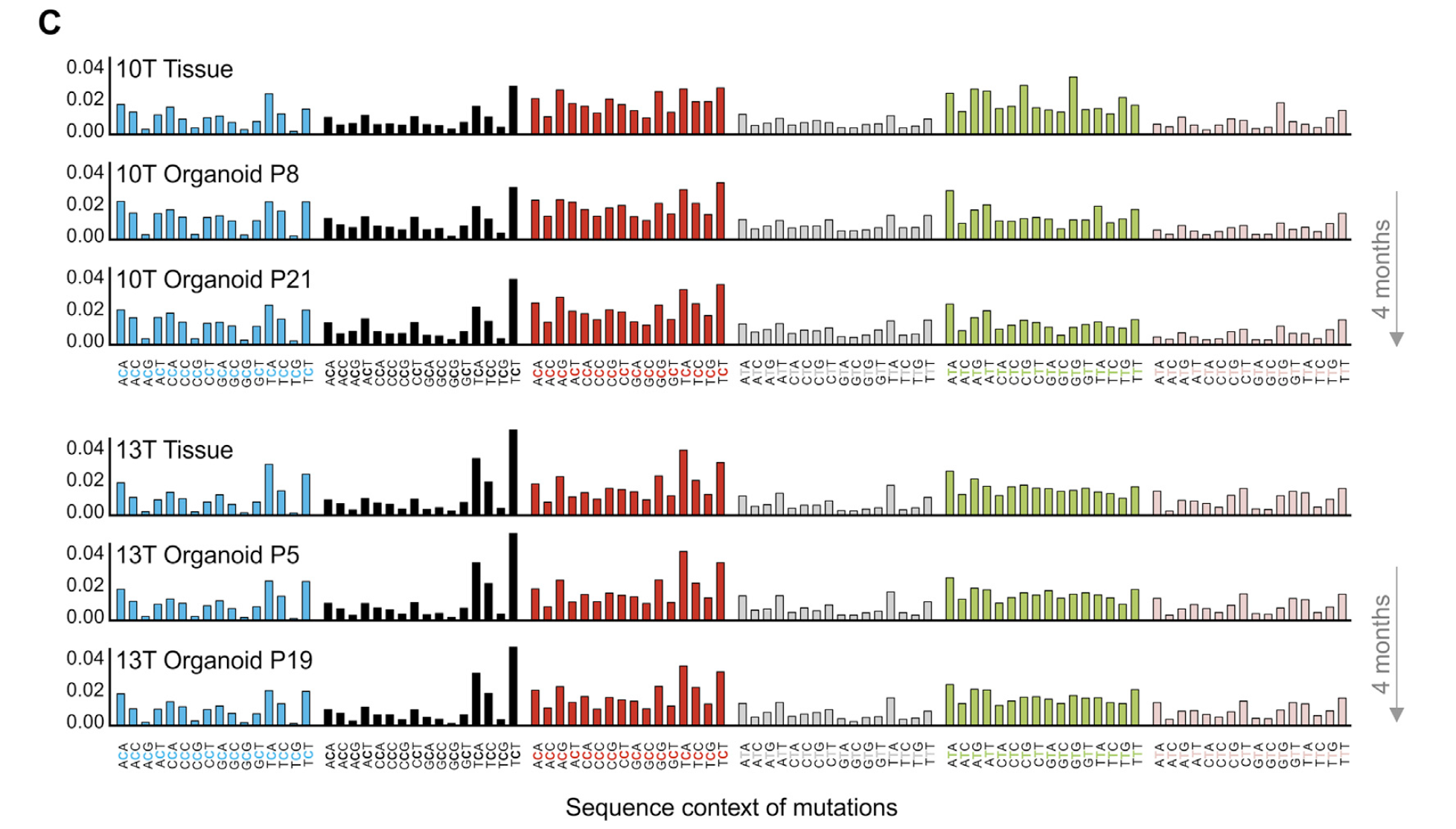
最后作者的结论就是organoids保留了其肿瘤的分子特性,而且培养时间影响不大。转录组数据分析
首先看作者的转录组数据处理方法:

转录组表达量矩阵的利用率比较低,基本上就看了看相关性,还不如PCA图来的直观,而且也就是跟TCGA的乳腺癌糅合在一起进行聚类,如下;
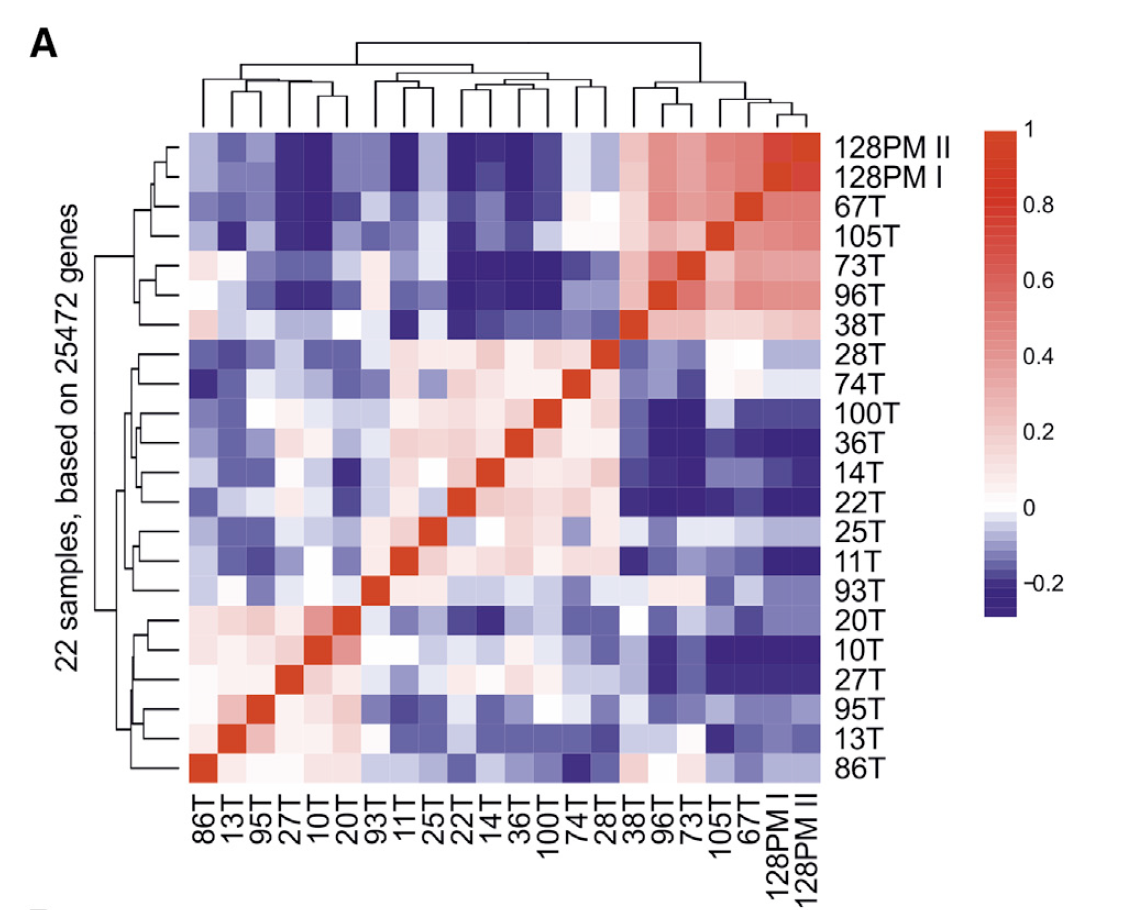

药物处理数据分析
目前不是我急需的, 后来肯定会花时间补充这部分知识点。
因为数据并不能下载重新分析,所以本文解读到此结束。