发表于:Clin Cancer Res. 2015 Apr 文章题目是:Comprehensive Genomic Analysis Identifies Novel Subtypes and Targets of Triple-negative Breast Cancer 主要是考虑到TNBC是异质性较大的一种乳腺癌,而且预后非常差,所以临床用药指导急需对TNBC本身更加细致的分类,研究团队通过贝勒医学院分两次 收集了 198个TNBC病人, (discovery set: n=84; validation set: n=114) 比较稳定的把TNBC分成了4个亚型,而且还分析了7个公共数据集来验证其结论。
作者整理的4个亚型以及其可能的靶点:
- 1) LAR: androgen receptor and the cell surface mucin MUC1;
- 2) MES: growth factor receptors (PDGF receptor A; c-Kit);
- 3) BLIS: an immune suppressing molecule (VTCN1)
- 4) BLIA: Stat signal transduction molecules and cytokines.
其中BLIA组的预后最差,LAR的拷贝数变异比较独特。背景知识
肿瘤异质性一直是热点,主要有3种研究策略:
- Deep-sequencing studies (Balko et al., 2012, Balko et al., 2014, Shah et al., 2012)
- multi-region sequencinganalysis (Yates et al., 2015)
- single-cell sequencing studies (Gao et al., 2016, Navin et al., 2011, Wang et al., 2014)
通过IHC(免疫组化)分型方法,乳腺癌被划分为激素受体(ER、PR)阳性组和阴性组,后者根据HER2表达情况进一步分为HER2过表达乳腺癌和三阴性(ER、PR、HER2阴性)乳腺癌(TNBC)。
乳腺癌病人的异质性在治疗领域是很大的问题,有文章根据表达量分6类,如下: - luminal A
- luminal B
- ERBB2-enriched
- basal-like
- claudin-low
- normal-like
当然,PAM50的分类也是可以的。
其中 表达量得到的basal-like 亚型和 IHC得到的TNBC 有重合,但并不等价。
而在2011就把TNBC分类过,如下: - 1) androgen receptor positive
- 2) claudin-low-enriched mesenchymal
- 3) mesenchymal stem-like
- 4) immune response
- two cell cycle-disrupted basal subtypes 5) BL-1 and 6) BL-2.
实验设计
其中 Affymetrix U133 Plus 2.0 的表达芯片数据使用的是 R语言里面的affy包处理得到表达矩阵,数据上传到了GEO里面,
而 Illumina 610K and 660K 这样的基因分型拷贝数芯片数据使用的是Illumina Genome Studio v2011 Genotyping这样的官方工具,数据并没有上传。数据处理
挑选 median absolute deviations (MADs) 最大的 1000个基因在所有样本的表达信息矩阵,通过 Non-negative Matrix Factorization (NMF) 这个R包来进行聚类,发现可以比较稳定的聚为 4 类。
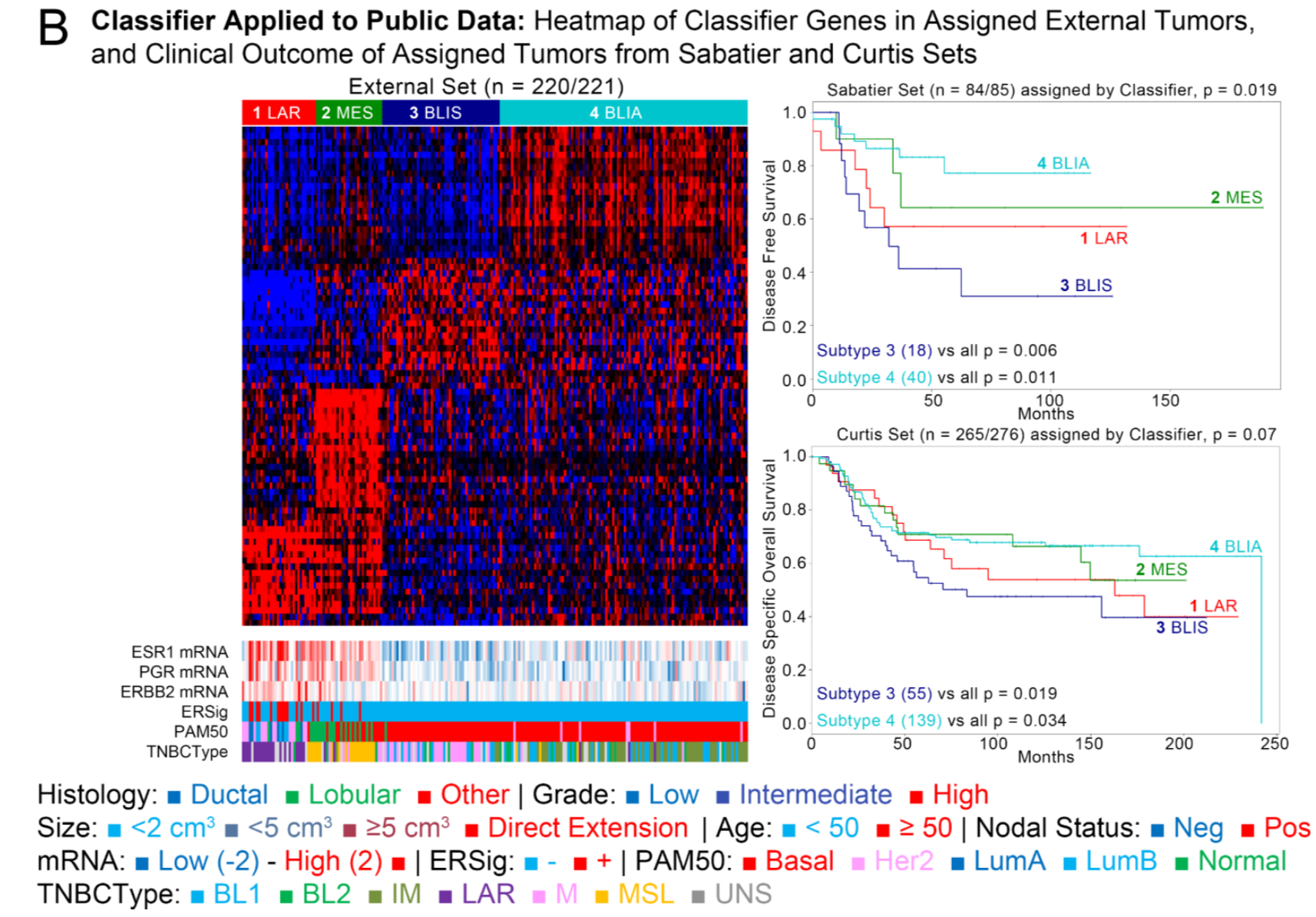
还针对最明显的两个类别,即 basal-like versus the remaining intrinsic subtypes进行了差异表达分析,然后根据 log2(Fold Change) (“FC”) 和 Benjamini-Hochberg (BH) False Discovery Rate (FDR) 值来挑选 20个基因作为分类器。
作者使用付费软件 Ingenuity Systems’ Interactive Pathway Analysis (IPA) 进行基因集的功能分析。
针对基因分析及拷贝数芯片,作者使用 Allele-Specific Piecewise Constant Fitting (ASPCF) 和 Allele-Specific Copy Number (CN) Analysis of Tumors (ASCAT)算法得到拷贝数变异情况,然后使用 Genomic Identification of Significant Targets in Cancer (GISTIC) 2.0 软件找 统计学显著的拷贝数变异区域。
生存分析,包括 disease-free and overall survival (DFS and OS) 时间点。主要分析结果解读
首先是NMF得到的4类 :
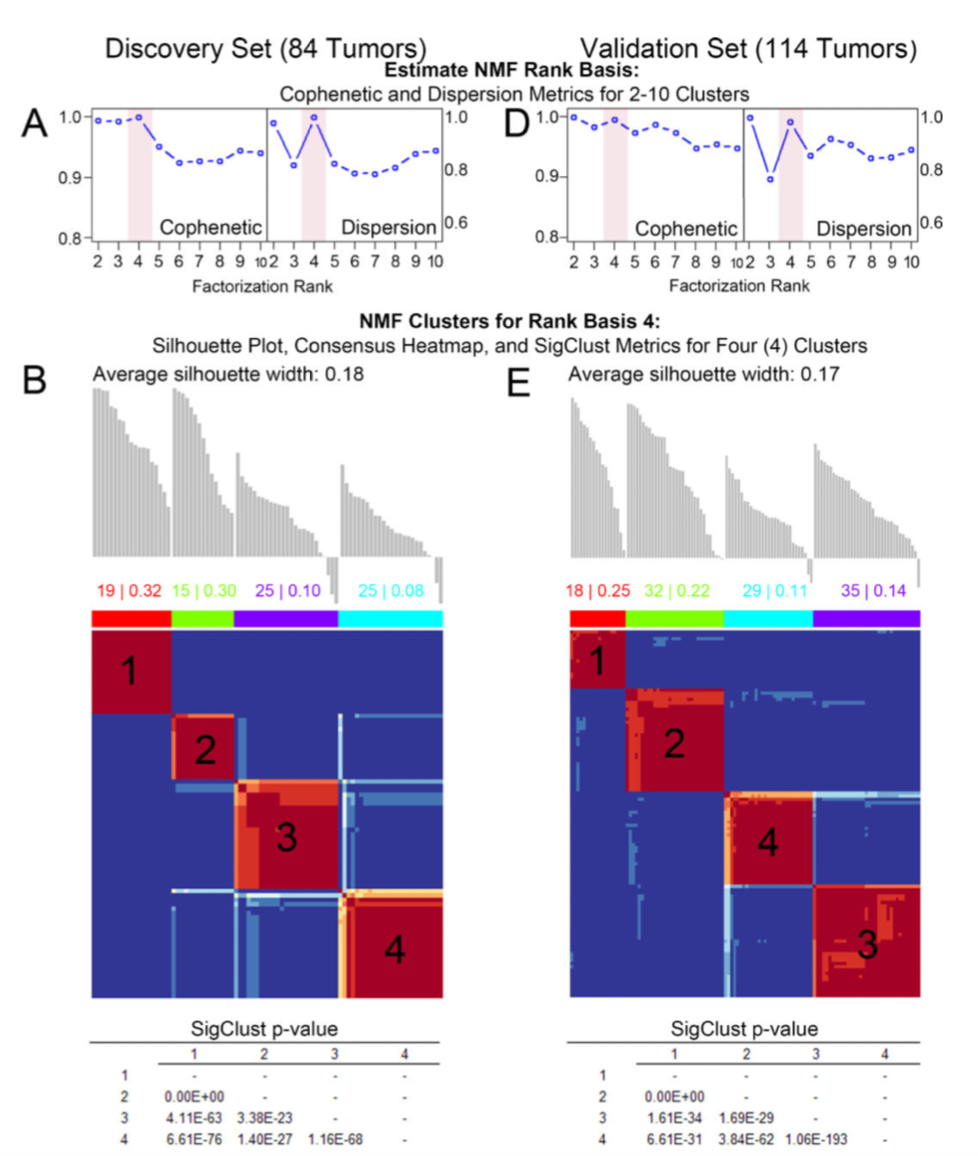
只要有这个表达矩阵,挑取top1000的mad的基因,然后时候该R包调用NMF算法即可出结果。84 (discovery set) and 114 TN breast tumors (validation set) both demonstrate 4 stable clusters by NMF of mRNA expression across the top 1000 genes (IQR summarized) selected by DEDS aggregate rank of median absolute deviations (see complete methods) of the discovery set.
然后是作者数据集的分类结果和一下公共分类方法结果进行比较:
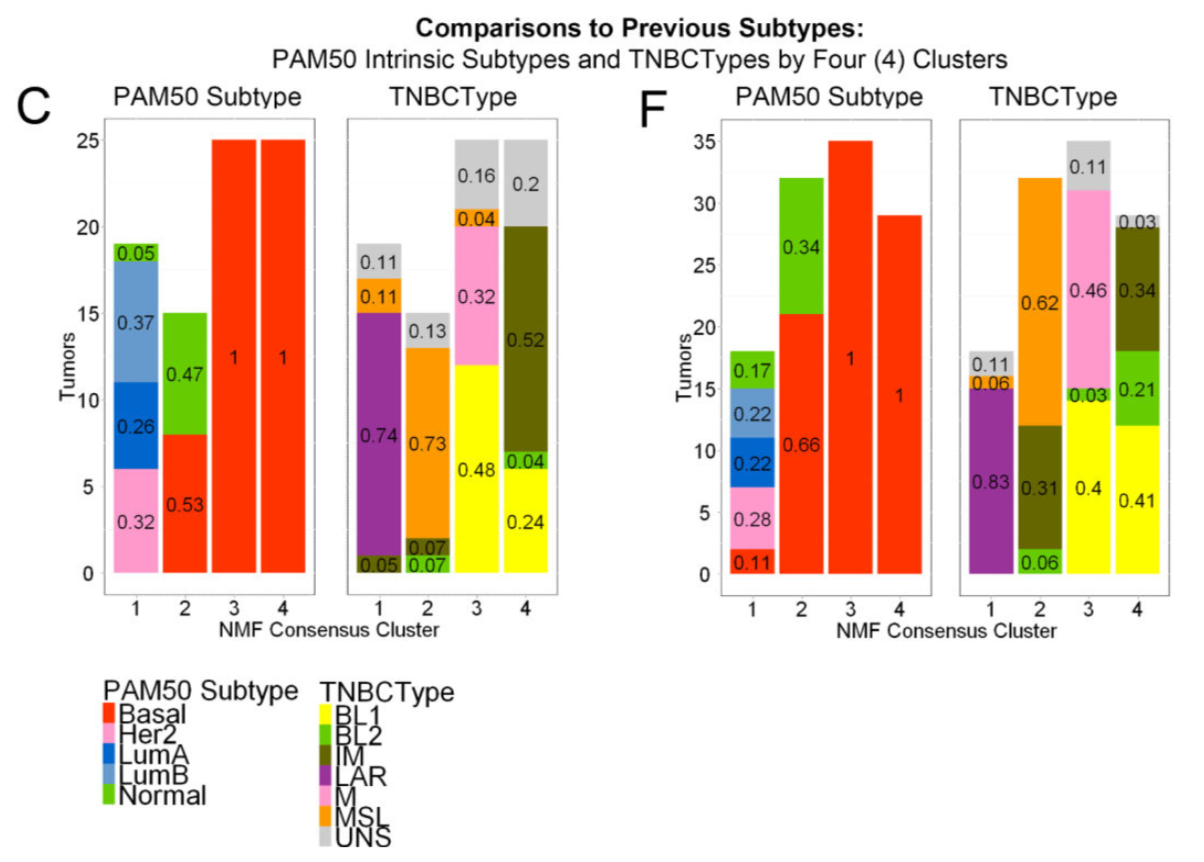
这2个分类方法: - Perou’s “PAM50” TNBC molecular classification (luminal A, luminal B, HER-2-positive, basal-like and normal-like subtypes)
- Lehmann/Pietenpol “TNBC Type” molecular classification (basal-like-1, basal-like-2, immunomodulatory, luminal androgen receptor (LAR), mesenchymal, and mesenchymal stem-like subtypes)
也是有成熟的R包可以调用,就可以进行简单的比较。
接着是基因集的展示,主要是热图看看是否表达量很明显在不同亚型有差异,首先在训练集和测试集里面进行检查,如下图:

然后在外部公共数据集里面查看:
然后对3个数据集的4个亚型的基因集都进行GO/KEGG注释,这里作者选择的是IPA这个收费软件,结果如下:
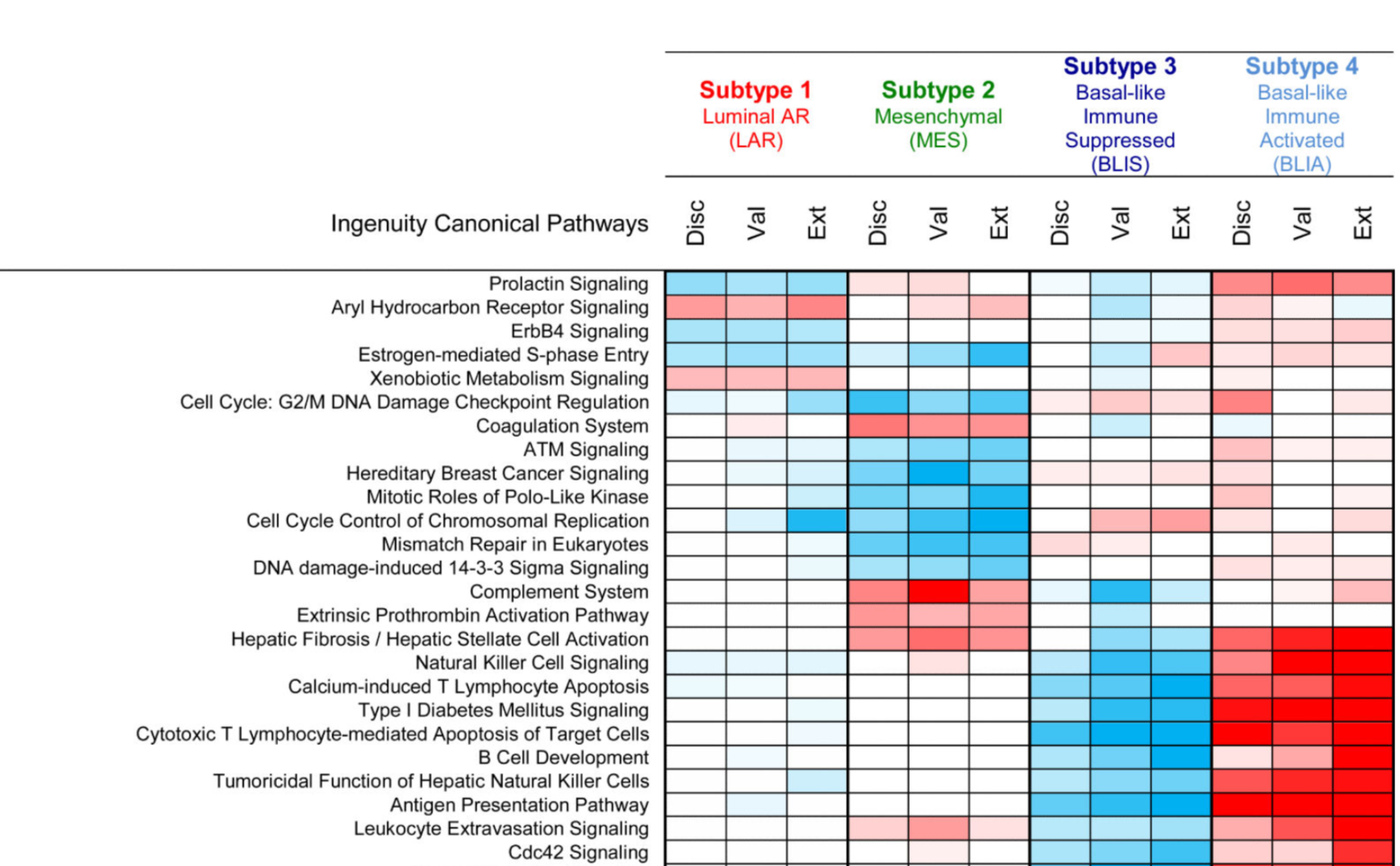
重点:有了上面的数据库注释信息,就可以根据注释结果对我们的4个分组进行命名!!!
最后,有了转录水平的分类信息,然后就可以对他们同步获得的CNV信息也是进行分类总结,简单的全局CNV信息如下:

作者没有提供单独的每个样本的segment化的cnv数据,所以没办法重复他们的分析。作者单独指出来LAR这个亚型和另外3个亚型的CNV信息差异很大。后记
作者的这个大数据结果只是用来做了分类,而且是TNBC群体的,算是乳腺癌的热点,那么是不是可以进行深度挖掘呢?
比如:
- PMID: 25208879
- PMID: 26921331
- PMID: 30175120