发现自己的基因集使用KEGG数据库进行分析的时候,很多免疫相关通路被富集,所以怀疑是不是这些免疫通路互相交叉很多,就试着探索一下,是一个蛮好的练习题:
这里举个例子,需要探索的通路如下:
from to 307 hsa04650 Natural killer cell mediated cytotoxicity 308 hsa04657 IL-17 signaling pathway 309 hsa04658 Th1 and Th2 cell differentiation 310 hsa04659 Th17 cell differentiation 311 hsa04660 T cell receptor signaling pathway 312 hsa04662 B cell receptor signaling pathway
我本来准备用KEGG.db想起来它有点老旧,又准备用KEGGREST,毕竟之前生信技能树写过这个教程,但是正好在跟Y叔聊天,就用它的包吧!
代码如下,很简单。
library(clusterProfiler)
# https://www.kegg.jp/dbget-bin/www_bget?pathway+hsa05169
# library(KEGG.db) library(KEGGREST)
kg=download_KEGG('hsa')
head(kg[[1]])
head(kg[[2]])
ps=c('hsa04660','hsa04659',
'hsa04658','hsa04657','hsa04662',
'hsa04650')
kg[[2]][kg[[2]][,1] %in% ps,]
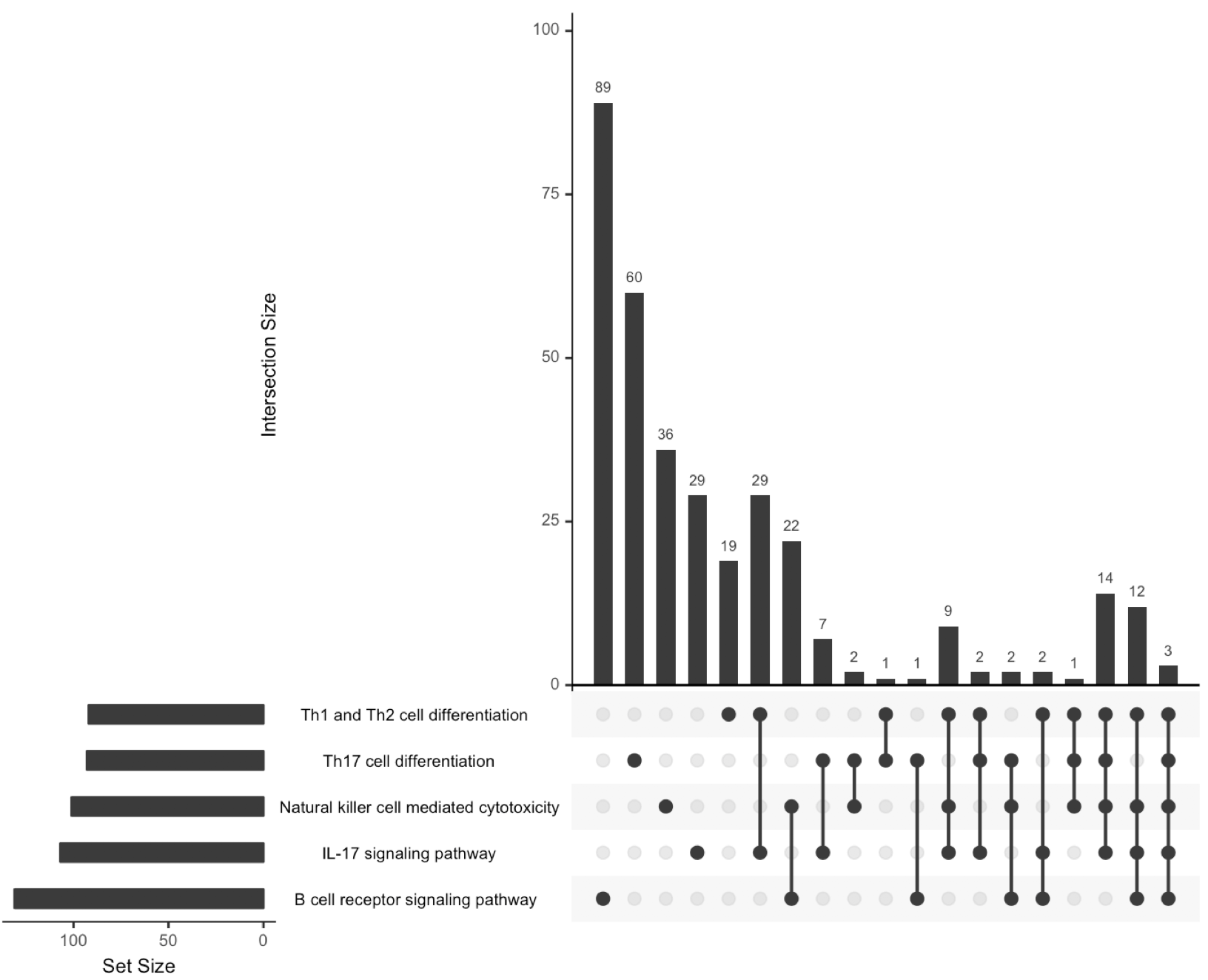
library(UpSetR)
allgs=unique(kg[[1]][kg[[1]][,1] %in% ps,2])
u=do.call(cbind,lapply(ps, function(x){
as.numeric(allgs %in% kg[[1]][kg[[1]][,1]==x,2])
}))
u=as.data.frame(u)
rownames(u)=as.character(allgs)
colnames(u)=kg[[2]][kg[[2]][,1] %in% ps,2]
upset(u)
出图如下;