一:下载安装该软件
下载安装该软件: wget https://codeload.github.com/TransDecoder/TransDecoder/tar.gz/2.0.1
解压进入该目录,查看里面的文件
make一下就可以用了,看起来好像是依赖于perl模块的
这个TransDecoder.LongOrfs就是我们这次需要的程序,查看该程序,的确真是一个perl程序,看来perl还是蛮有用的。
二:准备数据
它里面有个测试数据,是比较全面的,也比较复杂,我就不贴出来了,反正我是那trinity组装好的fasta格式的转录组数据来预测ORF的。
三:运行命令
它给的测试命令也很复杂
## generate alignment gff3 formatted output
../util/cufflinks_gtf_to_alignment_gff3.pl transcripts.gtf > transcripts.gff3
## generate transcripts fasta file
../util/cufflinks_gtf_genome_to_cdna_fasta.pl transcripts.gtf test.genome.fasta > transcripts.fasta
## Extract the long ORFs
../TransDecoder.LongOrfs -t transcripts.fasta
当然我们只需要看最后一步,这是重点
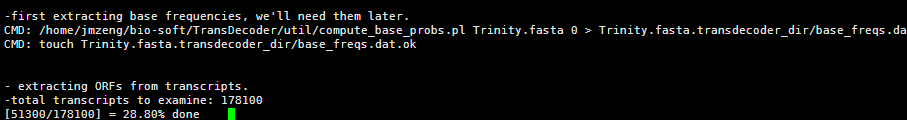
我这里是直接对我们的trinity组装好的转录本进行预测ORF
/home/jmzeng/bio-soft/TransDecoder/TransDecoder.LongOrfs -t Trinity.fasta
命令很简单
输出来的文件就有预测的蛋白文件,这个文件是trinotate对转录本进行注释所必须的文件
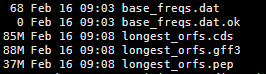
四:输出文件解读
longest_orfs.cds 这个是预测到的cds碱基序列,
longest_orfs.gff3 这个是预测得到的gff文件
longest_orfs.pep 这个就是预测得到的蛋白文件